Aedes aegypties el principal vector de arbovirus causantes de las enfermedades más comunes en las áreas tropicales y subtropicales del mundo 1. Las poblaciones de este vector varían en su capacidad para infectar, propagar y transmitir Flavivirus como el del dengue (DENV1-4), el Zika (ZIKV), el chikungunya (CHYKV) y la fiebre amarilla (YFV) 2-4. La transmisión biológica de arbovirus por mosquitos está asociada tanto a factores ecológicos como a factores intrínsecos relacionados con el acervo genético del vector (variantes genéticas), la propensión a la infección y la permisividad para el desarrollo del patógeno 5-8.
Para el análisis de la variabilidad genética de Ae. aegypti,se han utilizado múltiples marcadores y técnicas moleculares, como el análisis de polimorfismos de la longitud de los fragmentos amplificados (Amplified Fragment-Length Polymorphisms, AFLP) 9, los polimorfismos de la longitud de los fragmentos de restricción (Restriction Fragment Length Polymorphism, RFLP) 10, la amplificación aleatoria de ADN polimórfico (Random Amplification of Polymorphic DNA, RAPD) 11-13, el polimorfismo de nucleótido único(Single Nucleotide Polymorphism, SNP) 14, las isoenzimas 15, el polimorfismo de conformación de cadena simple (Single-Stranded Conformation Polymorphism, SSCP) y los espaciadores transcritos internos (Internal Transcribed Spacer, ITS) 16, los micro-satélites 17,18 y el marcador nuclear EPIC (Exon-PrimedIntron-Crossing) 19.
Los genes mitocondriales han demostrado ser una herramienta útil para la diferenciación genética y el estudio del flujo génico en varios organismos 20. Entre los principales marcadores mitocondriales usados para el estudio de poblaciones de Ae. aegypti, están el gen mitocondrialND4 (NADH dehydrogenase subunit 4) 11,20-25, el gen mitocondrial ND5 (NADH dehydrogenase subunit 5) 26,27 y el gen COX1 (cytochrome C oxidase subunit I) 28-30.
El gen ND4, que codifica para la subunidad 4 de la enzima NADH deshidrogenasa, ha sido ampliamente usado como marcador para estudios de genética de poblaciones y permite inferir la historia evolutiva de las poblaciones del vector 11,20,22,24,25; además, la disponibilidad en los bancos de datos (GenBank, BOLD y EMBL) de secuencias nucleotídicas homólogas de poblaciones del mosquito de distintos países, facilita este tipo de estudios.
En el departamento de Sucre existe información limitada sobre la composición genética de Ae. aegypti, el flujo de genes y las relaciones evolutivas entre las poblaciones del vector; esta información es necesaria para comprender la dinámica poblacional, la distribución del vector en la región y la epidemiología de la enfermedad. A la fecha, solo se ha caracterizado la variabilidad genética del mosquito en dos municipios de Sucre 25.
El presente estudio tuvo como objetivo determinar la variabilidad y la estructura genética de las poblaciones de Ae. aegypti en cuatro municipios del departamento de Sucre, mediante el análisis de la secuencia de nucleótidos del gen mitocondrial ND4.
Materiales y métodos
Área de estudio y recolección de larvas de Aedes aegypti
El estudio se llevó a cabo en cuatro municipios del departamento de Sucre, en la región Caribe de Colombia, que fueron Sincelejo (julio a agosto de 2011 y abril a mayo de 2013), Sampués (abril de 2013), Corozal (mayo de 2013) y Guaranda (abril de 2012) (figura 1). Las localidades seleccionadas en cada municipio y sus respectivas coordenadas geográficas, se describen en el cuadro 1. La escogencia de estas localidades se basó en los registros epidemiológicos sobre la presencia del vector, de la Secretaría de Salud de Sucre (datos sin publicar).

Figura 1 Mapa del departamento Sucre en el que se indica la ubicación de los cuatro municipios en los cuales se desarrolló el estudio.
En cada localidad se hizo una búsqueda activa de larvas de Ae. aegypti, en la que se inspeccionaron los depósitos de almacenamiento de agua encontrados en el interior y alrededor de las viviendas seleccionadas; la distancia entre cada vivienda fue de 50 metros, aproximadamente. Se recolectaron larvas de tercer y cuarto estadio de Ae. aegypti, las cuales se transportaron al Laboratorio de Investigaciones Biomédicas de la Universidad de Sucre, donde se criaron bajo condiciones estándares hasta alcanzar el estado adulto. La identificación taxonómica se llevó a cabo con las claves de Rueda 31.
Extracción de ADN, amplificación y secuenciación del gen ND4
Se hizo la extracción del ADN total a partir de hembras adultas de Ae. aegypti, según el protocolo descrito por Caldera, et al. 25, pero modificando el tiempo de incubación con proteinasa K, el cual fue ajustado a cinco horas a una temperatura de 65 °C 32. El ADN obtenido se cuantificó en el espectrofotómetro Nanodrop 2000™ de Thermo Scientific.
Para la reacción en cadena de la polimerasa (PCR), se utilizaron los cebadores ND4F5’-ATTGCCTAAGGCTCATGTAG-3’ y ND4R 5’- TCGGCTTCCTAGTCGTTCAT-3’ descritos por Bracco, et al. 22, los cuales flanquean una región de 361 pb, aproximadamente, del gen mitocondrial ND4 de Ae. aegypti. La reacción se llevó a cabo en un volumen final de 25 µl, compuesto por MgCL2 5 mM, DNTP 0,2 mM, cebadores 0,4 µM, solución tampón de PCR 1X, dos unidades de Taq polimerasa y 1 µl de la solución con el ADN, que contenía aproximadamente 100 ng/µl. En cada serie de PCR, se incluyó un control negativo constituido por agua estéril en lugar de ADN.
El perfil térmico consistió en una etapa inicial de desnaturalización a 94 °C durante cinco minutos, seguida por 35 ciclos de desnaturalización a 94°C por 60 segundos, alineamiento a 56°C durante 30 segundos y extensión a 72°C por 60 segundos, y una etapa final de extensión a 72°C durante siete minutos.
Los productos de PCR fueron separados por electroforesis en gel de agarosa al 1 % (peso/volumen), desarrollada en solución tampón TBE 0,5X a 90 V por 50 minutos. El ADN se tiñó con Gel Star™ y se visualizó con luz ultravioleta. Los productos amplificados fueron secuenciados mediante electroforesis capilar en un secuenciador automático, con los mismos cebadores empleados para la PCR.
Análisis genético
Las secuencias derivadas de los electroforegramas fueron editadas y alineadas con el programa MEGA, versión 5.0 33. Cada secuencia de consenso fue analizada con el BLASTn (34) para corroborar que correspondía al genND4 de Ae. aegypti.
Con el programa DnaSP, versión 5.1.1.8 35, se determinaron el número de haplotipos, la frecuencia de haplotipos, el número de sitios polimórficos (S), el número promedio de diferencias de nucleótidos (k), la diversidad de haplotipos (DH) y la diversidad de nucleótidos (π). Asimismo, se calcularon los valores de las pruebas de neutralidad D de Tajima 36, y la D y la F de Fu y Li 37. La genealogía entre los haplotipos fue construida utilizando el programa Network, versión 4.6.1.1 38 con el algoritmo median joining39. Además, con el programa Arlequin, versión 3.5.1.2 40, se estimó la estructura genética mediante el índice de fijación (F ST ), y se calculó el flujo genético (Nm), y se hizo un análisis de varianza molecular (AMOVA).
La significación del efecto de las distancias geográficas en la diferenciación genética fue evaluada mediante el test de Mantel 41, el cual utiliza el coeficiente de regresión lineal entre el valor de F ST (1-F ST ) 42 y el logaritmo de las distancias geográficas entre cada par poblacional. Las distancias geográficas entre los sitios de captura se obtuvieron a partir del calculador de distancias por coordenadas (disponible en la web). Los resultados del test de Mantel fueron tomados después de 1.000 permutaciones, usando el programa Mantel, versión 1.19 43.
Las secuencias obtenidas durante esta investigación fueron registradas en el GenBank.
Resultados
Secuencias de nucleótidos
Se obtuvieron 108 secuencias de 357 nucleótidos del gen ND4 de Ae. aegypti. El número de mosquitos de cada localidad de los cuales se derivan las secuencias de nucleótidos, se presenta en el cuadro 1. Las secuencias parciales del gen ND4 de Ae. aegypti no mostraron inserciones, deleciones o codones de parada.
El análisis reveló la presencia de cuatro haplotipos mitocondriales denominados H1, H2, H3 y H4, los cuales fueron registrados en el GenBank, bajo los números de acceso KX580042, KX580043, KX580044 y KX580045, respectivamente. En conjunto, los cuatro haplotipos mostraron 343 sitios conservados y 14 sitios polimórficos, entre los cuales 13 sitios fueron parsimoniosamente informativos y uno correspondió a una mutación única.
En los sitios polimórficos se encontraron 10 transiciones de tipo T?C, en las posiciones 15, 18, 21, 72, 123, 141, 189, 234, 276 y 312, tres transiciones de tipo A?G en las posiciones 111, 216 y 267, y una transversión de tipo T?A en la posición 270 (cuadro 2).
Distribución de haplotipos
El haplotipomás frecuente fue H1, seguido de H3 y H2. Los haplotipos H1 y H3 se encontraron en todos los municipios estudiados, el H2 se detectó en Sincelejo, Corozal y Guaranda, y el H4 se halló únicamente en el municipio de Sincelejo. La distribución de los haplotipos por municipio y la frecuencia de haplotipos, se muestran en la figura 2. La red de haplotipos inferida mostró a H1 como el haplotipo más antiguo, mientras que los demás haplotipos aparecieron como derivados suyos (figura 3). El haplotipo H3 se segregó de H1 por 12 pasos de mutaciones, en tanto que H2 y H4 se separaron de H1 por cuatro y dos pasos de mutaciones, respectivamente.

Figura 2 Frecuencias de haplotipos de nucleótidos del gen ND4 en cuatro poblaciones naturales de Aedes aegypti del departamento de Sucre, Colombia

Figura 3 Red de haplotipos de nucleótidos del gen ND4 en cuatro poblaciones naturales de Aedes aegypti del departamento de Sucre, Colombia. El área de cada círculo es proporcional a la frecuencia absoluta del haplotipo. Los círculos negros representan los pasos de mutaciones y, los círculos blancos, los haplotipos desaparecidos o no muestreados. La fracción de cada círculo indica la frecuencia de los haplotipos presentes en las poblaciones estudiadas.
Variabilidad genética, pruebas de neutralidad y AMOVA
En el cuadro 3 se presentan los valores estimados de los índices de diversidad genética y de las pruebas de neutralidad, para cada una y el total de todas las poblaciones de Ae. aegypti. Se destaca que los valores más altos de diversidad de nucleótidos y de haplotipos se encontraron en la población de Sincelejo y de Corozal.
Cuadro 3 Parámetros de diversidad genética y pruebas de neutralidad en cuatro poblaciones naturales de Aedes aegypti del departamento de Sucre, Colombia

NH: número de haplotipos en cada población; S: sitios polimórficos; k: número promedio de diferencias de nucleótidos; DH: diversidad de haplotipos; π: diversidad de nucleótidos
En las pruebas de neutralidad se encontraron diferencias estadísticas (p<0,05*), (p<0,01**), (p<0,001***) y (p<0,02++).
Por otro lado, la prueba de neutralidad D de Tajima arrojó valores positivos y significativos para el grupo de las cuatro poblaciones y para la población de Sincelejo. La prueba D de Fu y Li mostró valores positivos y significativos en cada población, en tanto que los valores obtenidos con la prueba F de Fu y Li fueron positivos y significativos para las poblaciones de Sincelejo, Sampués y Corozal, y para todas las poblaciones.
Respecto al AMOVA (cuadro 4), los resultados mostraron que el 74,02 % de la variación es atribuible a diferencias genéticas intrapoblacionales, mientras que las diferencias entre poblaciones contribuyeron con el 25,98 %. El índice de fijación estimado (F ST =0,259) indica la presencia de diferencias genéticas entre las poblaciones.
Estructura genética y flujo de genes
La estructura genética estimada mediante el índice de fijación (F ST ), y el flujo de genes calculado entre las cuatro poblaciones estudiadas, se muestran en el cuadro 5. Los valores del índice de fijación (F ST ) evidenciaron una gran diferenciación entre Sampués y Guaranda, Sincelejo y Sampués, Corozal y Sampués, y Corozal y Guaranda. Todos los valores del índice F ST fueron significativos, excepto el del obtenido al comparar las poblaciones de Sincelejo y Corozal.
El número efectivo de migrantes por generación entre poblaciones, indica que existe un número de migrantes que garantizan el intercambio genético entre las poblaciones de Sincelejo y Corozal, y las de Sincelejo y Guaranda.
Cuadro 5 Estructura genética (FST) y flujo de genes (Nm) entre cuatro poblaciones naturales de Aedes aegypti del departamento de Sucre
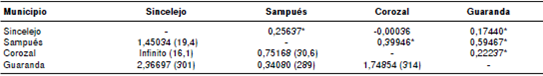
Entre paréntesis se presenta la distancia geográfica en km.
No se encontró correlación entre la distancia geográfica y la distancia genética en el test de Mantel (r=0,508; p=0,8477).
Discusión
En el presente estudio se detectaron cuatro haplotipos en las poblaciones de Ae. aegyptide los municipios de Sincelejo, Sampués, Corozal y Guaranda. Estas poblaciones compartieron tres haplotipos (H1, H2 y H3), con la excepción de la población de Sampués que solo compartió dos(H1 y H3).
El haplotipoH1, que fue el más frecuente, corresponde probablemente al más antiguo en el departamento de Sucre, como se puede inferir de su ubicación en la red de haplotipos (figura 2). Este haplotipo había sido identificado por Caldera, et al. 25, en los municipios de Sincelejo y Guaranda, y también está registrado en Bello, Antioquia 44, asícomo en Brasil 23,24, Perú 20 y México 19. Según Jaimes, et al. 44, el haplotipo H1 pertenece a un grupo ancestral en Colombia, el cual está relacionado con poblaciones de Ae. aegypti de Camerún, Guinea y Costa de Marfil.
El haplotipo H3, que fue el segundo más frecuente y estuvo presente en las cuatro poblaciones, también había sido reportado en Sucre 25, así como en Antioquia 44, Brasil 23,24, México 11 y Bolivia 18. Este haplotipo se encontraría estrechamente relacionado con poblaciones del mosquito de Tanzania 41.
El haplotipo H2, que no se encontró en la población de Sampués, está registrado en los municipios colombianos de Sincelejo 25, Bello, Riohacha y Villavicencio 44, y en países vecinos como Brasil 24 y Perú 20, pero se presume que se originó en África occidental 24.
Entre los cuatro haplotipos detectados en el departamento de Sucre, se destaca la presencia del haplotipo H4, que únicamente se encontró en la población de Sincelejo. Este haplotipo solo había sido registrado en Brasil 23,24, donde presenta una amplia distribución, por lo cual se presume que pudo haber surgido en ese país 24. El presente hallazgo constituye el primer registro de este haplotipo en Colombia y pone en evidencia su posible dispersión en América.
Comparado con el estudio realizado por Caldera, et al. 25, en la población de Sincelejo (localidades de El Cortijo y Botero) y Guaranda (localidades de San Camilo y Calle Tercera), en el presente trabajo se encontró un menor número de haplotipos, aunque cabe resaltar que las localidades de la población de Guaranda estudiadas no son las mismas, pero el efecto temporal de las recolecciones y los fenómenos de extinción local y recolonización de haplotipos más comunes en las poblaciones, pueden ser la causa de la ausencia o pérdida de los haplotipos H5 al H10 y de la aparición de nuevas variantes.
Con respecto a la población de Sincelejo, donde se analizaron más localidades y más especímenes que en el estudio previo, es importante resaltar que en esta ciudad, catalogada como ‘hiperendémica para dengue’ 45, los insecticidas son la estrategia más usada para controlar la enfermedad. Por este motivo, las poblaciones de insectos en este municipio están expuestas constantemente a una presión de selección por insecticidas, lo que podría llevar a que los haplotipos menos frecuentes tiendan a desaparecer, mientras que los haplotipos más frecuentes en la población disminuirían en número, pero se mantendrían en ella 10,12.
En estas circunstancias, se originarían procesos de extinción local y de colonización de las variantes que lograron permanecer en la población a pesar del control químico implementado para disminuir las poblaciones del vector. Es posible formular la hipótesis que el control con insecticidas pudo haber eliminado los haplotipos H4 y H5 encontrados por Caldera, et al. 25 en el año 2010 en la localidad de El Cortijo. Sin embargo, el no haber detectado estos haplotipos también podría atribuirse a las limitaciones del presente trabajo, que fueron la restricción en la cantidad de muestreos y el escaso número de ejemplares analizados.
Los valores de diversidad de haplotipos y de nucleótidos encontrados en este estudio (DH= 0,655; π=0,01631), fueron menores que los registrados en otros trabajos para la misma especie 22,44,46. Por población, los valores más altos de DH y π se observaron en Sincelejo (DH=0,686; π=0,01612) y Corozal (DH=0,630; π=0,01284).
Aunque en la población de Sincelejo esto se explicaría por la presencia de cuatro haplotipos, lo que en parte podría atribuirse al mayor número de individuos analizados en este municipio, es destacable la gran diversidad de la población de Corozal a pesar de que el número de individuos estudiados fue semejante al de Sampués y Guaranda. No obstante, los valores de DH encontrados en el presente estudio son similares a los registrados por Lima y Scarpassa 24 en poblaciones de la Amazonia brasileña (DH=0,666), y por Caldera, et al. 25 en el departamento de Sucre, Colombia (DH=0,6794).
Los resultados de las pruebas D de Tajima, y D y F de Fu y Li, permiten rechazar la hipótesis de neutralidad para estas poblaciones. Los valores de estos estimadores fueron positivos y significativos, lo cual indica que estas poblaciones están bajo selección positiva 47, en la que se favorecen los organismos que no presentan variación con respecto a las características promedio de la población, es decir, en relación con aquellos que están bien adaptados 48.
El AMOVA mostró que el componente que más pesó en la variabilidad encontrada, fue la variación intrapoblacional (74,02 %). Esta variación podría atribuirse principalmente a la gran diversidad de haplotipos encontrada en las poblaciones de Sincelejo y Corozal. La gran variación intrapoblacional observada indicaría que las poblaciones de Sincelejo, Sampués, Corozal y Guaranda están diferenciadas, es decir que parte de la variabilidad existente está restringida dentro de las respectivas poblaciones. Por otro lado, la menor variación interpoblacional (25,98 %) podría atribuirse a la gran frecuencia del haplotipo H1, presente en las cuatro poblaciones bajo estudio, y a la poca frecuencia del haplotipo H4, encontrado en la población de Sincelejo. El valor significativo del índice de fijación(F ST =0,25977; p<0,000) indica que existe diferenciación genética entre las poblaciones.
El análisis de la estructura genética de las poblaciones naturales de Ae. aegypti en Sincelejo, Sampués, Corozal y Guaranda, muestra la existencia de una diferenciación genética muy grande entre Sampués y Guaranda, gran diferenciación entre las poblaciones de Sincelejo y Sampués, las de Sincelejo y Guaranda, las de Corozal y Sampués, y las de Corozal y Guaranda, y una escasa o nula diferenciación entre las de Sincelejo y Corozal, según los criterios propuestos por Wright 49. La escasa diferenciación observada entre Sincelejo y Corozal demuestra que las frecuencias alélicas son iguales en estas poblaciones 50.
Esto puede ser efecto de la cercanía geográfica de estas dos poblaciones, por lo que es muy probable que compartan un acervo genético similar; además, la dispersión de tres kilómetros, aproximadamente, del mosquito, también puede influir en la similitud genética de estas poblaciones 51. Es posible que la diferenciación que se presenta entre las poblaciones de Sampués y Guaranda, de Sincelejo y Sampués, de Sincelejo y Guaranda, de Corozal y Sampués, y de Corozal y Guaranda, se deba a la ausencia del haplotipo H2 en Sampués y a la presencia de H4 en Sincelejo; pero también, a la frecuencia de los haplotipos en cada una de las poblaciones.
La estimación del flujo de genes reveló un restringido intercambio de genes entre las poblaciones. Sin embargo, el valor de Nm obtenido (2,36697) para el par de Sincelejo y Guaranda, evidencia un flujo genético entre estas poblaciones que geográficamente están separadas por una distancia de 301 km. Considerando lo anterior, es posible que el aparente flujo de genes entre estas dos poblaciones obedezca a la presencia de los tres haplotipos más frecuentes, en especial de H1 que puede ser la cepa más antigua del departamento, y que de este se derivaron los otros haplotipos, como se muestra en la red. Caldera, et al. 25, concluyeron que el H1 pudo ser uno de los primeros haplotipos introducidos y que, posteriormente, se diseminó en el departamento.
Otro aspecto importante es que la comunicación entre estas dos poblaciones es bastante restringida debido a su ubicación geográfica en zonas ecológicamente diferentes, por lo que el flujo de genes entre ellas sería inexistente: Sincelejo se ubica en la subregión Sabanas y, Guaranda, en la subregión Mojana. Asimismo, la presión selectiva ejercida por los insecticidas es diferente, pues en Sincelejo, considerado un municipio hiperendémico 45, se hacen constantemente aspersiones con insecticidas para disminuir las poblaciones del vector, mientras que en Guaranda, solo se asperja cuando se presenta un aumento significativo de casos 25.
En el par de poblaciones de Sincelejo y Corozal, el valor de Nm fue ‘infinito’, lo que demuestra que el flujo de genes es grande y que estas poblaciones se comportan como una gran población. Probablemente, esto se debe a dos factores, que son la corta distancia entre estas ciudades y la baja o nula diferenciación genética presente en ellas.
En conclusión, existen diferencias genéticas entre las poblaciones del mosquito Ae. aegypti de municipios del departamento de Sucre, sustentadas por los valores del F ST y el Nm encontrados entre los pares de poblaciones.
Se destaca la presencia del haplotipo H4 en la población de Sincelejo, el cual solo estaba registrado en Brasil, lo que implica múltiples reintroducciones de otras áreas geográficas adyacentes al departamento de Sucre y la costa Caribe.
Se recomienda evaluar si existen diferencias entre estas poblaciones en cuanto a su capacidad para infectarse y transmitir arbovirus, y la sensibilidad o resistencia a los insecticidas, teniendo en cuenta que Atencia, et al. 32 reportaron la presencia de la mutación F1534C en el gen kdr de Ae. aegypti en el municipio de Sincelejo, la cual confiere resistencia al piretroide permetrina y al organoclorado DDT.

















