Introducción
La salmonelosis es una enfermedad de alta prevalencia, con 1,2 millones de casos y 450 muertes por año, solo en Estados Unidos 1 y un estimado de un millón de muertes anualmente a nivel mundial2. El cuadro clínico en humanos incluye signos como gastroenteritis, náuseas, vómito, diarrea, cólico abdominal. Además, puede ocasionar fiebre, septicemia y es una causa potencial de muerte 1-5.
La infección por Salmonella spp. es causada por el consumo de comida y/o agua contaminada (transmisión fecal-oral), transmisión persona a persona, exposición ambiental o contacto con animales infectados de manera indirecta o directa 3,5-9. Debido a la habilidad de la Salmonella spp. para sobrevivir en productos cárnicos mal cocidos, estos se convierten en una de las vías principales de transmisión 10.
Las tortugas pueden ser portadoras asintomáticas frecuentes de Salmonella spp. Esta bacteria es comensal de su tracto gastrointestinal y es eliminada por la materia fecal del animal 5,11-13. En casos de estrés se pueden evidenciar signos, como enteritis o septicemia, lo cual se suele presentar en cautiverio 3,11,12. Se estima que cada año 93,000 personas se infectan de Salmonella spp. como resultado del contacto con reptiles en Estados Unidos 15, considerando los niños como la población en mayor riesgo. Las tortugas excretan la bacteria de manera intermitente en sus heces 5,8,16,17. También se puede encontrar en la superficie corporal de estos animales y en el agua en las que estas habitan 1,3,5.
La infección por Salmonella spp. ha sido poco reportada en tortugas en libertad 11,18,19. Dentro de los estudios realizados, pocos han demostrado diferencias en la presencia de la bacteria entre tortugas en libertad y en cautiverio.
No se ha reportado la prevalencia de Salmonella spp. en tortugas de río en la región del Urabá antioqueño, siendo esta una zona de alto riesgo por las condiciones ambientales y socioculturales que presenta; como la tenencia ilegal de especies silvestres como mascotas, practicas recreativas de la comunidad en el hábitat natural de estos reptiles y el consumo de carne y huevos de tortuga. Además, es una de las regiones más biodiversas de Colombia, alberga al menos tres especies diferentes de tortugas de río y una variedad de fuentes hídricas; esto la hace susceptible a la alta extracción ilegal de animales silvestres y el uso de estas especies en sus prácticas culturales.
En este contexto, el objetivo de este estudio fue determinar la presencia de Salmonella spp. en tortugas de río, tanto en libertad como en cautiverio en el Urabá antioqueño.
Materiales y métodos
Consideraciones éticas
Esta investigación fue aprobada por el Comité Institucional para el Uso y Cuidado de los Animales (CICUA) de la Universidad CES, en el acta 11 del 18 de diciembre del 2014.
Localización
Los puntos de toma de muestras se ubicaron dentro del municipio de Carepa (Urabá, Antioquia), zona de bosque húmedo tropical con cuatro ríos dentro de su territorio y una pluviosidad promedio anual de 2.000 mm. Altitud promedio de 28 msnm y temperatura entre 20 y 35°C 18; (Figura 1).
Población y toma de muestras
Estudio transversal prospectivo. La población de estudio incluyó un total de 105 individuos, divididos en dos categorías, en cautiverio (n = 55) y en libertad (n = 50). Los individuos en cautiverio se encontraban en la Estación de paso de Fauna Silvestre en el Municipio de Carepa (-76,6627; 7,7727), y provenían de entregas voluntarias o decomiso por su tenencia ilegal. Para la población en libertad, se realizó la captura de los individuos por medio de una red, en dos ríos del Municipio (-76,8194; 7,8266). Los individuos de esta población fueron marcados en el caparazón mediante una señal realizada con lima industrial para evitar recaptura e inmediatamente fueron retornados al sitio de captura posterior a la toma de la muestra.
Se obtuvieron datos de cada individuo, especie (Trachemys venusta, Rhinoclemmys melanosterna, Kinosternon leucostomum), sexo (hembra, macho o indefinido), peso (en kilogramos), tamaño (largo y ancho del caparazón en centímetros) y edad (juvenil, adulto o indefinido). Se realizó lavado del área cloacal con abundante agua y se desinfectó con alcohol, se tomó la muestra de materia fecal por medio de un hisopado cloacal (Figura 2A y 2B), posteriormente el hisopo se introdujo en un medio de transporte (500 μL de Caryblair®), y fue refrigerado a 4°C hasta el momento del procesamiento.

Figura 2 Descripción de procedimientos en el desarrollo del estudio. (A) Mediciones de tortuga palmera (Rhinoclemmys melanosterna) para identificación del individuo y edad. (B) Introducción cloacal de hisopo estéril en una tortuga caja (Kinosternon leucostomum) para toma de muestra. (C) Izquierda: medio de cultivo Hecktoen con colonias no fermentadoras punto negro productoras de ácido sulfídrico, sospechosas de ser Salmonella spp. Derecha: medio de cultivo McConkey con colonias no fermentadoras sospechosas de ser Salmonella spp. (D) Electroforesis en gel de agarosa al 1,5% que muestra las amplificaciones obtenidas para la banda de 854 pb del gen hilA específico del género Salmonella spp. Línea 1: Marcador de peso molecular 100 pb (Fermentas®); línea 2: Control negativo; líneas 3-16: Muestras 21 a 34 negativas para el gen hilA; línea 17: Muestra 35 positiva para el gen hilA; Líneas 18 y 19: Controles positivos.
Procesamiento de muestras en laboratorio
Cultivo microbiológico. Las muestras de materia fecal se sembraron en caldo Selenito (Becton Dickinson, Franklin Lakes, NJ, USA) y se incubaron durante 4 horas a 37°C. Posteriormente, se sembraron 10µL del caldo selenito en agar Hecktoen y Mcconkey (Becton Dickinson, Franklin Lakes, NJ, USA) y los agares se incubaron a 37°C de 18 a 24 horas, de acuerdo a lo descrito14 (Figura 2C).
Extracción de ADN. La extracción de ADN se realizó a partir de 500 µl del cultivo pre enriquecido en caldo selenito, utilizando un método comercial (The PowerFecal® DNA Isolation Kit, MO BIO), según indicaciones del fabricante.
También se realizó extracción de ADN por el método de ebullición a partir de colonias compatibles con Salmonella spp. obtenidas del cultivo en agar McConkey (medio selectivo y diferencial para enterobacterias) y XLD (medio selectivo diferencial para crecimiento de Salmonella). Para esta extracción se tomaron colonias fermentadoras y no fermentadoras, las cuales se mezclaron con 100 µLl de agua destilada estéril y se llevaron a 100°C durante 10 minutos; esto se centrifugó a 14.000 rpm durante 5 minutos; finalmente se tomó el sobrenadante y se usó para la realización de PCR12. Posteriormente, se cuantificó el ADN extraído y se almacenó hasta su procesamiento a -20°C.
Reacción en cadena de la polimerasa. Se realizó PCR del ADN extraído, se amplificó el gen hilA, específico de Salmonella spp. Los iniciadores usados son los siguientes:
hilA US 5’- GCATGGATCCCCGCCGGCGAGATT GTG-3’
hilA DS 5’- CGGAAGCTTATTTGCGCCATGCTGAGGTAG-3’ para un producto de amplificación de 854 pb.
Se usaron 14 µl de supermix, (Platinum® PCR SuperMix. Kit x100rxns. Invitrogen), 6 µL de agua destilada, iniciador forward y reverse 0,5 µL 10 μM de cada uno y 4 µl de ADN de cada muestra. Luego se utilizó un termociclador (Biorad, Hercules, California, USA) con el siguiente protocolo: Un ciclo de desnaturalización inicial a 95°C por 5 minutos, luego 30 ciclos a 94 grados 1 minuto, 65 grados 1 minuto y 72 grados 1 minuto; finalmente ciclo de extensión a 72°C por 10 minutos. El ADN amplificado se almacenó a - 20°C hasta la electroforesis.
Electroforesis. 12 µL del ADN amplificado fueron cargados en un gel de agarosa al 1,5% teñido con bromuro de etidio. El gel se corrió durante una hora y treinta minutos a 100 voltios en buffer TBE (tris, ácido bórico, EDTA) 1X. Finalmente, se visualizó el gel en un transiluminador (UVP); (Figura 2D).
Análisis estadístico. La información fue incluida en una base de datos en Microsoft Excel® y se realizó un análisis descriptivo en SPSS 21.0 (Licencia Universidad CES). La ubicación de los lugares de muestreo se realizó con ArcMap 10.2 (Licencia Universidad CES).
Resultados
En el estudio se incluyó una población de 105 tortugas, distribuidas en dos poblaciones: cautiverio y libertad, representando la primera un 52,3% del total de la muestra.
En la población en general se encontró una proporción igual de machos y hembras. Se encontró una mayor proporción de individuos adultos (n= 75, 71,4%), a comparación de los juveniles (n= 30, 28,6%). Tres especies de tortugas fueron incluidas en el estudio, todas con distribución natural en la región: R. melanosterna (n= 63; 60%), T. venusta (n= 28, 26,7%) y K. leucostomum (n = 14, 13,3%), como se puede apreciar en la Figura 3.

Figura 3 Distribución de las especies incluidas en el estudio según su estado: libertad y cautiverio.
La distribución entre cautiverio y libertad presentó una proporción similar en R. melanosterna (50,8% y 49,2%, respectivamente). Por el contrario, la especie K. leucostomum presentó un porcentaje de positividad mayor en libertad que en cautiverio (85,7%) y la especie T. venusta mayor en cautiverio que en libertad (75%) (Figura 3).
No se presentó ningún individuo positivo a la presencia de Salmonella spp. en las 50 tortugas en libertad muestreadas. Por el contrario, en la población de en cautiverio se presentaron dos individuos positivos a Salmonella spp. por lo tanto, la proporción de positividad en cautiverio es 3,6%. Ambos individuos machos adultos, pertenecientes a la especie R. melanosterna, como se observa en la Tabla 1.
Tabla 1 Descripción de las características de la población de estudio de acuerdo con el resultado de PCR para Salmonella spp.
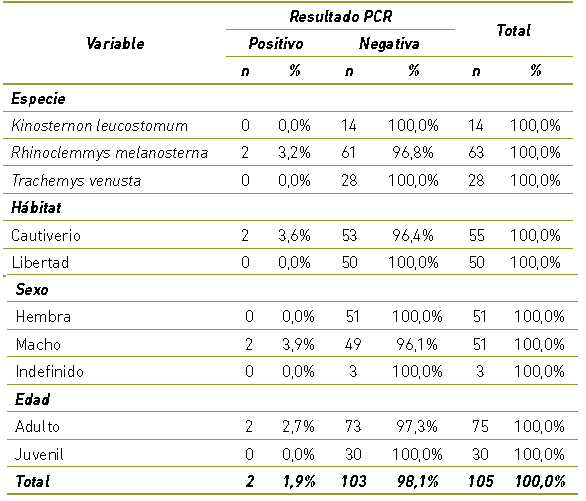
La proporción de positividad de Salmonella spp. en tortugas de río (cautiverio y libertad) en el Urabá antioqueño es de 1,9%.
Discusión
La presencia de Salmonella spp. en reptiles ha sido ampliamente descrita, resaltando que pueden ser portadores en su tracto gastrointestinal 5,11-13, y aproximadamente 6% de las infecciones por Salmonella spp. en humanos se adquieren por contacto directo o indirecto con reptiles 3, aunque también se suelen infectar por ingestión de alimentos o agua contaminados con materia fecal 1,3,21. Las mascotas, incluyendo reptiles bajo condiciones de cautiverio, han mostrado ser una fuente de infección de Salmonella spp. para los humanos 6,14,16 y su prevalencia ha sido estudiada en diferentes países 14,17,21, mientras que la presencia de Salmonella spp. en tortugas silvestres en libertad ha sido poco estudiada 11,18,19.
La frecuencia detectada en este estudio fue menor a la reportada en una investigación realizada en otro centro de atención de fauna en Antioquia (Colombia), donde encontraron que 35% (n = 110) de las tortugas semi-acuáticas en cautiverio eran positivas a la presencia de Salmonella spp14, utilizando el mismo método sobre las muestras incluidas en el presente reporte. También, la proporción se encuentra por debajo de otros reportes; Italia (36,8%) 22, Corea (30% incluyendo tortugas y otros reptiles) 23, Taiwan 24,3% (97/400) 24 y Japón 72,2% (13/18) 21.
Por el contrario, el número de casos en tortugas de río en cautiverio, fue mayor a la encontrada en un estudio realizado en un zoológico de Brasil, donde evaluaron por medio de hisopado cloacal 39 individuos adultos de la especie Trachemys scripta elegans, que llevaban 7 años en cautiverio y no se detectó la presencia de Salmonella spp. en heces 16. La presente investigación no incluyó datos acerca del tiempo en cautiverio, ya que las especies involucradas en el estudio provienen de tráfico ilegal y la información no es confiable. Sin embargo, se considera que el tiempo de cautiverio, puede influenciar el periodo de exposición, y, por lo tanto la potencial infección de las tortugas ex situ.
Respecto a la población en libertad, no se encontró presencia de Salmonella spp. arrojando resultados similares a los obtenidos en una población de tortugas silvestres en Estados Unidos donde no se detectó ningún individuo positivo (0/94) 19. Contrario a esto, en España se reportó una prevalencia del 11% de Salmonella spp. en tortugas en libertad incluyendo especies nativas y exóticas 8. Los resultados encontrados no excluyen la posibilidad de infección, debido a que puede haber intermitencia en la excreción de la bacteria 8,16,17. La toma de muestras consecutivas a los individuos sería una recomendación para investigaciones futuras, teniendo en cuenta consideraciones éticas con individuos en libertad y la tenencia en cautiverio por unos días para la obtención de estas muestras seriadas. Algunos falsos negativos pueden estar asociados al hisopado cloacal, el cual no permite una cantidad de muestra considerable, siendo insuficiente en la detección de la bacteria 8,19, además de la excreción intermitente.
Las diferencias de prevalencia pueden estar asociadas a factores epidemiológicos en las áreas estudiadas y a la sensibilidad de las pruebas utilizadas 14. La detección es altamente dependiente de una adecuada selección del procedimiento de muestreo combinado con el método de detección más sensible 5,8. Con el propósito de identificar la presencia de la bacteria de manera indirecta es posible considerar un muestreo a las fuentes de agua en la cuales sean capturadas, y disminuir la probabilidad de falsos negativos a causa del método de muestreo 8,25.
La variación de la presencia de Salmonella spp. entre poblaciones en cautiverio y libertad, puede deberse a la potencial excreción de la bacteria en situaciones de estrés, como es la extracción del hábitat y el mantenimiento bajo condiciones de vida no naturales. Sin embargo, otra hipótesis planteada por algunos autores se refiere a que los reptiles no son portadores naturales, pero se infectan estando en cautiverio 8,19.
Este estudio presenta evidencia de la presencia de Salmonella spp. en tortugas en la región de Urabá. Esta es una zona en la cual culturalmente se consume carne, vísceras y huevos de tortugas, todas ellas producto de la caza ilegal, por lo tanto plantea la posibilidad latente de transmisión a través de estas prácticas, e incluso por contacto directo con los individuos. Se considera necesario dar continuidad al estudio de este potencial patógeno en los reservorios y su relación con casos de salmonelosis no tifoidea en esta región del país en humanos, convirtiéndose en una herramienta y un argumento para desincentivar la tenencia de fauna silvestre en cautiverio en la zona y sus alrededores.
Conclusiones
Este estudio evidencia la presencia de Salmonella spp. en tortugas en cautiverio en un municipio del Urabá Antioqueño. Sin embargo, se requieren investigaciones posteriores donde se evalúe la intermitencia en la excreción de la bacteria, realizando muestreos seriados en la misma población de tortugas y que permitan determinar la posibilidad de infección durante condiciones de cautiverio, y con ello el potencial vínculo zoonótico con sus tenedores producto del tráfico ilegal de estas especies.















