INTRODUCCIÓN
La anemia infecciosa aviar-AIA (chicken infectious anemia) es causada por el virus de la anemia infecciosa aviar (chicken anemia virus, CAV) y ocurre en aves de menos de 2 semanas de edad sin anticuerpos maternos 1. El CAV se transmite tanto vertical como horizontalmente 2, se excreta a través de las heces 3 y probablemente también a través de las plumas 4. El virus infecta los hemocitoblastos y linfoblastos de células T 5,6, lo cual genera inmunosupresión 7 y puede permitir la entrada de otros agentes infecciosos 8.
El CAV es uno de los diez miembros del género Gyrovirus dentro de la familia Anelloviridae9. Su genoma de 2.3 kb es circular, con ADN monocatenario negativo 1. Codifica un ARN policistrónico 5) con tres marcos abiertos de lectura para tres proteínas virales: VP1, VP2 y VP3 10. El fragmento que codifica el gen VP1 se caracteriza por su alta variabilidad 2,11, lo que lo hace útil para la caracterización y el análisis filogenético del CAV 12. Aunque el virus ha sido detectado y caracterizado en varias partes del mundo 1, en Colombia no se han realizado estudios al respecto a pesar de que las gallinas reproductoras son vacunadas antes de la postura.
Debido a la falta de información en el país, este estudio tuvo como objetivo identificar la presencia del CAV, realizar una caracterización filogenética de las cepas circulantes y determinar los niveles de anticuerpos en granjas comerciales de aves ponedoras de diferentes edades y en aves de traspatio en el oriente y norte de Antioquia, Colombia.
MATERIALES Y MÉTODOS
Área de muestreo. Entre 2014 y 2015 se tomaron muestras de sangre y plumas de aves ponedoras Lohmann Brown e Isa Brown clínicamente sanas y sin antecedentes de haber sido vacunadas contra el CAV, et virus objeto del estudio. Las aves pertenecían a cuatro granjas avícolas ubicadas en el departamento de Antioquia, Colombia. En cada granja se eligieron al azar tres aves de seis edades distintas (1, 15, 30, 60, 90 y 120 días de edad). También se incluyeron muestras de aves de traspatio provenientes de predios situados cerca de las granjas del estudio. El método de muestreo fue por conveniencia, y no fue posible tomar muestras de todas las aves a los 90 días de edad ni de todas las aves de traspatio. Los procedimientos y métodos con animales que se utilizaron en el estudio fueron aprobados por el Comité de Ética para la Experimentación con Animales de la Universidad de Antioquia (Acta 99 de 2015).
Detección de anticuerpos contra el CAV por ELISA. Para identificar presencia de anticuerpos contra el CAV en los animales, se utilizó un kit comercial (IDEXX®, Estados Unidos). Las muestras de sangre se centrifugaron a 2000 xg durante 5 minutos para obtener las fracciones de suero, que se diluyeron en una relación 1:100 para la prueba. La lectura de los resultados se realizó por espectrofotómetro (BioTek®. Vermont, Estados Unidos) a una longitud de onda de 650 nm.
Los procedimientos y análisis se realizaron siguiendo las instrucciones del fabricante (IDEXX®, Estados Unidos). Para calcular los resultados, se empleó la relación entre las lecturas de densidad óptica de las muestras y la del control negativo (S/N). Las muestras con valores inferiores a 0.2 se definieron como positivas y aquellas de valores superiores a 0.8 se definieron como negativas. Se estableció una relación entre el resultado, los títulos correspondientes y la protección. Se consideró que títulos muy bajos (por debajo de 1000) no conferían protección; títulos moderados (entre 1000 y 8600) conferían una protección baja, y títulos altos (superiores a 8600) proporcionarían una protección alta 13.
Detección del CAV en muestras de sangre y plumas por PCR. El ADN de las muestras de sangre se extrajo utilizando un kit comercial (Qiagen®, Estados Unidos) siguiendo las instrucciones del fabricante. Para extraer el ADN de las plumas, se utilizó el mismo kit con un protocolo modificado: se cortaron los folículos de plumas en fragmentos de aproximadamente 2 mm, se mezclaron con 180 μL del tampón ATL y 20 μL de proteinasa K. La mezcla se incubó a 56°C durante una hora, agitándola cada 15 minutos. El ADN obtenido se almacenó a -20°C.
Se analizaron un total de 81 muestras de ADN de sangre completa (3 granjas con 18 muestras cada una, 1 granja con 17 muestras, y 10 muestras de aves de traspatio), 29 muestras de ADN de plumas (6 grupos por granja y 5 grupos de aves de traspatio) y 78 muestras de suero sanguíneo (3 granjas con 18 muestras cada una, 1 granja con 17 muestras, y 7 muestras de aves de traspatio).
Para la detección, se llevó a cabo PCR para amplificar un fragmento de 713 pb del gen que codifica la proteína CAV VP2, con los cebadores 5-GCG CAC TAC CGG TCG GCA GT-3 y 5-GGG GTT CGG CAG CCT CAC ACT AT-3 (14). Se realizó la amplificación en tampón de PCR que contenía 2 mM de MgCl2, 0.4 mM de desoxinucleótido 5'-trifosfato, 0.4 pM de cada cebador y 5 U de Taq ADN polimerasa (ThermoFisher Scientific®, Estados Unidos) en un volumen final de 25 μL. La reacción se llevó a cabo en un termociclador automatizado (MJ Research®, Massachusetts, Estados Unidos) con el siguiente perfil térmico: desnaturalización inicial a 94°C durante 5 minutos, seguida de 34 ciclos de desnaturalización, hibridación y extensión a 94°C durante 1 minuto, 63°C durante 1 minuto y 72°C durante 1 minuto, respectivamente.
PCR dirigida al gen VP1. En las muestras positivas para el CAV, se realizaron ensayos de PCR para amplificar de manera independiente dos fragmentos del gen que codifica VP1. Uno de ellos utilizó el siguiente par de cebadores: directo 5-GAC TGT AAG ATG GCA AGA CGA GCT C-3 e inverso 5-GGC TGA AGG ATC CCT CAT TC-3 para amplificar un fragmento de 675 pb (15). El otro utilizó el siguiente par de cebadores: directo 5-AGC CGA CCC CGA ACC GCA AGA A-3 e inverso 5-TCA GGG CTG CGT CCC CCA GTA CA-3 para amplificar un fragmento de 1390 pb (14). Las reacciones se llevaron a cabo en tampón de PCR que contenía 2 mM de MgCl2, 0.4 mM de desoxinucleótido 5'-trifosfato, 0.4 pM de cada cebador y 5 U de Taq ADN polimerasa (ThermoFisher Scientific, Estados Unidos) en un volumen final de 25 μL.
Las condiciones de PCR para la amplificación del fragmento de 675 pb fueron las siguientes: desnaturalización inicial a 95°C durante 5 minutos, seguida de 30 ciclos de desnaturalización, hibridación y extensión a 95°C durante 30 segundos, 50°C durante 30 segundos y 72°C durante 1 minuto, respectivamente. Por otra parte, las condiciones de PCR para la amplificación del fragmento de 1390 pb fueron las siguientes: desnaturalización inicial a 94°C durante 5 minutos, seguida de 34 ciclos de desnaturalización, hibridación y extensión a 94°C durante 1 minuto, 56°C durante 1 minuto y 72°C durante 2 minutos, respectivamente.
RFLP para la caracterización de cepas. Los productos de PCR del fragmento de 675 pb del gen VP1 de las muestras del estudio y de la cepa vacunal Cux-1 se digirieron independientemente con endonucleasas: Dde I (HpyF3I), Hae III (BsuRI), Hha I, Mbo I, Hpa II y Hinf I (ThermoFisher Scientific®, Estados Unidos) 15,16. La reacción en un volumen final de 32 μL contenía 10 μL del producto PCR, 1X del tampón recomendado para cada enzima y 20 U de la enzima. La reacción se incubó a 37°C durante 6 horas y se desnaturalizó a 80°C durante 30 minutos. Los productos digeridos se analizaron y separaron por electroforesis con tampón TBE 1X en gel de poliacrilamida al 15% y se tiñeron con el Silver Kit (ThermoFisher Scientific®, Estados Unidos) siguiendo las recomendaciones del fabricante. Se determinó el número de bandas y su peso a partir de los marcadores de peso molecular de 50 pb y 100 pb (GeneRuler DNA Ladder, ThermoFisher Scientific®, Estados Unidos).
Análisis filogenético. Las secuencias M5S3_N_ CO_2015, D5S2_N_CO_2015 y C+_vaccine_ Cux_1_RegICA8649BV derivadas del gen VP2 se editaron y ensamblaron utilizando Geneious® 9.1.8 (Biomatters Ltd®, Nueva Zelanda). Después, se alinearon entre sí y con respecto a secuencias homólogas disponibles en GenBank, incluida la secuencia de la cepa CAV Cux-1 (NC_001427.1) como referencia (únicamente se incluyó el fragmento correspondiente a la longitud de las secuencias obtenidas en este estudio), utilizando el algoritmo ClustalW integrado en el software MEGA 7.0 17. Se identificaron las posiciones de los nucleótidos y aminoácidos según las secuencias de referencia NC 001427.1 y NP 056774.1.
Con el fin de establecer la relación entre las secuencias obtenidas en el presente estudio y otras secuencias de cepas circulantes a nivel mundial, se construyó un árbol filogenético mediante el software MEGA 7.0, utilizando el método de máxima verosimilitud (maximum likelihood, ML) con el modelo de sustitución nucleotídica de Kimura de 2 parámetros 17,18. Se usó el método de soporte bootstrap con 1000 réplicas para establecer la confianza de la topología del árbol. Las secuencias nucleotídicas generadas en el presente estudio fueron despositadas en la base de datos GenBank.
RESULTADOS
Amplificación de CAV VP2 a partir de muestras de sangre mediante PCR. De acuerdo con los resultados de la PCR de amplificación del gen VP2, el 84% (68/81) del total de muestras analizadas fueron positivas para la detección del CAV. Se encontraron aves CAV-positivas en todas las edades: el 92% (11/12) de las aves de 1 día; el 83% (10/12) de las aves de 15, 30 y 60 días; el 82% (9/11) de las aves de 90 días; y el 100% (12/12) de las aves de 120 días. Respecto a las aves de traspatio, el 60% (6/10) arrojó resultados positivos para el CAV (Tabla 1).
Tabla 1 Resultados de PCR del fragmento VP2 y serología.
| Edad | PCR del gen VP2 (713 pb) | Serología para CAV | ||||||
|---|---|---|---|---|---|---|---|---|
| PCR en sangre | PCR en plumas | S/N <0.2a | S/N 0.8-0.2b | |||||
| 1 día | 92% | (11/12) | 100% | (4/4) | 50% | (6/12) | 17% | (2/12) |
| 15 días | 83% | (10/12) | 50% | (2/4) | 8% | (1/12) | 25% | (3/12) |
| 30 días | 83% | (10/12) | 75% | (3/4) | 0% | 0 | 17% | (2/12) |
| 60 días | 83% | (10/12) | 100% | (4/4) | 0% | 0 | 8% | (1/12) |
| 90 días | 82% | (9/11)* | 75% | (3/4) | 9% | (1/11) | 27% | (3/11) |
| 120 días | 100% | (12/12) | 25% | (1/4) | 50% | (6/12) | 17% | (2/12) |
| Aves de traspatio | 60% | (6/10)* | 40% | (2/5) | 43% | (3/7) | 29% | (2/7) |
| Total de positivos | 84% | (68/81) | 66% | (19/29) | 22% | (17/78) | 19% | (15/78) |
a Positivos con títulos de anticuerpos elevados; b Positivos con títulos moderados; *El muestreo se realizó por conveniencia, pero en estas granjas no fue posible muestrear 12 aves como en las demás granjas.
Amplificación de CAV VP2 a partir de muestras de plumas mediante PCR. El 66% (19/29) de las muestras de plumas resultaron positivas para el CAV. Se encontraron muestras de pluma de aves comerciales positivas en todas las edades. Las cifras de los resultados positivos fueron las siguientes: 100% (4/4) de las aves de 1 y 60 días; 50% (2/4) de las de 15 días; 75% (3/4) de las de 30 y 90 días; y 25% (1/4) de las de 120 días. Además, el 40% (2/5) de las aves de traspatio dieron positivo para el virus (Tabla 1).
Serología para CAV. Del total de muestras de suero analizadas, el 41% (32/78) resultaron positivas para anticuerpos contra el CAV. Hubo un 22% (17/78) de aves positivas con títulos de anticuerpos elevados y un 19% (15/78) con títulos moderados. Los porcentajes de animales que presentaron títulos de anticuerpos elevados contra el CAV fueron los siguientes: 50% (6/12) de las aves de 1 y 120 días, 8% (1/12) de las aves de 15 días y 9 % (1/11) de las aves de 90 días. Los porcentajes de aves que presentaron únicamente títulos de anticuerpos moderados fueron los siguientes: 17% (2/12) de las aves de 30 días y 8% (1/12) de las aves de 60 días. Del total de aves de traspatio analizadas, el 43% (3/7) presentaron títulos de anticuerpos elevados y el 29% (2/7) títulos moderados (Figura 1).

Figura 1 Porcentaje de animales CAV-positivos frente a animales con anticuerpos contra el virus (aves de 1 a 120 días de edad).
La línea azul denota el porcentaje de animales CAV-positivos en sangre por PCR. La línea azul claro denota el porcentaje de animales CAV-positivos en folículo de pluma por PCR. Las líneas verdes indican el porcentaje de aves con anticuerpos detectados por ELISA (títulos de anticuerpo elevados: S/N <0.2; títulos de anticuerpo moderados: S/N 0.8-0.2; títulos bajos o ausencia de anticuerpos: S/N>0.8).
Caracterización molecular de la cepa de CAV circulante en Colombia. Los resultados de la prueba de RFLP de las muestras colombianas mostraron un patrón que difería del de la cepa de control (cepa vacunal Cux-1)(Figura 2).

Figura 2 Caracterización por RFLP del control positivo (cepa Cux-1) mediante gel de poliacrilamida (15%) a través de las enzimas Hha I, Mbo I, Dde I, Hae III, Hinf I y Hpa II. Columna 1* y 12**: marcadores de peso molecular de 100 pb y 50 pb, respectivamente. Columnas 2, 3 y 11: posiciones de las bandas de los marcadores de peso molecular. Columnas 4 a 9: posiciones de las bandas de Cux-1 digeridas con endonucleasas. Columna 10: fragmento PCR de 675 pb del gen VP1 de la cepa vacunal Cux-1.
Se obtuvo un patrón de restricción como el de la cepa Cux-1 solo con las enzimas Hha I y Mbo I. Un patrón de restricción distinto al de Cux-1 se obtuvo con Dde I, Hae III, Hinf I y Hpa II. Cabe destacar que Hae III y Hpa II mostraron más cortes que Hinf I y Dde I (Tabla 2).
Tabla 2 Resumen de las bandas moleculares obtenidas por RFLP de las muestras estudiadas (M) en comparación con el control positivo (C+).

*: este resultado obtuvo el mismo número de bandas, pero su posición dentro del intervalo fue diferente.
Las secuencias parciales de nucleótidos del gen VP2 de las muestras positivas obtenidas en el estudio se identificaron como M5S3_N_CO_2015 y D5S2_N_CO_2015 y se depositaron en GenBank* con los números de acceso MF357710 y MF357711. Estas secuencias correspondían al CAV circulante en aves ponedoras de 90 días de edad en granjas ubicadas en el norte de Antioquia, Colombia. El control positivo de la reacción se identificó como C+_vaccine_Cux_1_RegICA8649BV y se depositó en GenBank* con el número de acceso MF357712. La longitud de estas secuencias, excluyendo las regiones de hibridación de los oligonucleótidos utilizados durante la amplificación por PCR, fue de 221 pb, localizadas entre los nucleótidos 640 y 860 del genoma de referencia. Se observaron dos grandes linajes en el árbol construido, que se identificaron como A y B (Figura 3). Las secuencias de Colombia pertenecían al linaje A junto con la mayoría de las secuencias analizadas, excepto Mhd8 y Mhd6 de Irán, que fueron las únicas secuencias situadas en el linaje B.

*ID de GenBank de las secuencias de este estudio: MF357710, MF357711, MF357712.
Figura 3 Análisis filogenético molecular de 26 secuencias de nucleótidos del CAV.
Árbol filogenético construido usando el método de máxima verosimilitud con el modelo de sustitución de nucleótidos de Kimura de 2 parámetros 18 en el software MEGA 7.0 17 con 1000 réplicas bootstrap. Se indican los valores relevantes de las ramas (≥60). Se identificaron dos clados (clado A y clado B); la mayoría de las secuencias pertenecían al grupo A. Las secuencias identificadas en las granjas estudiadas se indican con triángulos rosa (D5S2-N-Antioquia-CO-2015 y M5S3-N-Antioquia-CO-2015). La secuencia de control está denotada con un cuadrado azul (cepa vacunal C+_vaccine_Cux_1_RegICA8649BV). Las 26 secuencias disponibles en GenBank se identificaron con el número de acceso seguido del nombre, el país y el año.
Tres posiciones de nucleótidos fueron variables al comparar por pares las secuencias M5S3_N_ Antioquia_CO_2015, D5S2_N_Antioquia_ CO_2015 y C+_vaccine_Cux_1_RegICA8649BV. Cabe destacar que dos de las tres sustituciones fueron compartidas por las secuencias D5S2_N_ Antioquia_CO_2015 y C+_vaccine_Cux_1_ RegICA8649BV, y la otra sustitución fue compartida por las secuencias M5S3_N_ Antioquia_CO_2015 y D5S2_N_Antioquia_CO_2015 (Tabla 3).
Tabla 3 Cambios en aminoácidos de las secuencias seleccionadas de este estudio.
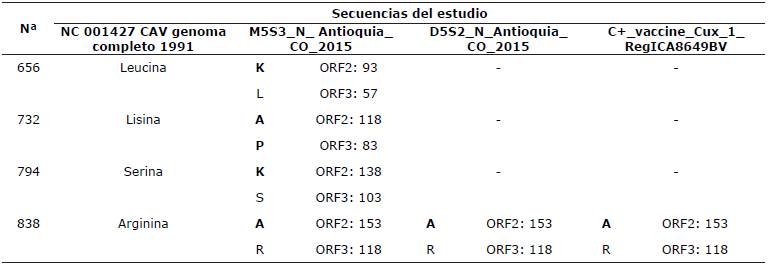
a N: nucleótido. Posición del cambio de nucleótido según la longitud de la secuencia de referencia del CAV (NC 001427) Los cambios se muestran según la longitud del CAV de referencia y su posición en cada marco abierto de lectura (open reading frame, ORF).
A nivel de aminoácidos, se produjeron cambios en las posiciones 93, 118, 138 y 153 del gen VP2 (K/L, A/K, K/S y A/R, respectivamente) y en la posición 83 del gen VP3 (P/K) con respecto a la secuencia de referencia (NC 001427). En particular, la secuencia M5S3 N Antioquia CO 2015 presentó una variación frente a las secuencias identificadas en este estudio y la que se empleó como referencia. Se encontraron variaciones menores en comparación con las demás secuencias analizadas, excepto en el caso de las secuencias de Irán y la secuencia del grupo externo. Los cambios en nucleótidos y aminoácidos se presentan en la tabla 3.
De acuerdo con la estimación de divergencia evolutiva, las secuencias colombianas D5S2_N_ Antioquia_CO_2015 y M5S3_N_Antioquia_ CO_2015 presentaron una distancia de 0,005 y 0,014 frente al control positivo (C+_vaccine_ Cux_1_RegICA8649BV) respectivamente, siendo menor o igual a la distancia encontrada entre este control y las cepas de otras partes del mundo, como Japón (VP2 VP3 VP1 JP 2003), Malasia (3 1 MY 2001; SMSC 1P9WT MY 2005), Brasil (BR265/00 BR 2004; BR91/99 BR 2004), China (TJBD40 CN 2004; JS China90 CN 2010), Estados Unidos (c0654p2d41v US 2003), Corea (CIAV89 69 KR 1991), Tailandia (BR6 11 TH 2011), Argentina (CAV 18 AR 2007) y Australia (SN0161NSW/chicken/Australia AU 2007). La mayor distancia se encontró al comparar con las secuencias de Irán (Mhd8 IR 2009; Mhd6 IR 2009) (Matriz de la estimación de divergencia evolutiva entre secuencias, como material suplementario).
DISCUSIÓN
Un número elevado de animales resultó positivo en la PCR para el CAV (84%), a pesar de no haber antecedentes de vacunación ni signos de enfermedad. La presencia del virus en muestras de sangre y tejidos se ha asociado a la migración de células linfoides infectadas al torrente sanguíneo y, posteriormente, a otros tejidos u órganos linfoides 19. A pesar de que esto evidenciaría una infección activa, el porcentaje de aves que dieron positivo en las pruebas de anticuerpos fue bajo (41%), posiblemente debido a la afectación del tejido linfoide previamente reportada 20 que dificulta la seroconversión, lo que hace que estas aves sean muy susceptibles de presentar la enfermedad.
Los resultados en aves de 1 día que mantuvieron la infección durante poco más de 15 días podrían explicarse suponiendo que pudieron adquirirla de la madre por vía vertical 21, ya que el virus invade y permanece en las gónadas 22 y en el tracto reproductor hasta 12 meses después de la infección sin presentar signos de la enfermedad y con transmisión del virus a la progenie 23.
Sin embargo, aunque se esperaba que el 100% de las aves de 1 día presentaran anticuerpos contra el CAV debido a la inmunidad pasiva (transferencia de anticuerpos de la madre), este porcentaje fue del 50%. Esto probablemente se asoció a una variación en los títulos de anticuerpos de la madre que afecta la transmisión a la progenie y las hace susceptibles al AIA 24. La seroconversión en las aves puede verse alterada por factores como el tipo de cepa, la alimentación, las condiciones de incubación, el estrés, las micotoxinas, los agentes infecciosos y los ambientes contaminados 25,26,27. Estas razones podrían favorecer la escasa transmisión de anticuerpos de las madres a la progenie21,28.
Teniendo en cuenta el periodo de infección a partir de la madre, las aves de edades posteriores a los 15 días pudieron reinfectarse por vía horizontal, ya que la probabilidad de adquirir el virus aumenta a medida que aumenta la edad 29 y la inmunidad pasiva decae a los 21 o 42 días 28. Lo anterior, junto con la afectación del tejido inmune 20, explicaría por qué tan solo unas pocas aves en las edades intermedias de 30 y 60 días presentaron anticuerpos a pesar de dar positivo al virus en sangre, lo que hace que las aves sean susceptibles a los efectos del virus y facilita la entrada de otros agentes como el virus de la bronquitis infecciosa 30 o el virus de la enfermedad de Marek 31. En particular, la presencia del virus de la enfermedad de Marek podría aumentar la susceptibilidad al CAV o a su infección persistente.
Las reinfecciones con el CAV o la reactivación del virus podrían estimular la producción de anticuerpos 2 semanas después de la infección 32,33, lo que daría lugar a que más animales dieran positivo en ELISA a edades avanzadas 34, como en el caso de este estudio que fue a los 120 días de edad. En esta etapa, el inicio de la madurez sexual podría favorecer la reactivación del virus debido a los cambios hormonales, lo que induce su replicación 22,35 y la posterior seroconversión. Sin embargo, se requieren más estudios para confirmarlo.
En cuanto a las aves de traspatio, no se esperaba encontrar animales positivos, ya que forman parte de pequeñas producciones y son criados para el consumo del propietario 36 y no cuentan con programas de vacunación. Sin embargo, un gran número de ellos tenía anticuerpos que teóricamente podrían neutralizar la cepa circulante, lo que pudo llevar a que solo un bajo porcentaje resultara positivo por PCR. Además, el virus circulante en estas aves podría ser vacunal o igual a la encontrada en este estudio, debido a que las aves comerciales que terminan su etapa productiva son vendidas y se convierten en aves de traspatio. Por otro lado, si estas aves no tuvieran anticuerpos, siendo susceptibles, podrían favorecer la propagación del virus en el campo 37. Por lo tanto, es fundamental caracterizar el CAV que circula en las aves de traspatio e identificar sus efectos en la producción avícola comercial, ya que pueden mantener el ciclo de transmisión del virus 38.
En cuanto al muestreo de plumas, éste ha sido útil para identificar el CAV a nivel poblacional, teniendo en cuenta que el virus se elimina por las heces únicamente hasta los 5 dpi 4,39. Además, es una técnica no invasiva que permite evaluar fácilmente la infección horizontal por CAV en las producciones avícolas 33,40. En consecuencia, una posible explicación para las aves de 1 día de edad que resultaron 100% positivas por pluma podría ser un entorno contaminado con el virus entre la eclosión y la llegada a la granja. En el caso de las aves de 90 y 120 días, los resultados por PCR fueron bajos, pero los porcentajes de ELISA fueron altos, esto permitiría pensar que los anticuerpos fueron capaces de detener la replicación del CAV en dichos animales. Sin embargo, el haber tenido resultados positivos por PCR en todas las edades, puede sugerir un fallo en las medidas de bioseguridad aplicadas en las granjas estudiadas 41 y una falta de seguimiento del estado sanitario, ya que las plumas funcionan como vehículo para diseminación del virus y lo mantienen hasta 32 días a 4°C 4,40. Por lo tanto, los resultados de PCR en pluma obtenidos en este estudio indican que el virus se encontraba en aves comerciales de todas las edades y también en aves de traspatio. Pero son necesarios más estudios para evaluar el papel de las plumas en la transmisión de la cepa identificada en condiciones naturales y su relación con los niveles de anticuerpos encontrados.
Igualmente, se deben evaluar las medidas de bioseguridad en estas granjas y se debe estudiar la vacunación como medida útil frente al CAV, especialmente en gallinas reproductoras, ya que esta es la primera inmunidad pasiva frente al virus que tendrá la progenie 42. Por otro lado, un diagnóstico a tiempo con las técnicas adecuadas favorecería la vigilancia sanitaria y evitaría pérdidas económicas por inmunosupresión 24.
Características de la cepa colombiana del CAV. Teniendo en cuenta las características de la infección, se espera que el virus transmitido de la madre a la progenie derive de la vacuna. En este trabajo, el patrón de restricción que presentaron las muestras mediante la prueba de RFLP fue diferente al de la cepa vacunal Cux-1; y esta diferencia podría afectar a la respuesta inmunitaria del animal 43. Esto concuerda con los resultados publicados por otros autores 44,45,46,47. De igual manera, se determinó que las enzimas útiles para diferenciar la cepa colombiana de la Cux-1 mediante RFLP son Dde I, Hha I, Hae III, Hinf I y Hpa II; algunas de las cuales han sido reportadas por otros autores 14,48. Se destaca la utilidad de Hae III y Hpa II debido al número de cortes que se pueden realizar, lo que aumenta la posibilidad de clasificar las cepas. Del mismo modo, la escasa información que puede aportar Hinf I se debe al bajo número de fragmentos que genera. Si bien el patrón obtenido con Mbo I para las muestras del estudio fue el mismo que para la cepa vacunal Cux-1, no se consideró que la cepa circulante correspondiera a Cux-1 debido a los resultados obtenidos con otras endonucleasas utilizadas en el estudio.
Pese a la limitación en la longitud del fragmento analizado, los resultados de la prueba de RFLP se ven respaldados por la secuenciación del ADN, lo que confirma que las cepas circulantes de las granjas del estudio no son 100% idénticas a la cepa (vacunal) Cux-1.
Esto se ve soportado por la distancia filogenética de las cepas identificadas, que fue menor con cepas que han causado brotes de CIA, por ejemplo, en Argentina 44,46. La distancia evolutiva que presentan las secuencias del CAV colombiano podría estar relacionada con los efectos que generan en animales, la capacidad de diseminación en cultivo celular 49 o el curso subclínico de la enfermedad 44,46. Por lo tanto, los cambios nucleotídicos de las cepas identificadas deben evaluarse más a fondo, junto con su patogenicidad y su capacidad para estimular la seroconversión, más aún cuando se asocia con otros agentes inmunosupresores 50.
En este sentido, se puede concluir que el CAV circulaba en las granjas estudiadas a lo largo de las diferentes etapas de crecimiento con baja seroconversión en los animales y que, de acuerdo con las secuencias obtenidas, el virus no fue idéntico a la cepa vacunal Cux-1 que se utiliza para la prevención del virus en Colombia.
Cabe mencionar que este constituye el primer reporte de CAV en Colombia utilizando técnicas moleculares, donde las diferencias moleculares entre la cepa colombiana y la vacunal podrían estar relacionadas con cuadros de inmunosupresión que podrían promover un curso subclínico de la infección. Esto debería ser objeto de más investigación, dado que, como hemos mencionado, el CAV tiene la capacidad de favorecer la entrada y permanencia de agentes secundarios como el virus de la enfermedad de Marek, así como llevar a una mala respuesta a la vacunación y a pérdidas económicas aún por determinar. Por último, se necesitan más estudios para profundizar en la información conocida sobre la cepa, su patogenicidad y las consecuencias económicas de su presencia en las granjas afectadas.















