INTRODUCCIÓN
Los hongos filamentosos o mohos se caracterizan por tener un soma vegetativo (talo) similar a las plantas, filamentos microscópicos continuos más o menos alargados y ramificados con paredes celulares definidas, la mayoría, constituidas por quitina, dispuestas en microfibrillas como la celulosa, además de otros polisacáridos como mananos, galactanos y quitosán reemplazan a la quitina en algunos grupos, con una la pared celular formada por carbohidratos (80-90 %), y son las proteínas, los lípidos, polifosfatos e iones inorgánicos el material cementante (Estrada et al., 2019).
Muchos hongos son beneficiosos, al degradar la materia orgánica compleja convirtiéndola a formas químicas simples que pasan a formar parte del suelo, absorbidas por otras generaciones de plantas, encargándose de la fertilidad de la tierra. Pese a los perjuicios que pueden causar estos organismos a otros seres vivos, incluyendo a humanos, son un grupo de organismos muy importantes y exitosos desde el punto de vista evolutivo trascendental para la vida en el planeta (Téllez et al., 2017; Mancilla et al., 2021).
Otros hongos filamentosos tienen potencial de aplicación biotecnológica en diferentes sectores productivos debido a su capacidad para producir antioxidantes (Smith et al., 2015), ácidos grasos polinsaturados, enzimas industriales, antibióticos, productos fermentados (Wakai et al., 2017), pigmentos que se utilizan en alimentos (Dufossé et al., 2014), antifúngicos, agentes hipolipidémico e inmunosupresores (Alberti et al., 2017), y debido a su capacidad metabólica y mecánica diferentes especies de mohos se utilizan para el desarrollo de biofertilizantes, biopesticidas y acondicionadores biológicos (Tellez et al., 2017). También, la necesidad de afianzar el crecimiento de los sistemas productivos agrícolas ha creado alterativas permitiendo avanzar y superar las deficiencias en procesos como la nutrición vegetal a partir del uso de fertilizantes de síntesis. Así, ha surgido la agricultura orgánica como una tecnología eficaz, respetuosa del medio ambiente, económica y factible de desarrollar, incluyendo la producción y uso de fertilizantes de origen microbiológico a partir de residuos agropecuarios para nutrir plantas, controlar plagas y enfermedades de plantas, e influir positivamente en las propiedades fisicoquímicas del suelo (Rojas, 2020).
Definir la identidad de las cepas es una fase insustituible en la investigación de cualquier agente biológico y la selección de las herramientas y mecanismo de identificación constituyen la confiabilidad de la información. El uso de la técnica de la Reacción de la Cadena de la Polimerasa (PCR) como mecanismo de identificación molecular se precisa debido a la fiabilidad y practicidad de la técnica para amplificar segmentos de ADN asistido por cebadores como los Espaciadores internos de transcrito (ITS) que conforman la región ITS que se caracteriza por ser altamente repetitiva dentro del ADNr, poseer regiones conservadas, tener evolución relativamente lenta, pero a su vez, contener regiones menos conservadas, propiedades otorgadas por ITS2, 5.8 S e ITS1 respectivamente (Froeschke at al., 2014); dichas características hacen de la región ITS un segmento significativamente de interés para estudios en taxonomía, incluso el espaciador interno de transcrito ha llegado a ser denominado por un grupo de micólogos como el marcador de barra para identificación de taxones y constituye uno de los mecanismos más confiables para determinar la identidad de mohos a nivel de especie (Bellemain et al., 2010).
Esta investigación determinó un mecanismo de identificación molecular para doce especies de hongos distribuidos en los géneros Trichoderma, Geotrichum, Purpureocillium, Laburnicola, Penicillium, Aspergillus, Beauveria, Bipolaris, Curvularia, y Alternaria. Además, se realizó una correlación filogenética y revisión de antecedentes que enlistan una serie de capacidades de las diferentes especies identificadas para futuras aplicaciones en diferentes áreas de biotecnología. También, importante tanto para el banco de cepas como para el laboratorio de biotecnología molecular de la sede campos Elíseos de la Universidad Francisco de Paula Santander que permitirá avances en procesos de docencia e investigación.
MÉTODO
Esta investigación se realizó en el Centro Experimental Campos Elíseos, de la Facultad de Ciencias Agrarias y del Ambiente de la Universidad Francisco de Paula Santander, ubicado en el municipio de Los Patios, Norte de Santander (Colombia) a una altura de 410 ms.n.m., y temperatura de 27 °C. Los aislamientos de los hongos conservados fueron caracterizados y procesados en los laboratorios Banco de Cepas y Biotecnología Molecular.
Extracción de ADN
Posterior al crecimiento en caldo Sabouraud, el micelio fue fraccionado con ayuda de una cuchilla de bisturí en trozos con diámetro no superior a 4 mm2, exceptuando las cepas HA002, HE004, HF002 y HF003 a las que el micelio se centrifugó a 5000 rpm durante 5 minutos, y se procedió utilizando los protocolos propuestos por Suárez (2016) y Blanco et al. (2021).
Obtención de los amplicones
Para la obtención de los fragmentos amplificados por medio de la PCR se utilizaron los cebadores ITS5 (5´-GGAAGTAAAAGTCGTAACAAGG-3´) e ITS4 (5´-TCCTCCGCTTATTGATATGC-3´) que amplificaron la región ITS en sentido y anti-sentido respectivamente, haciendo uso del protocolo propuesto por (Suárez, 2016).
Visualización y cuantificación de los amplicones de ADN
Para la visualización de los fragmentos de ADN amplificados se realizó en gel de agarosa al 1,5 % w/v y tampón TBE 1X, y las electroforesis se efectuó con un arranque de corrida de 120 voltios durante 5 minutos y posteriormente a 100 voltios durante 2 horas. Las muestras de ácidos nucleicos fueron marcadas con el intercalante gel red y revelado en el transiluminador y fotodocumentador ChemiDoc de Bio-Rad. Para la cuantificación de ADN se utilizó el espectrofotómetro NanoDrop de Thermo scientific (Suárez, 2005).
Secuenciación de amplicones
Los productos de PCR obtenidos de cada una de las cepas se enviaron a la empresa Isla SAS, (Corea del Sur) para la posterior decodificación de las secuencias amplificadas, utilizando el secuenciador 3730xl DNA Analyzer. Los resultados fueron recibidos en formato fasta y la lectura del secuenciador fue reflejado en un archivo por medio de un cromatograma.
Identidad por homología
A partir de las secuencias obtenidas, se realizó un análisis utilizando la base de datos del Centro Nacional de Información Biotecnológica (NCBI, 2019) mediante la herramienta Targeted Loci Nucleotide BLAST que permitió determinar la identidad de los organismos mediante la comparación de la secuencia target (mi secuencia de interés) con la base de datos específica para secuencias curadas y remarcadas de la región espaciadora transcrita interna (ITS). Para ello se cargó la secuencia en formato fasta en la interfaz del Blast Targeted Loci Nucleotide, se seleccionó la base de datos para ITS sin ninguna otra modificación de parámetro, y se dio la orden de cargar el análisis. Como resultado se obtuvo la información de las secuencias depositadas en la base de datos con alineaciones significativas, teniendo en cuenta especialmente dos parámetro para la elección de la identidad: el E-value, cuyo valor debe estar próximo a un puntaje de 0,0 y la identidad con un porcentaje del 90 al100 %.
Representación de la relación filogenética
Para la construcción del árbol filogenético se utilizó el software Mega 7,0; inicialmente, se realizó un alineamiento múltiple entre todas las secuencias incluida el outgrup (E. coli), se empleó el algoritmo de Muscle (Align by Muscle) en Mega 7,0, utilizando el método de agrupamiento UPGMA (Unweighted Pair-Group Method with Arithmetic); una vez culminado el análisis, el archivo que se generó fue guardado en formato Mega y se indicó que no correspondía a secuencias que codifican para proteínas, a partir del archivo de alineamiento múltiple se construyó el dendrograma en la opción phylogeny; para la reconstrucción de la filogenia se empleó el método estadístico de UPGMA, y el modelo Jukes-Cantor del software mencionado, logrando la representación del árbol inferido para la relación filogenética de las secuencias obtenidas en este estudio (Lessick, 2020).
RESULTADOS
Amplicones ITS mediante PCR
Con el protocolo utilizado en la PCR y con la implementación de los cebadores ITS4 e ITS5, se obtuvieron fragmentos de tamaños que variaron entre 0,5 y 0,8 Kilobases (Kb) (Figura 1), permitiendo un testeo de los amplicones obtenidos. A partir de este resultado se define lo que representa el fundamento de selección del proceso estándar para la identificación molecular de hongos filamentosos implementado por el Laboratorio de Biotecnología Molecular de la UFPS. Además, según González-Estrada et al. (2020), para la identificación molecular de Penicillium sp. fue también realizada por PCR, empleando las regiones internas transcritas ITS1-5.8S-ITS2, utilizando el par de iniciadores de secuencia ITS4/ ITS5, y los pares de base obtenidos se compararon con las secuencias reportadas en la base de datos del banco de genes de NCBI mediante el programa BLAST; además, Béjar et al. (2019), amplificaron ADN por PCR en tiempo real (qPCR), para identificar Aspergillus fumigatus.

Figura 1 Electroforesis en gel de agarosa al 1,5 % w/v, de los amplicones obtenidos a partir del ADN de hongos, utilizando ITS. M: Marcador molecular. 1, HA001; 2, HA002; 3, HA005; 4, HA006; 5, HA007; 6, HA008; 7, HD002; 8, HD008; 9, HD010; 10, HD011; 11, HE001; 12, HE002; 13, HE003; 14, HE004; 15, HF002; 16, HF003; 17, HF020; 18, HF021; 19, HI001; 20, HI002; 21, HI003; 22, HI004; 23, HI005; 24, JIF01; 25, JIF02; control (-); control (+), Baeauveria bass.
Determinación de identidad de hongos
Las 25 cepas se clasificaron en diez géneros identificados como Trichoderma, Geotrichum, Purpureocillum, Luburnicula, Penicillum, Aspergillus, Baeuveria, Bipolaris, Curvularia y Alternaria, distribuidos en doce (12) especies, de la siguiente manera: las cepas HA001, HA005 y HA006 corresponden a Trichoderma yunnanense; HA002, HE004, HF002 y HF003 son Geotrichum silvícola; HA007, HA008 y HE003 son Purpureocillium lilacinum; HD011, HI001, HI002, HI003, HI004 y HI005 corresponden a Penicillium citrinum; JIF01 y JIF02 son Alternaria destruens, la cepa HD002 es Laburnicola hawksworthii; HD008 es Penicillium chrysogenum; HD010 corresponde a Aspergillus fumigatus; HE001 a Penicillium rubens; HE002 es Beauveria bassiana; HF020 pertenece a Bipolaris sivanesaniana, y para finalizar HF021 es Curvularia pisi(Cuadro 1).
Cuadro 1 Identificación, valores de identidad y E-value de cada una de las secuencias analizadas utilizando la base de datos NCBI.
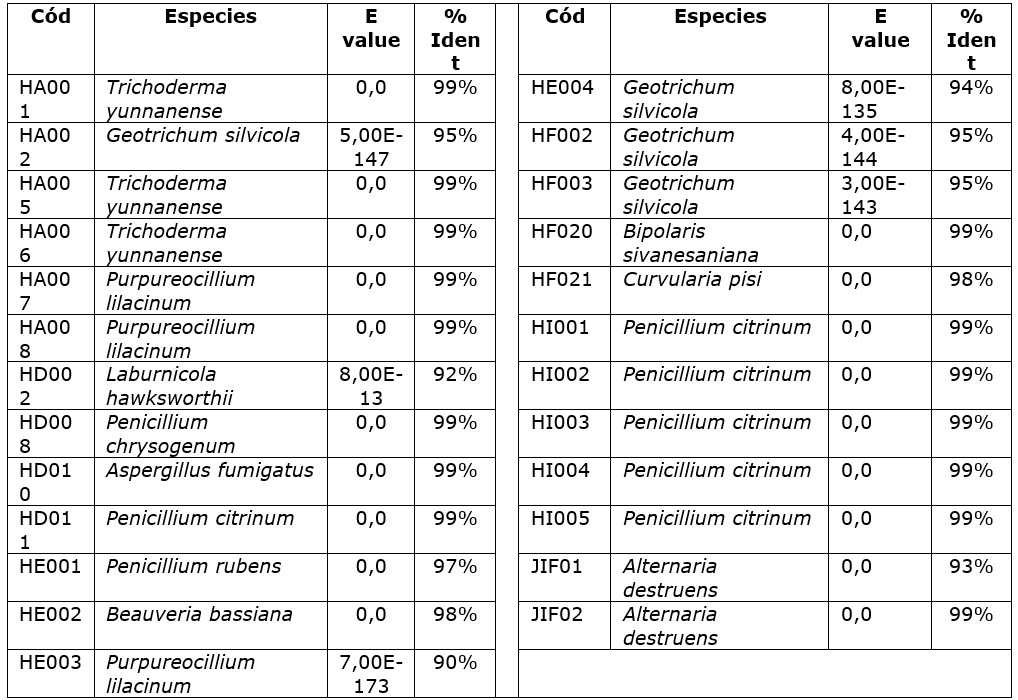
Los valores de identidad para todas las cepas superaron el 93 % y con E-value no inferior a 8x10-135, según los análisis realizados a las 50 secuencias utilizando la herramienta Targeted Loci Nucleotide BLAST exceptuando la cepa HD002 cuyo resultado fue inconcluso en la secuencia ITS4, mientras que con el ITS5 el valor de identidad alcanzó el 92 % relacionando la especie Laburnicola hawksworthii.
Se considera la importancia de los datos moleculares para estudios de biodiversidad y el potencial de la investigación micológica en países como Brasil y Colombia. Para el caso de Brasil se determinó la diversidad de hongos basada en las secuencias ITS con un límite del 98 % (Menolli, 2020), mientras que en este trabajo fue del 97 al 99 %.
Aunque los niveles de identidad son significativos y, a pesar de que la región ITS es considerada un código de barras para la identificación taxonómica de hongos (Melloni, 2020), el uso estándar de estos cebadores durante la PCR puede generar sesgos, incurriendo al error en la secuencia, generando una predisposición en la clasificación de especies dentro de las divisiones basidiomicetos y ascomicetos, dependiendo de los iniciadores utilizados (Bellemain et al., 2010). Por lo anteriormente señalado, se sugiere evaluar otros iniciadores para comparar los resultados y establecer un consenso mediante el análisis de las secuencias obtenidas por Béjar et al. (2019), logrando identificar diez cepas de Aspergillus fumigatus sensu stricto Involucradas en la aspergilosis invasiva (AI) en el Perú, contribuyendo a explicar la resistencia al tratamiento y evidenciando la importancia del ambiente en el incremento de los casos.
Análisis filogenético
El filo Ascomycota se caracteriza por su estructura sexual en forma de saco o asca que contienen las ascosporas y por la conjugación de hifas asimilativas por medio de la anastomosis, también se constituye como un grupo de hongos numeroso con aproximadamente 114.000 especies identificadas y distribuidas en diversos ecosistemas (Wijayawardene et al., 2021). Basados en la información extraída del repositorio de nomenclatura Index Fungorum (http://www.indexfungorum.org/names/Names.asp) la totalidad de las cepas identificadas en el presente estudio corresponden al filo Ascomycota, representando los subfilos Pezizomycotina y Saccharomycotina, enclaustrados en las clases Sordariomycetes, Eurotiomycetes, Dothideomycetes y Saccharomycetes, y asociados a los órdenes Hypocreales, Eurotiales, Pleosporales y Saccharomycetales. Las especies Trichoderma yunnanense, Purpureocillum lilacinum y Beauveria bassiana son hongos que se caracterizan por su efecto antagonista y son ampliamente utilizados para el control biológico (Hernández et al., 2019; Ruiz et al., 2020; Romero et al., 2020) y pertenecen al orden de los Hypocreales; Aspergillus fumigatus, Penicillium chrysogenum, Penicillium Rubens y Penicillium citrinum son especies productoras de metabolitos de uso farmacéutico y reconocidos reductores de azúcares de interés para la industria de los biocombustibles y hacen parte del grupo de los Eurotiales (Cabral et al., 2018; Ouephanit et al., 2019; Lima et al., 2019; Tejas et al., 2019; Lodha et al., 2020; Huber et al., 2020; Xianchun et al., 2020; Mondal et al., 2020); Bipolaris sivanesaniana, Laburnicola hawksworthii, Alternaria destruens y Curvularia pisi son especies vinculadas con efectos patógenos en diferencies especies vegetales (Manamgoda et al., 2014; Zhai et al., 2018) y son del orden de los Pleosporales; y en el orden de los Saccharomycetales se encuentra a Geotrichum silvícola.
En cuanto al cladograma (Figura 2) se puede inferir la relación entre los diferentes órdenes representado en las bifurcaciones que constituyeron clados entre Hypocreales - Eurotiales, Hypocreales - Pleosporales, Eurotiales - Pleosporales, Hypocreales - Eurotiales - Pleosporales - Saccharomycetales, respaldando los resultados obtenidos por Wang et al. (2009), que señalan la proximidad taxonómica entre los subfilos Pezizomycotina y Saccharomycotina basados en estudios de filogenia y que sugiere una filiación temprana entre los subfilos señalados.
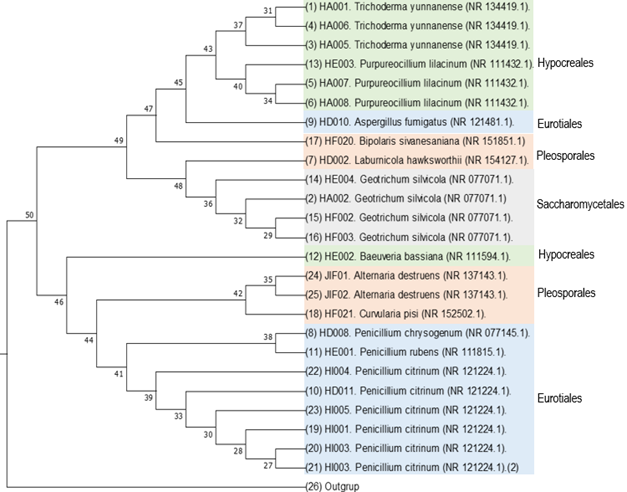
Figura 2 Cladrograma. Representación de la proximidad filogenética de las secuencias amplificadas de la región ITS de las especies identificadas.
A manera de una descripción textual de los resultados representados en el cladograma, se señala lo siguiente: el árbol se dividió en dos grandes ramas que corresponden a los nodos 25 y 50, el nodo 25 se ubicó el grupo externo (outgrup) que se seleccionó de una secuencia de E. coli, mientras que en el nodo 50 se generó la especiación de las 25 cepas identificadas. Del nodo 50 se ramificaron dos nodos superiores, el 49 y el 46: en el nodo 46 se produjo una bifurcación resultando los nodos 47 y 48, en el nodo 47 se presentó una especiación directa en el taxón Bipolaris sivanesaniana (nodo 17) y se generó el nodo 45 el cual también se bifurcó en dos clados representados en los nodos 43 y 9 en los que se ubicaron el orden de los Hypocreales y Eurotiales respectivamente; de los Hypocreales surgió la especiación de Trichoderma yunnanense (nodo 37) y Purpureocillum lilacinum (nodo 40) y en el nodo 9 se ubicó Aspergillus fumigatus, el nodo 48 se ramificó en los nodos 7 y 35 ubicando las especies Laburnicola hawksworthii y Geotrichum silvícola, respectivamente. El nodo superior 46 se ramificó en los nodos 12 y 44, en el nodo 12 se encontró el taxón Baeuveria bassiana que pertenece al orden de los Hypocreales, mientras que en el nodo 44 se formaron dos clados que corresponden a los nodos 42 y 41, en el nodo 42 se ubicaron especies del orden de los Pleosporales pero de los géneros Curvularia y Alternaria, y en el nodo 41 se agrupó el clado que corresponde al orden de los Eurotiales que pertenecen al género Penicillum; a su vez, en el nodo 42 se manifiestó la especiación de Curvularia pisi (nodo 18), y Alternaria destruens (nodos 24 y 25). Curvularia, tradicionalmente ubicada en el género Bipolaris, fue recientemente reclasificada en Curvularia, con base a análisis filogenéticos, incluyendo a B. australiensis, B. hawaiiensis y B. spicifera (Madrid et al., 2019).
Umaña et al. (2019), mediante un estudio de distancias génicas utilizando intermicrosatélites (ISSR), identificó distanciamiento entre cepas de la misma especie, lo que concuerda con lo hallad en esta investigación, pero difiriendo en el método utilizando e identificando diferentes especies. Para el caso de la especie Geotrichum silvícola, las cepas HF002 y HF003 demuestran mayor proximidad en comparación con las cepas HA002 y HE004; para la especie Trichoderma yunnanense se evidenció que las cepas HA001 y HA006 tienen mayor grado de parentesco en comparación con la cepa HA005; caso similar sucedió con la especie Purpureocillum lilacinum que reflejó un mayor grado de semejanza entre las cepas HA007 y HA008 comparada con la cepa HE003; para el género Penicillumi que presentó variedad de especies identificadas, se evidencia que las especie Penicillum chrysogenum y Penicillum rubens tienen mayor grado de parentesco al agruparse en el mismo nodo, mientras que Penicillum citrum mantuvo un mayor grado de semejanza con los de su misma especie, lo que evidencia que las variaciones genéticas entre aislamientos no dependen de las distancias geográficas (Umaña et al., 2019).
Potencial biotecnológico de los hongos identificados
Se realizó una recopilación bibliográfica en la que se describieron las capacidades reportadas por otros autores a cerca de cada género y especie de los hongos que se identificaron en esta investigación, buscando el potencial de las cepas para futuras aplicaciones.
Trichoderma es importante por su capacidad de adaptación y producción de metabolitos, como enzimas, compuestos promotores de crecimiento vegetal y compuestos volátiles, entre otros, de interés biotecnológico y ambiental. Este género es utilizado como agente de biocontrol contra hongos fitopatógenos debido a sus múltiples mecanismos de acción, destacando la antibiosis, el micoparasitismo, la competencia por espacio y nutrientes, y la producción de metabolitos secundarios. Varias especies de Trichoderma se han utilizado en sistemas acoplados de fermentación en sustratos sólidos o cultivos sumergidos, para generar energías alternativas como etanol. Los biorreactores como sistema de fermentación optimizan las condiciones del cultivo para favorecer la generación de biomasa y metabolitos (Hernández et al., 2019).
Purpureocillum lilacinum favorece el crecimiento, el contenido de clorofila y controla fitopatógenos como Botrytis que causan enfermedades del moho gris en una gran variedad de frutas y verduras, y la pudrición blanda y tizón foliar en la zanahoria (Ruiz et al., 2020). P. lilacinum es un nematófago que tiene capacidad ovicida contra Ancylostoma que es un parásito gastrointestinal (Menezes et al., 2017), también tiene la capacidad de ejercer biocontrol contra Meloidogyne incognita que es uno de los patógenos más importantes del pepino (Cucumis sativus L.) y ha demostrado su efectividad en el control de plagas como Meloidogyne javanica, Heterodera avenae Wollenweber, y Radopholus similis que afectan la plantaciones de tomate, cebada y banano respectivamente (Dahlin et al., 2019), además de la capacidad de solubilizar fosforo, producir sideróforos y ácido indol-3-acético (IAA) características de un candidato para uso como biofertilizante (Moreno et al., 2020).
Geotrichum silvicola es una especie de levadura artroconidial asexual que fue reportada en el año 2005 y, en la actualidad, la información relacionada con el potencial de sus aplicaciones es escasa, pero se ha encontrado que tiene estrecha relación genética con Galactomyces geotrichum, que es una especie con potencial en la industria de alimentos, confiriendo características organolépticas y nutricionales de sabor, aroma y ácidos grasos poliinsaturados (omega 3 y 6) (Grygier et al., 2019).
Se identificaron tres especies del género Penicillum, Penicillum citrium y Penicillum rubens. Son destacados productores de enzimas xilanolíticas, como Penicillium chrysogenum, capaz de metabolizar xilasa que es una enzima que degrada polisacáridos de material celulolíticos y lignocelulósico para producir etanol y bioetanol de segunda generación, también para el tratamiento de jugos, cervezas y vinos, y para producir xilitol (Cabral et al., 2018; (Delgado 2021); tiene la capacidad de remediar contaminación con dihidroxibencenos, sulfonatos lineales de alquilbenceno y desperdicios de grasas que son compuestos que actúan como contaminantes de fuentes hídricas ((Franco et al., 2020). Penicillum chrysogenum es un generoso productor de penicilina y derivados con potencial terapéutico y de control biológico de hongos, bacterias y virus (Huber et al., 2020), mientras que Penicillium citrinum ha demostrado tener capacidad lipasa, xilanasa y celulasa y de producir quitosano, pencitrin y pencitrinol (Ouephanit et al., 2019; Lima et al., 2019; Tejas et al., 2019; Lodha et al., 2020; Huber et al., 2020).
La primera sustancia antibiótica descubierta fue la Penicilina, por Alexander Fleming, quien observó que el hongo del pan inhibía el crecimiento de las bacterias de S. aureus que había sembrado, llamando al compuesto Penicilina (Penicillium notatum), pero como el efecto inhibitorio era mínimo llegó a pensar que esa sustancia carecía de interés comercial. Se propuso purificar Penicilina de una antibiótica 5-15 cepa que era mejor productora, dando comienzo a la era antibiótica (Asier, 2021).
Aspergillus fumigatus es un reconocido patógeno oportunista, causante de queratitis, reacciones alérgicas en las vías respiratorias y aspergilosis pulmonar (Nayak et al., 2018). También, tiene aplicaciones biotecnológicas por su capacidad de producir lovastatina que es un metabolito secundario fúngico frecuentemente utilizado con fines farmacéuticos para reducir los niveles de colesterol y eventos cardiovasculares, puede producir enzimas con características celulasas, β-xilosidasa, FPasa, xilanasa, peptidasa y amilasa que tienen aplicación en diferentes sectores productivos como la industria de producción de bioetanol (Xianchun et al., 2020; Mondal et al., 2020). Las celulasas son un conjunto de enzimas hidrolíticas que descomponen la celulosa, mediante la actividad sinérgica de tres diferentes componentes, las endoglucanasa, exoglucanasa y la ß - glucosidasa. A lo largo de los años, estudios de celulasas han demostrado ser potencial biotecnológico en diferentes áreas, aplicada principalmente por ser biodegradables e inofensivas con el medio ambiente. Su producción es bastante estudiada en residuos agrícolas o agroindustriales. La creciente industrialización de estas áreas, ha llevado a la producción de altas cantidades de residuos que no son tratados adecuadamente, siendo importante el aislamiento de microorganismos con potenciales celulolíticos, donde Fusarium sp. CCLM CX presentó la mayor actividad enzimática (Rodríguez, 2019).
Además, se han realizado otros trabajos para evaluar organismos antagonistas para el control de Sclerotinia sp. causante de la podredunbre blanca en lechuga, como, por ejemplo: Trichoderma sp., Aspergillus flavipes, Muscodor yucatanensis, Penicillium commune, Bacillus sp., Clonostachys sp., Paecilomyces lilacinus, Pseudomonas sp. y Coniothyrium sp. (Zubieta et al., 2021).
Baeuveria bassiana es un entomopatógeno que controla plagas de insectos como Nilaparvata lugen, Lasioderma serricorne, Leptinotarsa decemlineata, Plutella xylostella, Phenarium purpurascens, Kuschelorhynchus macadamiae y Carya illinoinensis que atacan los cultivos de arroz, patata, repollo, maíz, macadamia y nuez (Romero et al., 2020; Agboyi et al., 2020; Khun et al., 2020 ).
CONCLUSIONES
Se lograron identificar hongos pertenecientes al género y especies Trichoderma yunnanense, Geotrichum silvícola, Purpureocillium lilacinum, Laburnicola hawksworthii, Penicillium chrysogenum, Aspergillus fumigatus, Penicillium citrinum, Penicillium rubens, Beauveria bassiana, Bipolaris sivanesaniana, Curvularia pisi, y Alternaria destruens. Se mostró una relación taxonómica entre grupos como Trichoderma y Purpureocillum, entre Penicillum, y Alternaria - Curvularia, indicando que son géneros filogenéticamente próximos.
La caracterización molecular y la recopilación del potencial biotecnológico para hongos filamentosos permitirá avanzar en proyectos enfocados a la bioprospección, conocer cualidades y mecanismos de estos microorganismos con miras al desarrollo de procesos y productos biotecnológicos, comprender mecanismos de patogénesis y desarrollar alternativas de control de patógenos principalmente para cultivos de interés agrícola y comercial.














