Introducción
Acinetobacter baumannii es un patógeno hospitalario, relacionado ampliamente con infecciones asociadas a la atención en salud (IAAS) 1, que afecta con predilección a pacientes críticamente enfermos 1-3, de muy difícil control por su capacidad para sobrevivir en condiciones ambientales extremas y posibilidad de transmisión cruzada y aérea 2-4. Adicionalmente A. baumannii se ha diseminado rápidamente en el ambiente hospitalario por su capacidad de adquirir resistencia a múltiples antibióticos, en particular a los carbapenémicos, cuya resistencia es mediada por carbapenemasas, principalmente de clase D (oxacilinasas) y, con menos frecuencia, de clase B o metalobetalactamasas (MBL) 2-3, así como a desinfectantes en relación con bombas de expulsión tipo amv A5. El riesgo de mortalidad entre los pacientes que se infectan por este agente 1-2es muy alto: en un metaanálisis reciente se describe un 33%, con un rango de 8 a 40% y riesgo incrementado de mortalidad de 2,49 de los pacientes infectados por A. baumannii resistentes a carbapenémicos 1 (ABRC).
En Bogotá, la resistencia a A. baumannii en Unidades de Cuidado Intensivo (UCI) fue ≥50% para imipenem y >55% para meropenem, entre los años 2005 y 2007 6. A. baumannii es epidémico y puede pasar a ser endémico 7; el mayor riesgo se observa en las UCI, donde la mortalidad atribuible está entre el 20 y el 65%8. En un estudio de Colombia, se observó que la resistencia en esta población se asoció con incremento de un 60% en los costos de hospitalización9. En Latinoamérica, la información es escasa; por ello, es difícil ponderar el impacto de diferentes factores de riesgo en el desarrollo de resistencia. El propósito de este estudio es describir los factores de riesgo asociados a infección o colonización por ABRC, en adultos hospitalizados en UCI de 13 instituciones de Bogotá.
Materiales y métodos
Tipo de estudio y población
Estudio multicéntrico y prospectivo de casos y controles, en pacientes adultos hospitalizados en UCI de 13 instituciones de Bogotá (H1 a H13, nombradas aleatoriamente) y protocolo con previa aprobación del Comité de Ética. El criterio de inclusión fue que los pacientes tuvieran más de 18 años y que estuvieran hospitalizados en UCI con cultivo de AB. Como el objetivo fue establecer los factores de riesgo para infección o colonización por ABRC, solo se trabajó con pacientes diagnosticados con infección o colonización con aislamiento de este. Para confirmarlo, se realizó una prueba de referencia. Se incluyó a los pacientes que, con el primer aislamiento, presentaran A. baumannii sensible a carbapenémicos (ABSC). Se excluyó a pacientes con aislamiento concomitante de microorganismos diferentes.
El estudio se realizó entre abril de 2005 y el mismo mes de 2006. No fueron incluidos pacientes con aislamientos de ABRC o ABSR identificados antes del inicio del estudio. El emparejamiento se realizó a partir de pacientes hospitalizados en UCI por más de 48 h y hasta 7 días después de su traslado al hogar, al momento de la toma de la muestra con la que se identificó un aislamiento de AB. Se diseñó con error alfa inferior a 5% y beta inferior a 20%, hipótesis de no asociación de factores de riesgo para aislamientos de AB, diferencia de exposición entre casos y controles de 20% y pérdidas de 10%, con relación caso-control 1:1. En total, se tuvieron en cuenta 134 casos10.
Recolección y manejo de la información
Variables analizadas: género, edad, estancia hospitalaria, diagnósticos de ingreso a UCI, origen de la muestra, foco de infección, presencia y duración de dispositivos invasivos, enfermedad quirúrgica, antibióticos previos al cultivo, duración de tratamiento y dosis, puntaje APACHE II al ingreso a UCI, gravedad de sepsis -según criterios de Bone 11- y presencia de enfermedades médicas asociadas. Se conformó un comité de expertos encargados de aplicar a cada caso criterios de IAAS según CDC11 y de definir el aislamiento como infectante o colonizante, y si el inicio antimicrobiano empírico fue apropiado, según diagnóstico infeccioso y perfil de sensibilidad.
Caracterización fenotípica
Se analizó un aislamiento por paciente. La identificación se realizó con API20NE (bioMérieux). En los aislamientos de ABRC, se evaluó sensibilidad antimicrobiana por concentración mínima inhibitoria (CMI) a través de E-Test (AB biodisk) para aztreonam, amikacina, ceftazidima, ceftriaxona, cefepima, ciprofloxacina, imipenem, meropenem, piperacilina-tazobactam, ampicilina-sulbactam, cefoperazona-sulbactam y colistina. El procedimiento y la interpretación de resultados se realizó de acuerdo con las instrucciones del fabricante. La presencia de carbapenemasas MBL se determinó por E-test usando la tira imipenem/imipenem + EDTA (IP/IPI). Los aislamientos con resultado de CMI IP/IPI ≥8 fueron considerados positivos. La multirresistencia se definió como resistencia a 3 o más clases de antibióticos.
Detección de genes codificantes de carbapenemasas
El ADN de los aislamientos se obtuvo por lisis celular a partir de colonias bacterianas frescas de un cultivo en agar BHI, suspendidas en 100 uL de agua destilada estéril y llevadas a calentamiento hasta ebullición por 10 min. La detección de genes codificantes de carbapenemasas se realizó a través de reacción en cadena de la polimerasa (PCR). Para genes codificantes de carbapenemasas oxacilinasas se usó un ensayo de PCR múltiple convencional para blaOXA-23, blaOXA-24, blaOXA-51 y blaOXA-58 12 (tabla 1) y para carbapenemasas MBL se usó PCR simple convencional para los genes blaVIM y blaIMP 13 (tabla 1). Se utilizaron mezclas de reacción que contenían: solución tampón de PCR 1X, 3 mM de MgSO4 (PCR múltiplex de OXAs) y 2 mM de MgSO4 (PCR simple), 0,2 mM de dNTPs, 0,5 μM de cada iniciador excepto para blaIMP para el cual se utilizó 1 μM, y 1 U/μL de Taq AccuPrime (Invitrogen(r)) para la PCR múltiplex y 0,5 U/μL de Taq para la PCR simple. Las condiciones de amplificación usadas se estandarizaron en el laboratorio y fueron desnaturalización inicial de 95 °C por 5 min, 35 ciclos de 95 °C por 45 s, anillamiento de 53 °C para blaIMP y 56 °C para blaVIM y la PCR múltiplex de OXAs por un minuto, 72 °C por un minuto y una extensión final a 72 °C por 7 min. Las reacciones de PCR se realizaron en el termociclador iCycler(r) de Bio-Rad. Los productos de amplificación se visualizaron en geles de agarosa al 1,2%, teñidos con bromuro de etidio (0,5 μg/mL).
Secuenciación de ADN
Los productos de amplificación obtenidos fueron purificados con el estuche comercial PureLink PCR (Invitrogen(r)) y enviados a secuenciación en ambas cadenas con los iniciadores descritos (tabla 1). Las amplímeros fueron secuenciados en el equipo MegaBace DNA Analysis System (Amersham(r)) (servicio de secuenciación BioMol Ltda). Las secuencias de nucleótidos obtenidas se editaron con el software Molecular Evolutionary Genetics Analysis (MEGA version 4.0) y la identidad de las secuencias se evaluó comparándolas con las secuencias de ADN almacenadas en las bases de datos del GenBank disponibles en el sitio web BLASTN (https://blast.ncbi.nlm.nih.gov/Blast.cgi).
Electroforesis en gel de campo pulsado
La tipificación se realizó con electroforesis en gel de campo pulsado (EGCP), el ADN fue digerido con la enzima de restricción ApaI (Promega(r)) y separado en el equipo CHEFDRIII (BioRad(r)), usando las condiciones previamente descritas por Seifert et al. 14. Los patrones electroforéticos se analizaron con el software Fingerprinting II (Bio-Rad(r)). El coeficiente de DICE se usó para calcular las similitudes de los patrones, y para la construcción del dendrograma se usó el algoritmo Unweight Pair-Group Meted with Arithmetic Averages (UPGMA), con una tolerancia de 1,5% y una optimización de 1%. Los aislamientos con similitud ≥85% fueron considerados del mismo genotipo (relacionados clonalmente).
Análisis estadístico
Se realizó estadística descriptiva, análisis bivariado de diferencia de proporciones (X2), prueba de Fisher y rangos logarítmicos, según la necesidad. Se hizo un análisis multivariado de regresión logística. Para el modelo de regresión, se estableció como variable de desenlace la resistencia a carbapenémicos, definida según resultado de E-test a imipenem y meropenem. Las variables de predicción serán incluidas a partir de los resultados del análisis bivariado, con probabilidad de azar inferior a 20% (según recomendación de Kleinbaun).
Para controlar la confusión, se incluyeron dentro del modelo variables sin significación estadística, que, sin embargo, han sido reconocidas como fuente de confusión en diferentes publicaciones. El resultado final del análisis de regresión logística fue validado por 2 métodos diferentes: el de residuos de Shoenfeld y la prueba de bondad de ajuste. Se usó el programa STATA 8.0 (licenciado a la Universidad Nacional de Colombia).
Resultados
Incidencia de infección o colonización
El 1,47% de los pacientes en UCI se infectaron por AB. Un total de 115 correspondieron a IAAS, 2 a infección extrainstitucional y 48 a colonización, distribuidos así: 34 de 153 resultaron ser resistentes y 14 de 30, sensibles. Una institución presentó la mayor frecuencia de aislamientos, con 16%, y otra no presentó ningún aislamiento. La resistencia fue superior al 60% en la mayoría de las instituciones; una tercera institución no presentó resistencia en sus aislamientos (tabla 1). Se concluyó, a partir del seguimiento al AB, que este se presenta en brotes temporales y recurrentes.
Se identificaron desde 3 aislamientos, en una institución, hasta 52, en otra, con reconocimiento de 3 brotes y manifestaciones de endemicidad. Cerca del 75% de los pacientes habían estado hospitalizados 90 días antes. Las principales causas de ingreso a UCI fueron el compromiso del sistema nervioso central (17%) y enfermedades abdominales (15%). Un total de 41 pacientes presentaron enfermedades concomitantes, como diabetes mellitus (12%) y enfermedad pulmonar crónica (11%). Además, el 90% tuvo ventilación mecánica.
Los antibióticos más utilizados previamente en UCI fueron: cefepima 400 DDD, piperacilina-tazobactam 299 DDD, meropenem 227 DDD, ceftriaxona 217 DDD e imipenem 178 DDD. La estancia hospitalaria fue 35 días (IC 95%: 31-40) y los pacientes estuvieron hospitalizados en UCI cerca de 9 días (IC 95%: 7,3-11). Los aislamientos fueron recuperados principalmente de sangre (22,1%; n = 37) y tracto respiratorio (18,5%; n = 31) (tabla 2). La mediana de edad fue 51 años (IC 95%; 47-58), con APACHE entre 15 y 16; 131 pacientes estuvieron previamente hospitalizados en otros servicios antes del aislamiento de AB.
El uso de antibióticos en las diferentes instituciones no se sigue comúnmente por servicios. Se encontró una alta frecuencia de uso de carbapenémicos en las instituciones 7, 8 y 9; el más usado es meropenem. De los antibióticos evaluados, el más utilizado es cefepime, seguido de piperacilina, tazobactam y ceftriaxona (4a).
En el análisis bivariado inicial, los factores asociados con identificación de ABRC fueron: catéter venoso central (CVC) y días con CVC, presencia de sonda vesical (SV) y días con SV, días desde la primera cirugía, estancia hospitalaria previa, hospitalización previa, ingreso por cirugía general, exposición previa a carbapenémicos y cefalosporinas de tercera generación, por más de 3 días, e identificación del genotipo Ab22 (Tabla 3 and Tabla 4). Las variables que permanecieron en el análisis de regresión logística fueron: exposición a carbapenémicos por más de 3 días, OR de 10,4 (IC 95%: 1,7-62) hospitalización previa, OR de 7,99 (IC 95%: 1,3-47,9), tiempo de ventilación mecánica en días, OR de 16,4 (IC 95%: 1,9-138), aislamiento de Ab22, OR 449 (IC 95%: 25-798), e IAAS, OR de 14,12 (IC 95%: 2,4-82,2) (tabla 5).
Tabla 3 Variables demográficas de tipo categórico de pacientes con infección o colonización por A. baumanniiresistente a carbapenémicos en 13 UCI de Bogotá (Colombia), entre abril de 2005 y abril de 2006

Tabla 4 Variables demográficas de tipo continuo de pacientes con infección o colonización por A. baumanniiresistente a carbapenémicos en 13 UCI de Bogotá (Colombia), entre abril de 2005 y abril de 2006

Tabla 5 Resultado de análisis multivariado para factores de riesgo de desarrollar resistencia asociados a infección o colonización por A. baumannii resistente a carbapenémicos en 13 UCI de Bogotá (Colombia), entre abril de 2005 y abril de 2006

El 43% de los pacientes no recibieron tratamiento antibiótico adecuado. El 78% tuvieron cirugías, el 43% abdominales. La mortalidad atribuible a infección por AB fue mayor cuando la terapia inicial no fue apropiada (datos no mostrados).
Caracterización fenotípica
Se evaluó la susceptibilidad de los 165 aislamientos sin realizar diferencia entre los causantes de colonización o infección, 30 fueron ABSC y 135 ABRC. De estos últimos, 83,7% (n = 113) presentaron perfil de multirresistencia (tabla 6). El 35,8% de los aislamientos fueron identificados (por los sistemas automatizados MicroScan, MicroScan WalkAway y VITEK 2) como resistentes o con sensibilidad intermedia a imipenem o meropenem. Entre los aislamientos identificados como resistentes, con el sistema MicroScan WalkAway, se encontró que la concordancia en la identificación de la resistencia con el E-test fue: 100% con imipenem; 82,75% para meropenem; 76,6% para ciprofloxacina; 76,19% para piperacilina tazobactam; 65,71% para piperacilina tazobactam; 55,5% para aztreonam y 36,36% para amikacina.
Se analizaron 15 aislamientos con el sistema MicroScan. La concordancia en la identificación de aislamientos resistentes con el sistema manual E-test fue: 100% ciprofloxacina; 100% piperacilina tazobactam; 84,6% imipenem; 70% meropenem; 50% cefepime; 50% amikacina y 40% aztreonam. Un total de 19 aislamientos fueron analizados con el sistema VITEK 2.
La concordancia en la identificación de aislamientos resistentes con el método E-test fue: 100% aztreonam; 93,75% imipenem; 88,89% meropenem; 88,89% ciprofloxacina; 81,81% piperacilina tazobactam; 60% cefepima y 0% amikacina. De todos los aislamientos con resistencia a los carbapenémicos, el E-test confirmó dicho perfil en 88,7%. Los otros fueron, en realidad, sensibles a carbapenémicos. La resistencia a otros antibióticos fue para amikacina 9,85%; aztreonam 30,9%; piperacilina tazobactam 40,8%; ciprofloxacina 60,56% y cefepime 81,7%. El cálculo de las CIM50 y CIM90 se realizó con las cepas tratadas mediante la prueba del E-test. La mayor sensibilidad fue a colistina (99%, n = 134), excepto un aislamiento (CIM 16 μg/mL) y amikacina (23%; n = 31) (tabla 7) y 31 aislamientos fueron positivos para MBL.
Tabla 7 Distribución de los aislamientos del clon Ab22 en las instituciones a través del tiempo del estudio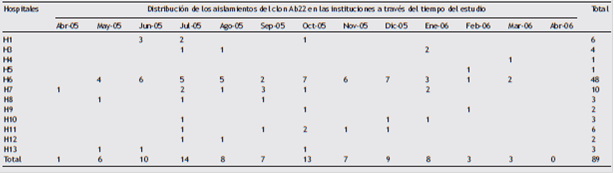
Detección de genes codificantes de carbapenemasas y secuenciación de ADN
Todos los aislamientos fueron positivos para OXA-51 (alelos OXA-66, OXA-69, OXA-95 y OXA-99). De los 135 aislamientos ABRC, en 129 se confirmó OXA-23, 5 fueron positivos únicamente para OXA-51 y uno amplificó OXA-24 (de modo que se confirmó OXA-72). Ningún aislamiento amplificó OXA-58, IMP ni VIM.
Electroforesis en gel de campo pulsado
Por EGCP se definieron 46 genotipos: 12 clones (aislamientos genéticamente relacionados) y 34 perfiles únicos (aislamientos no relacionados genéticamente con el resto) (tabla 1). El genotipo más grande fue Ab22 (n = 89, aislamientos de ABRC-OXA-23) presentes en 12 instituciones, excepto H2 (tabla 1). El primer aislamiento de Ab22 se hizo en abril de 2005 en H7, y para mayo se realizó en H6, H8 y H13 tabla 5; fig 1 .

Figura 1. Distribución de los aislamientos del clon Ab22 en las instituciones a través del tiempo del estudio.
Los aislamientos Ab22 se detectaron en 11 de los 12 meses de estudio (tabla 5). El segundo genotipo más grande fue Ab12, presente únicamente en H4 tabla 1 .
Discusión
A. baumannii es un agente potencialmente fatal de difícil tratamiento y erradicación en el entorno hospitalario. En este estudio, los factores de riesgo para infección/colonización por ABRC fueron: infección cruzada, hospitalización previa, clon (Ab22) y exposición previa a carbapenémicos por más de 3 días. La duración de la terapia antibiótica ha sido asociada previamente con el desarrollo de resistencia por Marra et al. 15, quienes evidenciaron reducción en la frecuencia de aislamientos de AB resistente a imipenem, de 2,7 a 0,2 durante 1.000 días por paciente, al disminuir a 14 días la duración de esquemas de tratamiento antibiótico en UCI. Falagas et al. 16 describieron como principal factor de riesgo el uso previo de antibióticos, principalmente carbapenémicos y cefalosporinas de tercera generación, seguido de fluoroquinolonas, aminoglucósidos y metronidazol.
Aunque en principio la exposición a carbapenémicos y cefalosporinas de tercera generación estuvo asociada en el análisis bivariado, en el multivariado la exposición previa a cefalosporinas de tercera generación no se mantuvo como factor de riesgo. Este resultado puede estar relacionado con el bajo poder obtenido, al encontrar una menor frecuencia de aislamientos a la esperada.
La hospitalización previa expone a los pacientes a uso de antibióticos y riesgo de colonización por gérmenes multirresistentes. Este fenómeno ha sido descrito recientemente en estudios de casos y controles en busca de factores de riesgo para resistencia en A. baumannii17,18. La identificación de un agrupamiento en común, definido como Ab22, favorece igualmente la teoría de la infección cruzada a partir de un foco de difícil erradicación y contaminación de superficies por una bacteria con alta capacidad de supervivencia en condiciones ambientales extremas. Por la metodología de este trabajo, no es posible determinar con seguridad los mecanismos de diseminación entre las diferentes instituciones, pero es posible que estos resultados puedan ser explicados por hallazgos de otros estudios de brote, con los que se identificó colonización de equipos, medio ambiente y manos de trabajadores de la salud como principal factor de riesgo 17,18.
Se encontró que AB es resistente a carbapenémicos en más del 60% de la mayoría de las instituciones. La diseminación de AB a nivel mundial se asocia con los clones internacionales europeos I, II, III y el panamericano (WW5); este último incluye cepas colombianas19. Estos clones altamente exitosos se propagan fácilmente por infección cruzada. Al parecer, el proceso se inicia con presión antibiótica o selección desde un ancestro común 7,20. En este estudio, el agrupamiento fue identificado principalmente en un hospital, el H7, donde tuvo un comportamiento endémico; esto indica que la diseminación en las UCI de Bogotá se asoció posiblemente a transmisión cruzada.
Al considerarse diseminación por infección cruzada, se propone registro previo al traslado de portadores de microorganismos de importancia epidemiológica 21,22. Las medidas estrictas y tempranas de aislamiento fueron claves para contener la infección; en contraste, la reacción tardía hizo imposible erradicar el clon Ab22 en un hospital, con incidencia del 16%. Corbella y Van den Broek 23,24proponen que la contención temprana de brotes es fundamental para evitar la perpetuación endémica y el riesgo de que, una vez consolidado el brote, sea difícil identificar la fuente y controlarlo 25,26. En este estudio se presentó otro agrupamiento de 13 cepas Ab12 restringido a un solo hospital. Su patrón de presentación indica pequeños focos de exacerbación de una cepa endémica de difícil control en este centro y su persistencia en el medio ambiente.
Más del 80% de los aislamientos fueron multirresistentes, con alta sensibilidad a polimixina, considerada una buena opción de tratamiento para ABRC2. Por otra parte, se considera que la tigeciclina también es una opción de tratamiento en infecciones que no incluyan el torrente sanguíneo y tracto urinario. Asimismo, la combinación terapéutica más eficaz es carbapeneme con polimixina intravenosa y micronebulizada, en algunos casos de neumonía nosocomial 27,28. Sulbactam tiene actividad intrínseca frente a AB; su combinación a altas dosis con colistina tiene respuesta del 60% en neumonía27,28.
La cuarta parte de ABRC fueron positivos para MBL con E-test; sin embargo, la PCR mostró resultados negativos para VIM e IMP. Estos falsos positivos con E-test, que han sido previamente descritos29, recomiendan confirmar molecularmente estos resultados fenotípicos o utilizar pruebas con otros inhibidores de MBL, como el EDTA/mercapto acetato de sodio30.
En los ABRC del estudio, OXA-23 fue la enzima prevalente, ratificando lo descrito previamente a nivel mundial y en Colombia 31-33. De acuerdo con la literatura, la diseminación de OXA-23 puede asociarse con la propagación de clones o la transferencia horizontal de esta carbapenemasa. Dicho evento se ha asociado con la presencia de la secuencia de inserción ISAba1, la cual puede localizarse en diferentes transposones como Tn200631,34. En Colombia estudios previos han detectado la presencia de ISAba1 32,33 asociada con OXA-23 y han identificado esta carbapenemasa tanto en cromosoma como en plasmido32. Aunque en nuestro estudio no determinamos la localización de OXA-23, podemos señalar que su diseminación se asoció con la propagación de un clon exitoso (clon Ab22) y en aislamientos no relacionados genéticamente, posiblemente con transferencia horizontal de OXA-23. Adicionalmente en esta investigación se identificó por primera vez OXA-72 35; en un aislamiento de A. baumannii en Colombia, el cual presentó un patrón de campo pulsado único.
La mayor prevalencia de infecciones correspondió al torrente sanguíneo, contrario a lo reportado por Baran et al.36, quienes identificaron que el foco más frecuente es la infección del sitio operatorio y, en segundo lugar, el torrente sanguíneo. Este hallazgo alerta sobre la importancia de prevenir la infección asociada a CVC.
Conclusión
Se puede decir que la optimización de medidas de control de infecciones -en algunas instituciones participantes- se tradujo en una menor frecuencia de aislamientos de AB, hecho que impidió alcanzar el tamaño de muestra calculado. También dificultó identificar otros factores asociados. En contraste, permitió deducir que las medidas para prevenir infección cruzada (como higiene de manos, agrupamiento de pacientes, protocolos de limpieza y desinfección y uso racional de antibióticos) pueden controlar y hasta erradicar episodios de infección por ABRC, siempre y cuando se realice una intervención temprana1 . Con los hallazgos descritos, se resalta la importancia de aplicar medidas efectivas para prevenir infecciones asociadas a la atención en salud especialmente la bacteriemia asociada a dispositivo y de usar prudentemente los antimicrobianos, con el fin de prevenir la selección y propagación de microorganismos resistentes a los antimicrobianos.
Responsabilidades éticas
Protección de personas y animales
Los autores declaran que los procedimientos seguidos se conformaron a las normas éticas del comité de experimentación humana responsable y de acuerdo con la Asociación Médica Mundial y la Declaración de Helsinki.
Confidencialidad de los datos
Los autores declaran que han seguido los protocolos de su centro de trabajo sobre la publicación de datos de pacientes.
Derecho a la privacidad y consentimiento informado
Los autores han obtenido el consentimiento informado de los pacientes o sujetos referidos en el artículo. Este documento obra en poder del autor de correspondencia.
Financiación El presente proyecto fue financiado por Colciencias CÓDIGO: 11010416355, Contrato 444 y la Universidad Nacional de Colombia código: 20201005279.

















