Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Colombia Médica
On-line version ISSN 1657-9534
Colomb. Med. vol.42 no.1 Cali Jan./Mar. 2011
Polimorfismos de 17 marcadores STR del cromosoma-Y en una muestra poblacional del altiplano cundiboyacense*
Korina María Rojas, MSc1, Martha Roa, MSc2, Ignacio Briceño, MD, PhD3, Carolina Guaneme, Microbiol1, Alberto Gómez, PhD4
* Apoyo recibido del Grupo de Genética, Cuerpo Técnico de Investigación, Fiscalía General de la Nación, Instituto de Genética Humana, Facultad de Medicina, Pontificia Universidad Javeriana, Bogotá, DC, Colombia.
1. Perito, Grupo de Genética, Cuerpo Técnico de Investigación, Fiscalía General de la Nación, Bogotá, DC, Colombia. e-mail: korinamaria@hotmail.com, cguaneme@gmail.com
2. Directora Técnica, Grupo de Genética, Cuerpo Técnico de Investigación, Fiscalía General de la Nación, Bogotá, DC, Colombia. e-mail: marthroa@gmail.com
3. Profesor Titular, Instituto de Genética Humana, Facultad de Medicina, Pontificia Universidad Javeriana, Bogotá, DC, y Profesor Investigador, Facultad de Medicina, Universidad de la Sabana, Chía, Colombia. e-mail: ibriceno@javeriana.edu.co, ignaciobb@sabana.edu.co
4. Profesor Titular, Instituto de Genética Humana, Facultad de Medicina, Pontificia Universidad Javeriana, Bogotá, DC, Colombia. e-mail: agomez@javeriana.edu.co
Recibido para publicación Julio 6, 2010 Aceptado para publicación Noviembre 23, 2010
RESUMEN
Introducción: La tipificación molecular de ADN de cromosoma-Y es una herramienta de reconocida importancia en el proceso de identificación de individuos de género masculino en diversos casos forenses. Actualmente es una herramienta de apoyo para los laboratorios de genética estatales en la identificación de víctimas del conflicto armado en Colombia, dentro de los procesos enmarcados en la Ley de Justicia y Paz. En este estudiose determina el haplotipo del cromosoma-Y que será comparado con parientes por línea paaterna de género masculino, con el fin de realizar el análisis estadístico de estos marcadores, aportar a una base de datos colombiana y comparar con parientes de la línea masculina.
Objetivo: Realizar una caracterización de haplotipos mediante análisis de marcadores moleculares, STR del cromosoma-Y en una muestra de población del altiplano cundiboyacense colombiano.
Metodología: De las muestras del «Programa de Banco de muestras de sangre de funcionarios de la Fiscalía General de la Nación (FGN)» fueron seleccionadas aleatoriamente 74 muestras de individuos que no reportaron relación por línea paterna entre sí y cuyo origen geográfico por línea paterna dos generaciones atrás correspondía a los departamentos de Cundinamarca o Boyacá. Se amplificaron y tipificaron 17 polimorfismos de repeticiones en tandem en el cromosoma-Y, el que se aplicó el kit comercial AmpFiSTR®YFilerTM (DYS456, DYS389I, DYS390, DYS389II, DYS458, DYS19, DYS385a/b, DYS393, DYS391, DYS439, DYS635, DYS392, Y-GATA-H4, DYS437, DYS438 y DYS448).
Resultados: Distribución de frecuencias alélicas y haplotípicas para los 17 marcadores analizados: se determinaron 71 haplotipos con 17 marcadores STR-Y, de los que 68 eran únicos y 3 fueron observados en 2 individuos con un haplotipo compartido para los dos departamentos del altiplano cundiboyacense. La diversidad haplotípica para la muestra poblacional fue de 99.9% con un poder de discriminación de 95.9% y probabilidad de coincidencia al azar de 1.5%. La comparación de las dos poblaciones entre sí no presentó diferencias estadísticas significativas, es decir, no hubo variaciones de las frecuencias alélicas entre Cundinamarca y Boyacá.
Discusión: Los valores de diversidad haplotípica, poder de discriminación y probabilidad de coincidencia al azar corroboran la utilidad del análisis de STR-Y en casos de filiación por línea paterna y son coherentes con los valores observados en el inicio del desarrollo de bases de datos de haplotipos.
Conclusión: Los datos del análisis de los 17 marcadores STR-Y, recogidos en el presente estudio, aportan haplotipos de población del altiplano cundiboyacense, la cual es una de las concentraciones de población más significativas en Colombia. Estos resultados corresponden a una recopilación de datos informativos que permiten mejorar una base de datos en la que se genere la estimación real de frecuencias haplotípicas de STR-Y para su aplicación en la práctica forense y estudios de poblaciones humanas.
Palabras clave: Cromosoma-Y; Haplotipo; STR-Y; Diversidad haplotípica; Colombia.
Colomb Med. 2011; 42: 88-97
Polymorphism of 17 Y-chromosome STR Markers in a sample from the Cundiboyacense Plateau
SUMMARY
Introduction: The application of Y-Chromosome molecular DNA typing is a tool of recognized importance in the process of identification of male individuals in various forensic cases, and currently it is now a support tool for genetic laboratories seeking to identify victims of the armed conflict in Colombia within the legal process of «Justice and Peace». In this report, the Y-chromosome haplotype is determined and statistical analyses are performed to improve databases of Colombian Y-chromosome for comparison with relatives of the male line.
Objective: Characterization of haplotypes through analysis of Y-chromosome STR molecular markers in a sample of Colombian Cundiboyacense highland population.
Methods: From the «Program of blood Bank from officials at the Attorney General's Office (FGN)», 74 samples were randomly selected from individuals who reported no paternal relationship with each other and whose paternal geographic origin belonged at least for two generations to the departments of Cundinamarca and Boyacá. Seventeen (17) tandem repeat polymorphisms in the Y chromosome were amplified and typed (DYS456, DYS389I, DYS390, DYS389II, DYS458, DYS19, DYS385a/b, DYS393, DYS391, DYS439, DYS635, DYS392, Y-GATA-H4, DYS437, DYS438, and DYS448).
Results: Distribution of allele and haplotype frequencies for the 17 markers were analyzed: 71 haplotypes were determined with 17 Y-STR markers of which 68 were unique and 3 were observed in two individuals, with a single shared haplotype for the two departments of the Cundiboyacense Plateau. The haplotype diversity for the sample population was 99.9% with a discrimination power of 95.9% and random match probability of 1.5%. The comparison of the two populations together had no significant differences, and therefore no regional specificity was found in allele frequencies between Cundinamarca and Boyacá.
Discussion: The values of haplotype diversity, discrimination power, and probability of random coincidence showed the usefulness of Y-STR analysis in cases of patrilineal descent and are consistent with values observed in the early development of haplotype databases.
Conclusion: The data analysis of the 17-Y STR markers obtained in this study provide haplotypes for the Cundiboyacense highlands, one of the most significant concentrations of population in Colombia, and serves as an informative database for forensic practice and genetic studies for human populations.
Keywords: Y-Chromosome; Haplotype; STR-Y; Haplotype diversity; Colombia.
Colomb Med. 2011; 42: 88-97
Los marcadores moleculares del cromosoma-Y son importantes en la identificación humana por su diversidad o polimorfismo (gran variabilidad entre individuos), resultante de variaciones en la secuencia del genoma (alelos) en regiones no codificantes, determinadas por el número de veces que se presenta un alelo con respecto al número total de alelos en una población para un locus en particular (frecuencia génica), lo que refleja el grado de heterogeneidad y su poder de discriminación. El cromosoma-Y, por no recombinarse, transmite los polimorfismos en forma ligada de generación en generación en individuos de sexo masculino como un haplotipo (combinación de estados alélicos de secuencias polimórficas localizadas en el cromosoma-Y humano), y traza la evolución del linaje paterno1,2, además refleja las migraciones humanas y permite el uso en identificación humana.
En procesos de identificación por ADN, se comparan los perfiles genéticos de restos esqueléticos exhumados en varias poblaciones de Colombia con los perfiles genéticos obtenidos de muestras de referencia de familiares de las personas reportadas como desaparecidas, tipificadas con marcadores STR autosómicos de rutina (AmpFiSTR® IdentifilerTM de Applied Biosystems y PowerPlex16® System de Promega) o marcadores STR autosómicos utilizados para muestras en mal estado de conservación o con presencia de inhibidores (PowerPlex16HS® System de Promega y AmpFiSTR®MinifilerTM de Applied Biosystems). Para estos casos, las muestras de referencia de familiares en primer grado de consanguinidad (hijos y progenitores) son las más indicadas para dar una probabilidad informativa mayor a 99.9%. Sin embargo, no siempre se cuenta con familiares ascendientes y descendientes directos de los desaparecidos sino que esta comparación se hace con otros familiares, quienes pueden ser hermanos paternos, abuelos paternos u otros parientes, evaluándose un vínculo de parentesco patrilineal donde se obtiene un haplotipo con marcadores STR-Y del estuche comercial AmpFiSTR® YfilerTM que puede complementar el índice de parestesco y probabilidad que en algunos casos, es menor a 99.9%. También es una herramienta útil en desastres masivos en los que no se pueden individualizar los restos mortales, ni determinar la cuarteta básica (sexo, edad, estatura y raza) como tamizaje y correlación con los grupos familiares para seguir con el proceso de identificación. Igualmente, en paternidad civil de hijos varones, con ausencia del padre biológico, el haplotipo debe coincidir con el haplotipo de parientes masculinos del presunto padre, determinándose el linaje paterno del grupo familiar. Adicionalmente es útil en los delitos contra la libertad sexual de los que se obtengan mezclas de fluidos o varios perfiles de ADN de individuos de género masculino, o bien con el fin de esclarecer otro tipo de crímenes3-6.
En estos estudios es fundamental el conocimiento previo de las frecuencias génicas y haplotípicas de los marcadores del cromosoma-Y de las poblaciones representativas o las regiones que las engloban al momento de utilizarlas, para fortalecer la base de datos de referencia nacional que permita la asignación de valores de probabilidad, útiles en identificación humana7.
Por esta razón se presentan en este estudio los haplo-tipos y estimaciones de las frecuencias poblacionales resultado del análisis de 17 marcadores STRs del cromosoma-Y (DYS456, DYS389I, DYS390, DYS389II, DYS458, DYS19, DYS385a/b, DYS393, DYS391, DYS439, DYS635, DYS392, Y-GATA-H4, DYS437, DYS438 y DYS448) de individuos de poblaciones del altiplano cundiboyacense, con el fin de fundamentar este sistema de filiación por línea paterna para ser utilizado en una gran base de datos de referencia nacional que recopile frecuencias génicas y haplotípicas de los análisis poblacionales estudiados por los diferentes equipos de investigadores proponiendo una descripción de referencia en identificación humana de utilidad en procesos judiciales llevados por la Unidad Nacional de Justicia y Paz, por la Unidad Nacional de Derechos Humanos, o bien en casos criminales de procesos penales investigados por la Fiscalía General de la Nación.
De manera paralela, se relacionan otros hallazgos no genéticos con los haplotipos frecuentes con respecto a los apellidos paternos, para intentar trazar el recorrido patrilineal de poblaciones masculinas que migraron de Europa, principalmente de España, y que se establecieron en el período de la Conquista en regiones de nuestro país y en especial en el altiplano cundiboyacense, ubicado en la cordillera oriental de los Andes ecuatoriales de Colombia y forma el conjunto de altiplanos más grande y diverso del país. Además, comprende de sur a norte la Sabana de Bogotá, el Valle de Ubaté-Chiquinquirá, el altiplano de Samacá-Villa de Leyva y el de Tunja-Sogamoso-Lago de Tota. Estas regiones se han agrupado debido a su cercanía y a que comparten un mismo origen geológico y algunos procesos ambientales y ecológicos comunes.
En el curso del diagnóstico realizado por grupos de trabajo, direccionados por el centro de extensión académica de la Universidad Nacional sobre la resolución de la problemática de vivienda en Bogotá, sugiere que está ligado con la ubicación de la población y la dinámica de migración. Por ello, estudiaron componentes poblacionales, con los diferente censos del país que determinaron que la conformación de la Región Central (departamentos de Boyacá, Cundinamarca, Meta, Tolima y sus ciudades capitales), ha tenido intensos procesos de migración dirigidos especialmente de Cundinamarca, Boyacá y Tolima hacia el principal núcleo poblacional: Bogotá, observados desde el siglo XIX hasta el Censo de 1993. A partir de los datos preliminares del Censo 2005 y de estudios analizados en la última década, parecería que cambia la tendencia y Cundinamarca se convierte en el primer departamento receptor de bogotanos con el 61.4% que migran a Cundinamarca (DANE, 2006) y así, emigrantes de la región central, no llegan a Bogotá sino que se ubican en municipios limítrofes. El 38% de la población que llega a Bogotá viene procedente de Cundinamarca, Tolima y Boyacá. Los cuatro departamentos (incluyendo Bogotá), han mantenido el peso porcentual en alrededor del 28% a lo largo de 1938 y 2005 en relación con la población del país, lo que cambia drásticamente la forma de distribución de la población dentro del territorio de la Región Central. Por lo anterior, el crecimiento de Bogotá se ha dado principalmente por el decrecimiento de Boyacá, Tolima y Cundinamarca, población que va a la capital tras mejores condiciones de vida y desarrollo.
MATERIALES Y MÉTODOS
Población y muestra. La población sobre la que se realizó el muestreo es la de funcionarios de la Fiscalía General de la Nación (FGN) que acceden de manera voluntaria al «Programa de Banco de muestras de sangre de funcionarios de la Fiscalía General de la Nación» que diligencian un acta de toma de muestra y consentimiento informado en la que se relaciona el origen geográfico por línea paterna y materna del individuo en dos generaciones atrás e indican si laboran otros familiares en la institución.
En la base de datos de 2000 individuos se realizó un muestreo aleatorio simple con personas cuyo origen en dos generaciones atrás por línea paterna correspondieran a los departamentos del altiplano cundiboyacense (Cundinamarca: porcentaje de población 5.1% y Boyacá: 3.1% en relación con el porcentaje de la Región Central, Proyecciones DANE 2005) y no tuvieran relación de parentesco patrilineal. Fueron seleccionadas 74 muestras sanguíneas anticoaguladas de varones que cumplieron con los criterios de inclusión (Cuadro 1), en los que el componente étnico mayoritario es el mestizo, es decir, una mezcla de descendientes de europeos, amerindios y africanos, con predominio de fenotipo caucásico.
Esta investigación fue aprobada por el Comité de Investigaciones y Ética de la Pontificia Universidad Javeriana como consta en el Acta N° 07-2004.
Extracción del ADN. El ADN fue extraído de 300 µl de sangre a través del uso del estuche comercial Wizard® para purificación de ADN y se siguieron los procedimientos de Wizard® Genomic DNA Purification Kit (Instrucciones para el uso de productos A1120, A1123, A1125 y A1620 de Promega Corporation, Madison, USA).
Amplificación del ADN. La amplificación de un estuche comercial multiplex AmpFiSTR® YfilerTM (Manual del usuario del estuche de Applied-Biosystems, USA)8 con 17 marcadores cromosoma-Y (DYS456, DYS389I, DYS390, DYS389II, DYS458, DYS19, DYS385, DYS393, DYS391, DYS439, DYS635, DYS392, Y-GATA H4, DYS437, DYS438 y DYS448) se realizó en un volumen final de 12.5 ml con los siguientes componentes: mezcla de reacción para PCR (AmpFiSTR® Y-Filer MIX: MgCl2, dNTPs, albúmina de suero bovino en buffer con azida de sodio 0.05%) 4.6 ml; Primers forward y reverse (AmpFiSTR® Y-Filer Primer set) 2.5 ul; enzima ADN polimerasa (Amplitaq GoldTM DNA Polimerase 5 U/ml) 0.4 ml; ADN (0.5-1 ng/ml) 1.0 ml y 4 ml de agua destilada desionizada. La reacción en cadena de la polimerasa (PCR) se llevó a cabo en un Termociclador GeneAmp®PCR System 9700.
Tipificación de los polimorfismos del ADN. Se utilizó un Analizador Genético ABI PRISM® 310 (Applied-Biosystems, Foster City, CA)9 para tipificación molecular y se siguieron las instrucciones de la casa comercial. El software Genotyper®, analiza el estándar de tamaño de control interno GeneScan-500 LIZ® en cada muestra y la escalera alélica para asegurar la precisión en cada inyección del capilar. La asignación alélica se realizó por comparación de los fragmentos obtenidos en pares de bases para cada pico del alelo de las muestras analizadas con los picos de la escalera alélica provista por la casa comercial AmpFiSTR® Y-Filer de Applied Biosystems, y se tuvieron en cuenta las recomendaciones del grupo de referencia de la Sociedad Internacional de Genética Forense (ISFG)10.
Análisis estadísticos. La estimación de las frecuencias alélicas y haplotípicas se definió como la proporción en la que se encuentra cada alelo o haplotipo dentro de la población de estudio y la frecuencia de los haplotipos. La diversidad alélica (génica) y haplotípica fue calculada para evaluar la eficacia de los marcadores moleculares usados con respecto a la sumatoria de las frecuencias de cada polimorfismo y haplotipos en la población de estudio de acuerdo con lo reportado previamente por Nei11 y se usó el software Arlequín versión 3.1. La capacidad discriminatoria se determinó con el número de haplotipos observados sobre el total de individuos analizados y la probabilidad de coincidencia al azar se estimó para determinar la probabilidad que dos individuos seleccionados al azar en la población tengan un mismo haplotipo de ADN cromosoma-Y. El valor del Fst calculó la distancia genética correspondiente a 15 loci (el DYS385a/b no fue incluido) entre las poblaciones de este estudio: Cundinamarca, Boyacá y la región del Caribe colombiano (Atlántico, Bolívar, Cesar, Córdoba, Guajira, Magdalena y Sucre)12, con el software Arlequín versión 3.113.
RESULTADOS Y DISCUSIÓN
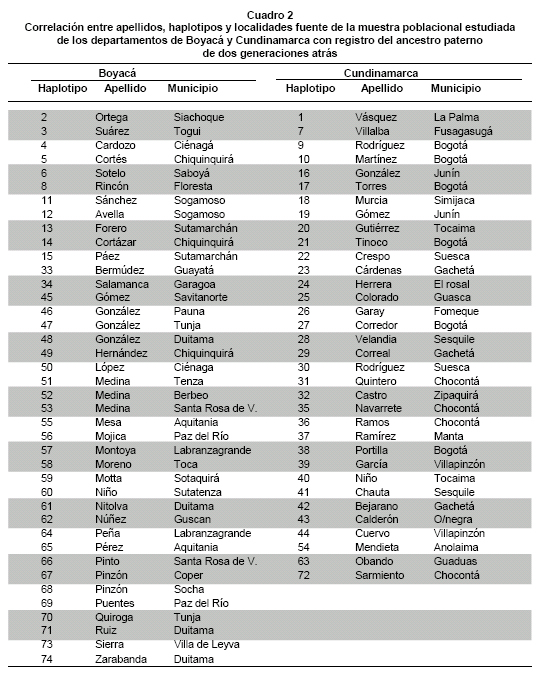
En la muestra poblacional del altiplano cundiboyacense se encontraron 71 haplotipos distintos con 17 loci de AmpFiSTR® Y-Filer (Cuadro 2), diferenciándose 68 haplotipos únicos de individuos que no comparten el mismo linaje paterno y 3 haplotipos compartieron el mismo linaje encontrados en dos individuos cada uno (Cuadro 3).
Se evaluó diversidad haplotípica de 0.9988% en la probabilidad de coincidencia al azar de 1.5% y su capacidad de discriminación de 95.9% para los 17 loci STR cromosoma-Y en relación con la población cundiboyacense. Este resultado arroja parámetros confiables para usar eficazmente este número de marcadores para fines de filiación en casos de identificación humana y análisis forense, datos concordantes con los resultados obtenidos en estudios realizados a poblaciones del Caribe colombiano12 con diversidad haplotípica 0.9995 y a poblaciones del sur occidente colombiano (Cauca, Valle del Cauca y Nariño) con diversidad haplotípica de 0.997714.
Al utilizar el número de combinaciones con los marcadores STR-Y que conforman el sistema genético AmpFiSTR® Y-Filer, la capacidad discriminatoria fue de menor a mayor: a. Haplotipo mínimo europeo de 81.1%, b. Haplotipo extendido de 89.2%, c. Y-Filer de 95.9%, con diversidades haplotípicas para 9 loci STR-Y de 98.7% (haplotipo mínimo), de 99.5% para 11 loci STR-Y (haplotipo extendido) y de 99.9% 17 loci STR-Y empleados. Cuando aumenta la capacidad discriminatoria y la diversidad haplotípica, la probabilidad de coincidencia disminuye, es decir, la probabilidad de que dos individuos seleccionados al azar en una población tengan el mismo haplotipo. Por lo anterior, la implementación de 17 marcadores STR del cromosoma-Y (DYS456, DYS389I, DYS390, DYS389II, DYS458, DYS19, DYS385a/b, DYS393, DYS391, DYS439, DYS635, DYS392, Y-GATA-H4, DYS437, DYS438 y DYS448) se convierte en una herramienta estadística poblacional de referencia en investigaciones criminales (delitos contra la libertad sexual, cuando hay más de dos victimarios en una evidencia biológica) y en la filiación de individuos al grupo patrilineal en casos cerrados como desastres aéreos.
Mediante el análisis de poblaciones pares entre Cundinamarca y Boyacá el valor Fst estimado fue de -0.0026 (software Arlequín versión 3.1) por lo cual se concluye que no se presenta diferenciación genética entre estas poblaciones. Igualmente, analizando cada población con la región Caribe12 se calculó un Fst de 0.0005 en relación con Cundinamarca y 0.003 en relación con Boyacá y se encontró una pequeña diferenciación genética con valores de referencia entre 0-0.05, coherente con otros estudios de marcadores microsatélites autosómicos en donde los valores Fst indican que no existe subestructura entre estas dos poblaciones y que puede clasificarse como un solo grupo en el que las distribuciones de frecuencias no muestran una diferencia significativa15. Esto se corrobora con la comparación realizada de Cundinamarca y Boyacá mediante el análisis molecular de varianza Amova (Cuadro 1) y escalamiento multidimensional en la base de datos YHRD con poblaciones mestizas reportadas de Bogotá, Atlántico, Bolívar, Caldas, Cesar, Guajira, Magdalena, Nariño, Quindío, Valle del Cauca y Colombia, utilizando los 17 marcadores moleculares del cromosoma-Y.
El haplotipo más frecuente compartido entre Cundinamarca y Boyacá se encontró en dos individuos H16 y H59 (DYS 456*15, DYS389I*13, DYS390*23, DYS389II*30, DYS458*18.2, DYS19*14, DYS385a/b*13, 18, DYS393*12, DYS391*11, DYS439*11, DYS635*20, DYS392*11, Y-GATA-H4*11, DYS 437*14, DYS 438*10 y DYS448*20), apellidos González y Motta con cero coincidencias en la base de datos YHRD (27.532 haplotipos distribuidos en 182 poblaciones). El haplotipo más frecuente en Boyacá se encontró en dos individuos H55 y H60 (DYS456*15, DYS389I*14, DYS390*23, DYS389II*31, DYS 458*22.2, DYS 19*14, DYS385a/b*13, 15,20, DYS393*12, DYS 391*11, DYS439*11, DYS635*20, DYS392*12, Y-GATA-H4*11, DYS437*14, DYS 438*10 y DYS 448*20) con duplicación del locus DYS385a/b* (Figura 1), apellidos Mesa y Niño, sin coincidencias en la base de datos YHRD. El haplotipo más frecuente en Cundinamarca se encontró en dos individuos H20 y H35 (DYS456*15, DYS389I*13, DYS390*24, DYS389II*29, DYS458*18, DYS19*14, DYS385a/b*11, 15, DYS393*13, DYS391*11, DYS439*12, DYS635*23, DYS392*13, Y-GATA-H4*12, DYS 437*15, DYS438*12 y DYS448*19) apellidos Gutiérrez y Navarrete, respectivamente, con dos coincidencias de 9470 haplotipos (distribuidos en 69 poblaciones de origen europeo) en Central Portugal Central y Mendoza, Argentina. Esta diferencia entre apellidos sugiere un evento de no paternidad en dos generaciones atrás o bien una transmisión atípica del apellido por vía paterna (Cuadro 2).
En todos los casos de haplotipos compartidos no hubo coincidencia de linaje en cuanto al apellido, lo que se corresponde con lo reportado en un trabajo paralelo en la región suroccidental de Colombia en el que se halló una inconsistencia de linaje cercana al 40% al comparar apellidos y genotipos. La frecuencia haplotípica para cada uno de los 3 haplotipos compartidos corresponde a 0.027014.
El marcador DYS385a/b que presentó la duplicación de 13, 15 y 20 en dos individuos (H55, H60) de la población de Boyacá no relacionados por apellido paterno, fue confirmado con nueva extracción de ADN y procesamiento de las muestras. Este hallazgo podría estar relacionado con la inserción de polimorfismos en la región cromosómica debido a la falta de recombinación16,17, y es importante porque las duplicaciones así como las variantes alélicas son características individualizantes que se deben tener en cuenta para interpretar un resultado forense o en investigaciones de personas desaparecidas relacionadas por línea paterna para ser usadas como muestras de referencia18. Uno de los usos de los marcadores STR-Y en el campo forense es la determinación del número de individuos contribuyentes en una muestra que involucre mezcla de células masculinas y femeninas, tanto como mezclas de células masculinas. Para poder interpretar un resultado en el que el número de victimarios es mayor a 1 y estos no están relacionados, es necesario observar el número y altura de picos por loci presentes en la muestra. La posible existencia de duplicaciones en la población debe ser considerada para no asignar falsamente polimorfismos a los individuos involucrados17.
Conflicto de intereses. Los autores declaran que no hay conflicto de intereses en el presente manuscrito.
REFERENCIAS
1. Pérez-Lezaun A, Calafell F, Seielstad M, Mateu E, Comas D, Bosch E, Bertranpetit J. Population genetics of the Y-chromosome short tandem repeats in humans. J Mol Evol 1997; 45: 265-70. [ Links ]
2. Jobling MA, Tyler-Smith C. Fathers and sons the Y chromosome and human evolution. Trends Genet. 1995; 11: 449-56. [ Links ]
3. Betz A, Bässler G, Dietl G, Steil X, Weyermann G, Pflug W. DYS STR analysis with epithelial cells in rape case. Forensic Sci Int. 2001; 118: 126-30. [ Links ]
4. Gusmão L, Alves C, Amorim A. Molecular characteristics of four human Y specific microsatellites (DYS434, DYS437, DYS438, DYSS439) for population and forensic studies. Ann Hum Genet. 2001; 65: 285-91. [ Links ]
5. Gusmão L, Alves C, Beleza S, Amorim A. Forensic evaluation and population data on the new Y-STRs DYS434,DYS437, DYS438,DYS439 and GATA A10. Int J Legal Med 2002; 116: 139-47. [ Links ]
6. Butler JM, Schoske R, Vallone PM, Kline MC, Redd AJ, Hammer MF. A novel multiplex for simultaneous amplification of 20 Y chromosome STR markers. Forensic Sci Int. 2002; 129: 10-24. [ Links ]
7. Willuweit S, Roewer L. Y-chromosome haplotype reference database (YHRD): Update. Forensic Sci Int Genet. 2007; 1: 83-7. [ Links ]
8. APPLIED BIOSYSTEMS. AmpFlSTR Yfiler product bolletin. Human Identification; 2004. [ Links ]
9. ABI PRISM 310 Genetic Analyzer User´s Manual. Perkin Elmer, 1995; USA. [ Links ]
10. Gusmão L, Butler JM, Carracedo A, Gill P, Kayser M, Mayr WR, et al. DNA Commission of the International Society of Forensic Genetics (ISFG). An update of the recommendations on the use of Y-STRs in forensic analysis. Forensic Sci Int. 2006; 157: 187-97. [ Links ]
11. Nei M. Molecular evolutionary genetics. New York: Columbia University Press; 1987. [ Links ]
12. Romero RE, Briceño I, Del Pilar Lizarazo R, Willuweit S, Roewer L, Gómez A. A Colombian Caribbean population study of 16 Y-chromosome STR loci. Forensic Sci Int Genet. 2008; 2: e5-e8. [ Links ]
13. Schneider S, Roessli D, Excoffier L. Arlequin Version 2000: A software for population genetics data analysis. Genetics and Biometrics Laboratory. Switzerland: University of Geneva. [ Links ]
14. Ávila S, Briceño I, Gómez A. Genetic population analysis of 17 Y-chromosomal STRs in three states (Valle del Cauca, Cauca and Nariño) from Southwestern Colombia. J Forensic Legal Med 2009; 16: 204-11. [ Links ]
15. Rey M, Gutiérrez A, Shroeder B, Usaquén., Carracedo A, Bustos I et al. Allele frecuency for 13 STR's from Colombian populations: Bogotá and Boyacá. Forensic Sci Int. 2003; 136: 83-5. [ Links ]
16. Kayser M, Sajantila A. Mutations at Y-STR loci: implications for paternity testing and forensic analysis. Forensic Sci Int. 2001; 118: 116-21. [ Links ]
17. Butler JM, Decker AE, Kline MC, Vallone PM. Chromosomal duplications along the Y chromosome and their potential impact on Y-STR interpretation. J Forensic Sci. 2005; 50: 853-9. [ Links ]
18. Butler JM. Recent developments in Y-Short tandem repeat and Y single nucleotide polymorphism analysis. Forensic Sci Int. 2003; 15: 91. [ Links ]