Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Colombia Médica
On-line version ISSN 1657-9534
Colomb. Med. vol.43 no.2 Cali Apr. 2012
Análisis de la estructura genética poblacional a partir de polimorfismos de genes asociados con la regulación de la presión arterial en una muestra de Bucaramanga, Colombia
Analysis of population genetic structure from Bucaramanga (Colombia) based on gene polymorphisms associated with regulation of blood pressure
Francisco Javier León1, Fernando Rondón1*, Clara Inés Vargas2, Myriam Oróstegui3, Leonelo Bautista4, Norma Cecilia Serrano5, María c. Páez5*, Adriana Castillo2*.
1Escuela de Medicina, Escuela de Biología, Universidad Industrial de Santander, E-mail: franciskoleon@gmail.com
1*Escuela de Medicina, Escuela de Biología, Universidad Industrial de Santander, E-mail: ferongon@uis.edu.co
2Escuela de Medicina, Laboratorio de Genética, Universidad Industrial de Santander, E-mail: cvargas@uis.edu.co
2*Escuela de Medicina, Laboratorio de Genética, Universidad Industrial de Santander, E-mail: castillo@uis.edu.co
3Escuela de Medicina, Observatorio Epidemiológico de Enfermedades Cardiovasculares, Universidad Industrial de Santander, E-mail: diefsalud@uis.edu.co
4Escuela de Medicina y Salud Pública,Universidad de Wisconsin, E-mail: lebautista@wisc.edu
5Centro de Investigaciones Biomédicas, Universidad Autónoma de Bucaramanga, E-mail: nserrano@ unab.edu.co
5*Centro de Investigaciones Biomédicas, Universidad Autónoma de Bucaramanga,, E-mail: mpaez@unab.edu.co
Received 8 July 2011, Accepted 16 January 2012
ABSTRACT
Introduction: In spite nearly 40% of the variability in blood pressure can be explained by genetic factors, the identification of genes associated to essential high blood pressure is difficult in populations where individuals have different genetic precedents; in these circumstances it is necessary to determinate whether the population is sub-structured because this can bias studies associated with this disease.
Objectives: To determine the genetic structure of the population in Bucaramanga from genetic polymorphisms asso-Received in revised form 25 november 2011 ciated with the regulation of blood pressure: 448G>T, 679C>T y 1711C>T from the gene kinase 4 of the dopaminergic receptor linked to the protein G and Glu298Asp, -786T>C and the VNTR of the intron 4 of the gene of endothelial nitric oxide.
Methodology: A sample of 552 unrelated individuals was studied through analysis of Restriction fragment length polymorphism. The allelic, haplotypic and genotypic frequencies were calculated, the Hardy-Weinberg equilibrium was determined and a molecular analysis of variance was performed to determine the genetic structure.
Results: 38 Haplotypes were identified, with GCCTG4b as the most frequent (21.2%). The most diverse polymorphism was 448G>T with a frequency of 49.9% for heterozygous. The six polymorphisms were found in genetic equilibrium and genetic structure of populations was not evidenced (FST = 0,0038).
Conclusion: The population studied does not present a genetic sub-structure and the polymorphisms analyzed were found in genetic equilibrium, this indicates that the population mixes randomly and there are no sub-groups capable of affecting the results of the association studies.
Key Words: High blood pressure essence, Complex disease, GRK4, eNOS, Polymorphisms, Populations genetics
RESUMEN
Introducción: A pesar que cerca del 40% de la variabilidad en la presión arterial es explicada por factores genéticos, la identificación de genes asociados a la hipertensión arterial esencial es difícil en poblaciones constituidas por individuos con antecedentes genéticos diferentes; en esta circunstancia se debe determinar si la población está sub-estructurada porque esto puede sesgar los estudios de asociación con esta enfermedad.
Objetivo: Determinar la estructura genética de la población de Bucaramanga a partir de polimorfismos genéticos asociados con la regulación de la presión arterial: 448G>T, 679C>T y 1711C>T del gen de la quinasa 4 del receptor dopaminérgico acoplado a proteína G y Glu298Asp, -786T>C y el VNTR del intrón 4 del gen de la sintasa de óxido nítrico endotelial.
Metodología: Se estudió una muestra de 552 individuos no relacionados mediante análisis de polimorfismos de longitud de fragmentos de restricción. Se calcularon las frecuencias alélicas, haplotípicas y genotípicas, se determinó el equilibrio de Hardy-Weinberg y se realizó un análisis molecular de varianza para determinar la estructura genética.
Resultados: Se identificaron 38 haplotipos siendo GCCTG4b el más frecuente (21.2%). El polimorfismo más diverso fue el 448G>T con una frecuencia de heterocigotos del 49.9%. Los seis polimorfismos se encontraron en equilibrio genético y no se evidenció estructura genética poblacional (FST = 0,0038).
Conclusión: La población estudiada no presenta subestructura genética y los polimorfismos analizados se encontraron en equilibrio genético, lo que indica que la población se mezcla aleatoriamente y no existen subgrupos que puedan afectar los resultados de estudios de asociación.
Palabras claves: Hipertensión arterial esencial, Enfermedad compleja, GRK4, eNOS, Polimorfismos, Genética de poblaciones.
INTRODUCCIÓN
Los niveles de presión arterial tienden a agregarse en familias, debido en parte a predisposición genética compartida, de hecho cerca de 40% de la variabilidad en presión arterial es explicado por factores genéticos y el riesgo de desarrollarla después de los 50 años, se duplica por cada familiar de primer grado con historia de hipertensión1. La presión arterial es regulada por múltiples mecanismos que involucran varios genes no-alélicos con efectos aditivos pequeños. Aunque el mecanismo específico alterado no puede ser identificado en cerca del 90% de los casos, las variantes genéticas individuales (alelos) o las combinaciones de alelos (haplotipos) involucrados en la regulación de la presión arterial son los factores genéticos con mayores probabilidades de incrementar el riesgo de desarrollar hipertensión.
Las variantes o polimorfismos genéticos asociadas a la regulación de la excreción urinaria de sodio y a la regulación del tono vasomotor son factores de riesgo potenciales para el desarrollo de hipertensión. Entre los primeros se encuentran los polimorfismos 448G>T o R65L o rs2960306, el 679C>T o A142V o rs1024323 y el 1711C>T o A486V o rs1801058 del gen GRK4 que codifica la quinasa 4 de los receptores acoplados a la proteína G, específicamente los receptores D1 y D2 de la dopamina, que median el efecto natriurético de esta catecolamina a nivel del túbulo contorneado proximal de la nefrona2. Entre los segundos se destacan los polimorfismos 894G>T o Glu298Asp o rs1799983, el -786T>C o rs2070744 y el Intrón 4 del gen eNOS que codifica la sintasa de óxido nítrico endotelial3; el óxido nítrico (ON) induce vasodilatación y reducción de la presión arterial por medio de la inhibición de la contracción y crecimiento del músculo liso de la pared arterial4. La relación de estos polimorfismos con el riesgo de desarrollar hipertensión es todavía incierta5.
Uno de los obstáculos en la identificación de variantes genéticas asociadas a la hipertensión es la comparación de casos y controles que provienen de poblaciones con diferentes antecedentes genéticos. Este problema se conoce como estructura genética y genera un sesgo de selección, debido a que casos y controles tienen una distribución diferente de los alelos de los polimorfismos asociados a la enfermedad de interés6-8. En consecuencia, distintos métodos han sido propuestos para identificar y controlar la estructura poblacional en estudios de asociación genética9.
Teniendo en cuenta que en el contexto nacional y local son mínimos los estudios que involucren los citados polimorfismos, así como la deficiencia de investigaciones encaminadas a analizar su participación en la estructura genética en nuestra población, se diseñó un estudio genético poblacional en Bucaramanga, a partir de la genotipificación de los polimorfismos 448G>T, 679C>T y 1711C>T del gen GRK4 y Glu298Asp, -786T>C y el intrón 4 del gen eNOS, con el objeto de establecer el grado de estructura genética para esta población. Se espera que estos resultados den soporte a posteriores estudios de asociación de estos polimorfismos con la HAE en la población santandereana y así evitar posibles sesgos ocasionados ante la posibilidad de hallar asociaciones que podrían ser falsas.
MATERIALES Y MÉTODOS
Población de estudio
Se seleccionaron 552 individuos participantes en el proyecto INEFAC (Incidencia de Enfermedades Cardiovasculares y Factores de Riesgo en Colombianos), un estudio de cohorte en una muestra aleatoria de residentes de Bucaramanga-Colombia, de estratos socio económicos 2 y 3. La muestra incluyó 372 mujeres y 180 hombres entre 16 y 69 años de edad, con un promedio de 35,1 años y normotensos (presión arterial sistólica <120 mmHg y presión arterial diastólica <80 mmHg). Este estudio fue aprobado por el comité de ética de la Facultad de Salud de la Universidad Industrial de Santander y todos los participantes dieron su consentimiento informado por escrito.
El cálculo de la muestra se realizó teniendo en cuenta la frecuencia del alelo de menor prevalencia para los seis polimorfismos estudiados, en diferentes poblaciones10-13; adicionalmente se contemplaron los parámetros de nivel de confianza del 95%, poder de 86%, riesgo relativo esperado (RRE) de 2.0 y relación caso:control 1:1.
Extracción de ADN y métodos bioinformáticos
A todos los individuos se les tomó una muestra de sangre periférica con anticoagulante EDTA y a partir de ella se extrajo el ADN por el método de fenol-cloroformo14. Los polimorfismos de los genes GRK4 y eNOS fueron amplificados por medio de reacción en cadena de la polimerasa (PCR) y se identificaron por medio de restricción enzimática con identificación del tamaño de los fragmentos resultantes (RFLP's)15. Las secuencias genómicas de los genes GRK4 y eNOS y de los polimorfismos de interés fueron verificadas en las base de datos del National Center for Biotechnology Information (NCBI) de los Estados Unidos (http://www.ncbi.nlm.nih.gov) y (http://www.ncbi.nlm.nih.gov/projects/SNP). Los sitios de corte de cada una de las enzimas fueron verificados con el software RestrictionMapper (http://www.restrictionmapper.org/).
Amplificación por PCR y genotipificación de SNPsLas secuencias de los cebadores para amplificar cada uno de los polimorfimos se observan en el cuadro 1. Se utilizó la técnica de PCR-RFLP discordante para detectar los polimorfismos 679C>T y 1711C>T, en la que se cambió una base en uno de los cebadores de modo que se genera un fragmento que difiere en una base respecto del ADN molde, como consecuencia el producto amplificado del alelo ancestral adquiere un sitio de restricción que no está presente en el alelo mutando16.
La reacción de PCR para los polimorfismos del gen GRK4 contenía 1X de Buffer, 3,5 mM de MgCl2, 0,5 µM de cada cebador, 0,8 µM de desoxirribonucleótidos trifosfatados (dNTPs), 1 U de enzima Taq DNA polimerasa (Promega®) y 3,9 ng de ADN en un volumen final de 10µl. El protocolo de amplificación incluyó un paso inicial de 94°C por 5 minutos; seguido de 38 ciclos así: Denaturación a 95°C por 15 segundos, anillamiento a 60°C por 15 segundos y extensión a 72°C por 30 segundos y un paso final a 72°C por 7 minutos. En cada reacción de PCR se utilizaron controles negativos de amplificación y positivos para los alelos silvestres y mutados. Los productos de PCR fueron verficados mediante electroforesis en gel de agarosa al 1% incluyendo un marcador de peso molecular de 50 a 500pb, para verificar el tamaño del amplificado.
Los productos de PCR fueron sometidos a restricción empleando las enzimas reportadas en el cuadro 2, bajo el siguiente protocolo: 1X de buffer, 0,1ug/µl de BSA acetilada, 1U de la enzima de restricción correspondiente y 0,2ng del ADN amplificado, en un volumen final de 10 µl; la reacción se llevó a cabo por incubación durante 10 horas a 37°C13.
El montaje de la PCR para el polimorfismo Glu298Asp contenía 1X de Buffer-GoTaq® Green Master Mix, 0,4 µM de cada cebador y 2,4 ng de ADN, para un volumen final de 25µl y el protocolo de amplificación consistió en un paso inicial de 95°C por 2 minutos; seguido de 35 ciclos así: Denaturación a 95°C por 45 segundos, anillamiento a 63°C por 45 segundos y extensión a 72°C por 45 segundos y un paso final a 72°C por 5 minutos. El montaje de la PCR para el polimorfismo -786T>C contenía 1X Buffer, 2,5 mM de MgCl2, 0,4 µM de cada cebador, 0,8 µM de dNTPs, 1 U de enzima Taq DNA polimerasa (promega®) y 2,4 ng de ADN en un volumen final de 25 µl. El protocolo de amplificación incluyó un paso inicial de 94°C por 4 minutos; seguido de 35 ciclos así: Denaturación a 94°C por 30 segundos, anillamiento a 63°C por 30 segundos y extensión a 72°C por 1 minuto y un paso final a 72°C por 5 minutos. La amplificación de estos polimorfismos fue verificada mediante electroforesis en gel de agarosa al 1%, los productos de PCR fueron sometidos a restricción enzimática empleando el siguiente protocolo: 1X de buffer, 0,1ug/µl de BSA acetilada, 1U de la enzima de restricción correspondiente (cuadro 2) y 0,5 ng de ADN en un volumen final de 15 µl; la reacción se llevó a cabo porincubación durante 14 horas a 37°C. Los productos de las digestiones enzimáticas de los SNPs estudia dos se separaron por medio de electroforesis en geles de agarosa al 3% y para la visualizacion de las bandas se adicionó bromuro de etidio. Los geles se corrieron en una cámara de electroforesis Power Pac 300 (BioRad®) en buffer TBE 1X y se aplicó una tensión de 60 voltios por 80 minutos. Los sitios de reconocimiento de las enzimas, los tamaños de los fragmentos esperados y los genotipos asignados se muestran en el cuadro 2.
El montaje de la PCR para el intrón 4, se realizó con el mismo protocolo del polimorfismo -786T>C. Los tamaños de los fragmentos de este polimorfismo, producto de la amplificación, se visualizaron mediante electroforesis en gel de agarosa al 3% con una tensión de 60 voltios por 80 minutos. En el cuadro 3, se observa el tamaño de los productos de la amplificación de este intrón17.Los análisis de todas las muestras fueron efectuados en forma enmascarada para evitar sesgos y el 10% de las muestras se procesaron por duplicado sin que se presentara ninguna discrepancia.
Secuenciación
Una vez estandarizada la PCR, se seleccionaron seis muestras de cada polimorfismo para ser secuenciadas con el kit Big Dye terminator (Applied Biosystems®) y las secuencias obtenidas se alinearon con el software Clustal W (http://www.ebi.ac.uk/Tools/msa/clustalw2/) para confirmar la correspondencia con los fragmentos esperados.
Análisis estadísticos
Con los resultados obtenidos de la lectura de las electroforesis se construyeron dos bases de datos en Excel® Microsoft 2007 validadas con la aplicación Data Compare del Software Epi Info versión 3.5.118 y se estableció el genotipo para cada polimorfismo tipificado en las muestras. Con estos datos se calcularon para cada uno de los polimorfismos estudiados, las frecuencias genotípicas con el programa GenAlex 6.3®19; las frecuencias alélicas, las frecuencias haplotípicas, el test de Hardy-Weinberg (EHW) y el análisis de varianza molecular (AMOVA) para determinar la presencia de estructura genética poblacional, se realizaron con el programa Arlequín v 3.520; por último se verificó la estructura genética poblacional mediante el uso del programa Structure v 2.321.
RESULTADOS
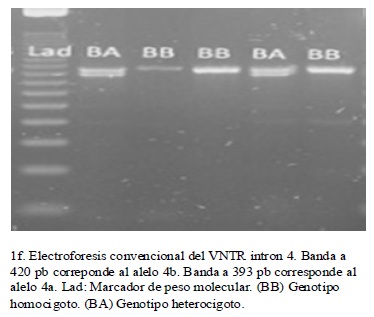
La tipificación por RFLP de los diferentes polimorfismos analizados, permitió detectar todos los genotipos posibles de cada uno de ellos, en la población estudiada.(figura 1) A partir de los genotipos observados se calcularon las frecuencias genotípicas y las alélicas para cada uno de los polimorfismos de los genes GRK4 y eNOS, así como se calcularon las frecuencias haplotípicas para los seis polimorfismos combinados. Se estableció que todos los polimorfismos estuvieron en EHW, puesto que los valores de p hallados fueron superiores a 0.05. (cuadro 4)
De acuerdo al AMOVA jerarquizado, no se evidenció estructura genética (FST = 0,0038) en la muestra analizada. Adicionalmente no se observó que los valores de FST obtenidos para cada polimorfismo estudiado (cuadro 4), contribuyeran significativamente en la diferenciación poblacional22.
La evidencia de no estructura fue confirmada por el análisis realizado con el software Structure v 2.3, al evaluar con K=2 (posibles ancestros) y con 10.000 réplicas asumiendo el modelo de mezcla21. Los resultados mostraron muy pocos individuos que eventualmente pueden pertenecer a otro grupo poblacional, además de evidenciar la ausencia de estructura genética en la muestra poblacional analizada (Figura 2A y 2B).
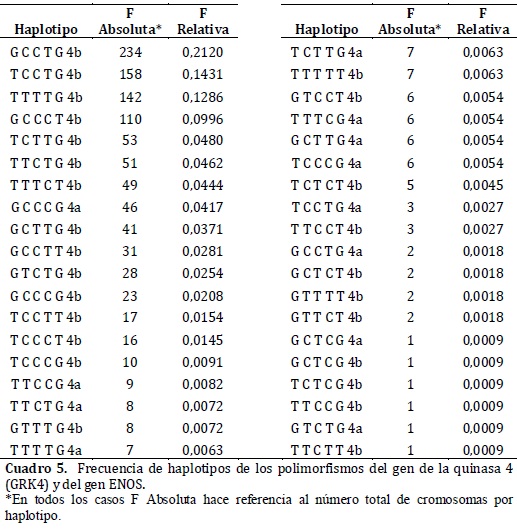
En la estimación de las frecuencias haplotípicas para los seis polimorfismos estudiados, se encontró, que el más frecuente fue el GCCTG4b (21.2%) de un total de 38 haplotipos detectados en 1104 cromosomas analizados.(cuadro 5)
DISCUSIÓN
De acuerdo con los valores obtenidos para las frecuencias alélicas en la población estudiada, el alelo más frecuente para cada polimorfismo del gen GRK4 fue: 448G>T alelo T (50.7%), 679C>T alelo C (70%), 1711C>T alelo C (69%), en un estudio previo realizado en población hispana del sur de California, los resultados coinciden para los polimorfismos 679C>T y 1711C>T y difieren para el polimorfismo 448G>T23.
El alelo más frecuente en cada polimorfismo del gen eNOS fue: -786T>C alelo T (76%), Glu298Asp alelo G (72%) y el intrón 4 alelo 4b (90%), estos resultados coinciden con los hallados en un estudio previo realizado en población bumanguesa15.
En el presente estudio se hallaron 38 combinaciones de haplotipos, siendo GCCTG4b el más frecuente con 21.2%, no se encontraron publicaciones en poblaciones donde se reporten frecuencias haplotípicas para estos seis polimorfismos, en población hispana ni colombiana, convirtiendo el presente estudio en el primer reporte de las mismas.
Todos los polimorfismos estudiados se encontraron en equilibrio de Hardy-Weinberg lo que indica que la población está conformada por individuos que se mezclan al azar, hallazgo que coincide con lo reportado en estudios previos en población bumanguesa para el gen eNOS15 y en población hispana del Sur de California para el gen GRK423, donde no se cuenta con datos reportados para población colombiana.
Adicionalmente no se encontró estructura poblacional en la muestra analizada, lo que coincide con lo reportado en un estudio previo realizado en la población de Bucaramanga, a partir del análisis de otros marcadores genéticos polimórficos24.
CONCLUSIONES
Los resultados hallados confirman que la población estudiada se encontró en EHW para todos los sistemas estudiados y no presen ta subestructura poblacional,lo que permite adelantar un estudio de asociación de estos polimorfismos con la hipertensión arterial esencial, puesto que es claro que las asociaciones entre genes candidatos a desarrollar enfermedades multifactoriales deben ser interpretadas dentro del contexto de la estructura genética de la población que está siendo estudiada.
CONFLICTO DE INTERESES
Los autores declaran que no hay conflicto de intereses en el presente manuscrito.
AGRADECIMIENTOS
Los autores manifiestan su agradecimiento al Instituto Colombia-no para el Desarrollo de la Ciencia y la Tecnología Francisco José de Caldas y a la Vicerrectoría de Investigación y Extensión de la Universidad Industrial Santander (UIS). A la Dra Leonor Gusmao, investigadora senior del Instituto de Patología e Inmunología Molecular de la Universidad de Oporto (IPATIMUP) y al Dr. Henry Bautista del Departamento de virología e Inmunología de Southwest Foundation for Biomedical Research.
FUENTES DE FINANCIACIÓN
Colciencias proyecto código 110240820434 de la convocatoria 408 de 2007 y la Vicerrectoría de investigación y extensión de la Universidad Industrial de Santander.
REFERENCIAS
1. Hopkins PN, Hunt SC. Genetics of hypertension. Genet Med. 2003; 5: 413-29. [ Links ]
2. Premont RT, Macrae AD, Stoffel RH, Chung N, Pitcher JA, Ambrose C, et al. Characterization of the G Protein-coupled Receptor Kinase GRK4. Identification of four splice variants. The Journal of Biological Chemistry. 1996; 271: 6403-410. [ Links ]
3. Hingorani AD. Endothelial Nitric Oxide Synthase Polymorphisms and Hypertension. Current Hypertension Reports. 2003; 5: 19-25. [ Links ]
4. Bautista LE. Inflammation, endothelial dysfunction, and the risk of high blood pressure: Epidemiologic and biological evidence. J Hum Hypertens. 2003; 17: 223-30. [ Links ]
5. Casas JP, Cavalleri GL, Bautista LE, Smeeth L, Humphries SE, Hingorani AD. Endothelial Nitric Oxide Synthase Gene Polymorphisms and Cardiovascular Disease: A HuGE Review. Am J Epidemiol. 2006; 64: 921-35. [ Links ]
6. Marchini J, Cardon LR, Phillips MS, Donnelly P. The effects of human population structure on large genetic association studies. Nat Genet. 2004; 36: 512-17. [ Links ]
7. Marroni A, Metzger I, Souza-Costa D, Nagassaki S, Sandrim V, Correa R, et al. Consistent interethnic differences in the distribution of clinically relevant endothelial nitric oxide synthase genetic polymorphisms. Nitric Oxide. 2005; 12: 177-82. [ Links ]
8. Iniesta R, Guinó E, Moreno V. Análisis estadístico de polimorfismos genéticos en estudios epidemiológicos. Gac Sanit. 2005; 19: 333-41. [ Links ]
9. Pritchard JK, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data. Genetics. 2000; 155: 945-59. [ Links ]
10. Bengra C, Mifflin TE, Khripin Y, Manunta P, Williams SM, Jose PA, et al. Genotyping of Essential Hypertension Single-Nucleotide Polymorphisms by a Homogeneous PCR Method with Universal Energy Transfer Primers. Clinical Chemistry. 2002; 48: 2131-140. [ Links ]
11. Speirs HJ, K Katyk, Kumar NN, Benjafield AV, Wang WY, Morris BJ. Association of G-protein-coupled receptor kinase 4 haplotypes, but not HSD3B1 or PTP1B polymorphisms, with essential hypertension. Journal of Hypertension. 2004; 22: 931-36. [ Links ]
12. Williams SM, Ritchie MD, Phillips JA, Dawsone E, Prince M, Dzhura E, et al. Multilocus Analysis of Hypertension: A Hierarchical Approach. Hum Hered. 2004; 57: 28-38. [ Links ]
13. Wang Y, Li B, Zhao W, Liu P, Zhao Q, Chen S, et al. Association study of G protein-coupled Receptor Kinasa 4 gene variants with essential hypertension in Northern Han Chinese. Annals of Human Genetics. 2006; 70: 1-6. [ Links ]
14. Valverde E, Cabrero C, Cao R, Rodríguez-Calvo MS, Díez A, Barros F, et al. Population genetics of three VNTR polymorphisms in two different Spanish populations. International Journal of Legal Medicine. 1993; 105: 251-56. [ Links ]
15. Serrano NC, Díaz LA, Casas JP, Hingorani AD, Moreno de Lucca D, Páez MC. Frequency of eNOS polymorphisms in the Colombian general population. BMC genetics. 2010; 11: 54. [ Links ]
16. Kwok S, Chang SY, Sninsky JJ, Wang A. A guide to the design and use of mismatched and degenerate primers. Genome Research. 1994; 3: 39-47. [ Links ]
17. Serrano N, Casas J, Diaz L, Paez C. Endothelial NO Synthase Genotype and Risk of Preeclampsia A Multicenter Case-Control Study. Hypertension. 2004; 44: 702-07. [ Links ]
18. Dean AG, Arner TG, Sunki GG, Friedman R, Lantiga M, San gam S, et al. Epi Inf version 3.5.1, a database and statistics program for public health professionals. Centers for Disease Control and Prevention, Atlanta. 2002. [ Links ]
19. Peakall R, Smouse PE. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research. Molecular Ecology Notes. 2006; 6: 288-95. [ Links ]
20. Excoffier L, Laval G, Schneider. Arlequin ver. 3.5: Anintegrated software package for population genetics data analysis. Evolutionary Bioinformatics Online. 2005; 1: 47-50. [ Links ]
21. Pritchard JK, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data. Genetics. 2000; 155: 945-59. [ Links ]
22. Hartl DL, Clark AG. Organization of genetic variation: Linkage and Linkage disequilibrium. En: Principles of population genetics. Sunderland Massachusetts. Sinauer Associates Inc. 1997; 71-109. [ Links ]
23. Lohmueller K, Wong L, Mauney M, Jiang L, Felder R, Jose P, et al. Patterns of Genetic Variation in the Hypertension Candidate Gene GRK4: Ethnic Variation and Haplotype Structure. Annals of Human Genetics. 2005; 70: 27-41. [ Links ]
24. Hincapié ML, Gil AM, Pico AL, Gusmão L, Rondon F, Castillo A. Análisis de la estructura genética en una muestra poblacional de Bucaramanga, departamento de Santander. Colombia Médica
[ Links ]