Los elementos móviles pueden constituir aproximadamente el 45% del genoma humano. Entre ellos el grupo más abundante lo compone la familia de elementos Alu. Las inserciones de Alu son marcadores bialélicos que se definen por la ausencia y presencia de unas secuencias de aproximadamente 300 pb, que con unas variaciones más o menos importantes derivan del gen 7SL RNA, el cual forma parte del complejo ribosomal por retrotransposición. Son especialmente interesantes en antropogénica por aparecer hace 65 millones de años y encontrarse con el origen y la expansión de los primates (Batzer & Deininger, 2002). La mayoría de las inserciones Alu son recientes, por lo que en general son polimórficas, pudiendo tomar diferentes valores de frecuencias en distintas poblaciones humanas (Stoneking et al., 1997). Es altamente improbable que la misma inserción Alu ocurra dos veces independientemente en el mismo lugar, por lo que se consideran eventos mutacionales únicos (Batzer & Deininger, 1994).A nivel mundial se han hecho diferentes reportes de los elementos Alu en diferentes poblaciones, Novick et al. (1998) comparó cinco inserciones Alu en treinta poblaciones de las cuales veinticuatro fueron del continente americano y seis de otras partes del mundo, reportando que las relaciones entre las poblaciones tenían una correlación con la distribución geográfica dentro del continente americano, lo que confirma la sensibilidad con que los marcadores Alu permiten comparar y asociar poblaciones. En Colombia, factores geográficos, climáticos e históricos han contribuido a los patrones de variación genética humana vistos en la actualidad y a nivel mundial. Por ejemplo, los procesos de población asociados a la colonización, los periodos de aislamiento geográfico, la endogamia socialmente reforzada y la selección natural todos han afectado las frecuencias de alelos en ciertas poblaciones (Jorde, Bamshad & Rogers, 1998; Jorde & Wooding, 2004). Sin embargo, la actualidad de nuestra ascendencia común y el flujo continuo de genes entre los grupos humanos han limitado la diferenciación genética en nuestra especie (Jorde & Wooding, 2004).
Las poblaciones del Caribe colombiano se caracterizan por ser híbridas o mezcladas, se tiene un aporte genético particular de los diferentes grupos amerindios, indígenas, afrodescendientes y europeos llegados de España y Portugal que conforman cada una de las poblaciones de Colombia (Gutiérrez, 1980). Tanto en Colombia como en el departamento de Córdoba se presenta un alto mestizaje y se desconoce su diversidad genética aún más en trabajos relacionados con los elementos Alu, con excepción del reporte de las estimaciones de mezclas genéticas por elementos Alu en poblaciones afrocolombianas y mestizas en Antioquia, donde se determinaron los procesos de mezclas que ocurren en las poblaciones de América Latina al examinar los perfiles genéticos de dos grupos étnicos de Antioquia (Gómez et al. 2010). En el municipio de San Pelayo hay un gran mestizaje, debido a la colonización de extranjeros españoles, mestizos, blancos y negros. Sin embargo, en este municipio ha aparecido en muchas generaciones el cruzamiento entre familias, lo que hay traído un problema que afecta como tal el deterioro de la diversidad genética en las próximas generaciones. Por lo tanto, en esta investigación se determinó la diversidad genética y la estructura poblacional en humanos de San Pelayo, utilizando cinco polimorfismos de inserción Alu.
Materiales y métodos
Área de estudio. Las 40 muestras fueron colectadas así: 20 en la subpoblación San-Pelayo Urbano (8° 57' 28'' N, 75° 50' 15'' O) y 20 en Sabana-Nueva (8° 58' 1''N, 75° 51' 0'' W) en el departamento de Córdoba, Colombia.
Obtención de muestras. Las muestras requeridas para el análisis genético se obtuvieron de adultos voluntarios no emparentados entre sí, residentes estables de la población del estudio, desde al menos tres generaciones. Se recolectaron 2 ml de saliva por cada individuo en dos tubos eppendorf de 1,5 ml, después se adicionó solución de lisis celular 500 μl; los participantes en el estudio firmaron el respectivo consentimiento informado. Posteriormente, las muestras fueron almacenadas en una cava con una temperatura de 4° C para ser trasladadas hasta el laboratorio de genética de la Universidad de Córdoba.
Aspectos éticos. Este trabajo cuenta con la aprobación del comité de ética para investigación en humanos de la Facultad de Ciencias de la Salud de la Universidad de Córdoba.
Extracción de ADN. Para la extracción de las 40 muestras de ADN se utilizó el kit de extracción y purificación de Wizard Genomic DNA Purification (Promega) y se siguieron las recomendaciones del fabricante sin modificaciones. La pureza y la concentración del ADN fue determinada utilizando 1 μL de la muestra en un espectrofotómetro Nanodrop 2000® (Thermo Scientific, Waltham, MA, EE. UU.) con relaciones de absorbancia A260/A280 nm y A260/A230 nm. La integridad del ADN se determinó de manera visual mediante electroforesis en geles de agarosa al 1.0%, con un voltaje de 120 V por 2 h en buffer TBE IX (Tris-HC1 500 mM, ácido bórico 60 mM y EDTA 83 mM) y agente de tinción GelRed IX. El gel se visualizó en un transiluminador Bio-Imagen System 312 nm, Neve Yamin, Israel.
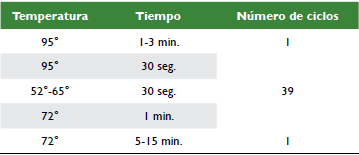
Amplificación por PCR. Los cebadores Alu: D1 (Batzer et al., 1995), FXIII, APO y PV92 (Batzer et al., 1994) y ACE (Tired et al., 1992) se observan en la tabla 1, fueron amplificados mediante la técnica de la PCR.
Tabla 1 Características de las secuencias de los elementos Alu

LC: localización cromosómica; SF: subfamilia, T °C: temperatura de hibridación
La técnica de la PCR (tabla 2) se realizó en un volumen total de 25 μl que contenía 0,25 μl de Taq polimerasa (Thermo Scientific) (Uklam-EE. UU.), 1,25 μl de cada primer (forward y reverse), 0,5 μl de dNTPs a 10 mM /μl, 2,5 μl de buffer de reacción a 10X, 1,25 μl de MgCl2, 5 μl de ADN y agua estéril hasta alcanzar el volumen final. La reacción de PCR se realizó en un termociclador Bioard T100 (Los Ángeles, EE. UU.) mediante la técnica PCR Tochdown.
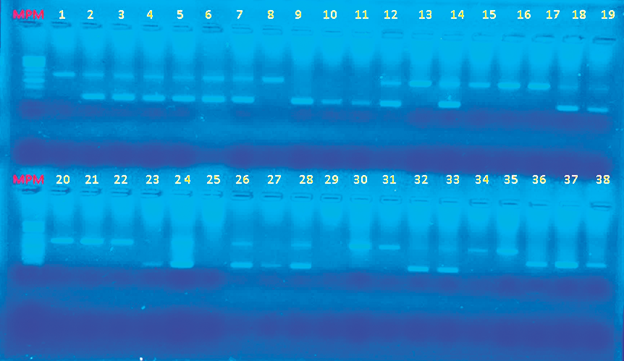
Visualización y cuantificación del ADN. Los amplificados de la PCR fueron sometidos a electroforesis en gel de agarosa al 2%, a 100 voltios durante 40 minutos, estos resultados fueron visualizados bajo luz ultravioleta en una cámara de (Cleaver Scientific) (Londres, UK). Además, en el gel de agarosa se añadió un marcador de peso molecular de separación 500 pb, con el objeto de identificar el tamaño de las bandas de la PCR (figura 1).
Análisis estadístico. Para la determinación de los parámetros de diversidad genética: frecuencias alélicas, heterocigosidad observada y esperada, equilibrio de Hardy-Weinberg, y estadísticos F de Wright se utilizó el software GenAlex 6.502 (Peakall & Smouse, 2012).
Resultados
En la figura 1 se puede observar la electroforesis de 38 muestras para el cebador ACE (Pb 490/190), donde se pueden observar homocigotos con la inserción: individuos 1,8, 13, 15, 16, 17, 21, 22, 23, 30,31, 34, 35, heterocigotos: individuos 2, 3, 4, 5, 6, 7, 12, 14, 18, 19, 24, 26, 28 y homocigotos sin inserción: individuos 9, 10, 11, 23, 27, 32, 33.
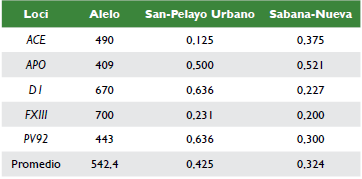
En la tabla 3 se observan los valores de las frecuencias alélicas obtenidas para los marcadores Alu, en las dos subpoblaciones estudiadas, todos los loci analizados resultaron polimórficos. Al determinar las frecuencias alélicas para cada subpoblación (tabla 3) se obtuvo que para San Pelayo urbano y Sabana-Nueva, las inserciones D1, PV92 y APO muestran los mayores valores y las menores frecuencias se encontraron para los marcadores ACE y FXIII respectivamente. La frecuencia alélica promedio resultó mayor en San Pelayo Urbano (0,425) que en Sabana-Nueva (0,324).
En la tabla 4 los valores obtenidos para el equilibrio de Hardy-Weinberg en las dos subpoblaciones mostraron ausencia de valores equilibrio de Hardy-Weinberg (p < 0,05) para los cinco loci analizados. La heterocigosidad observada (Ho) mostró valores entre 0,042 y 0,000 Los valores de heterocigosidad esperada (He) varían entre 0,219 para la subpoblación de San Pelayo Urbano hasta 0,500 y para la subpoblación de Sabana-Nueva se observaron valores de 0,320 y 0,499. El índice de fijación indica un valor de 1,000 en la subpoblación de San-Pelayo urbano en general, pero en la subpoblación de Sabana-Nueva solo el loci APO tuvo un valor de 0,917.
Tabla 4 Parámetros genéticos calculados en la zona de estudio
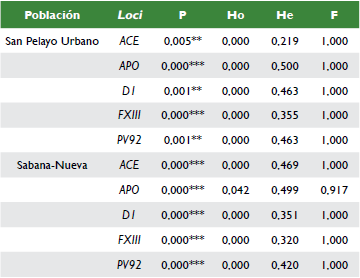
P: equilibrio Hardy-Weinberg (** p < 0,01, *** p < 0,001), Ho: heterocigosidad observada, He: heterocigosidad esperada, F: índice de fijación
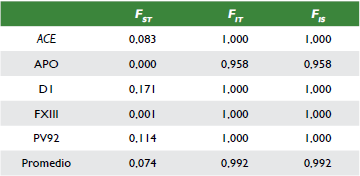
En la tabla 5 los valores de FST varían de 0,00 en el marcador APO a 0,17 para el locus D1. En el caso de las FIS y FIT mostraron valores de 0,958 a 1,000.
Discusión
Los cinco marcadores analizados resultaron polimórficos en las dos subpoblaciones, las inserciones Alu mostraron valores heterogéneos dentro del rango de las frecuencias alélicas. Se observa una afinidad con las frecuencias alélicas de las poblaciones europea-americana, y la africana-americana (Watkins et al., 2001). Los valores promedios de las frecuencias alélicas de San Pelayo Urbano fueron más cercanos a las reportadas en Rongmei, Inpui y Kuki y las de Sabana Nueva a las de Zeme Liangmai (Kshatriyaa, Panmeib & Kameihc, 2019).
La heterocigosidad observada (Ho) presentó valores bajos en comparación con lo reportado para las poblaciones de América y de otros lugares del mundo (Stoneking et al., 1997) lo cual pudo ser ocasionado por la endogamia. Los valores de la heterocigosidad esperada (He) fueron superiores debido a la homogeneidad de las frecuencias alélicas en los marcadores, por tanto, lo que se espera en la siguiente generación es un número de homocigoto mucho más altos que de heterocigotos, estos resultados de heterocigosidad esperada se asemejan a lo reportado para las poblaciones amerindias, las poblaciones africanas y las poblaciones asiáticas (Antúnez et al., 2002; Battilana et al., 2006).
Los cinco marcadores no estuvieron en equilibrio de Hardy-Weinberg para las subpoblaciones, esto pudo deberse a diferentes eventos como el efecto Wahlund, suceso que no afecta por igual a todo el genoma y produce un aumento de homocigotos y una disminución de heterocigotos, lo cual está en concordancia con los resultados obtenidos y también por la endogamia originada por un aislamiento de las dos subpoblaciones: en la historia de San Pelayo, durante el proceso de colonización entre los años 1690 y 1721, se reporta que las familias del municipio se casaban entre ellas (Fortich, 2014) y para el caso de Sabana Nueva en su reseña histórica se afirma que esta fue fundada por españoles que llegaron vía fluvial a finales del siglo XVIII, donde al igual que en San Pelayo, predominó un entrecruzamiento entre las familias fundadoras para poder mantener su abolengo en las siguientes generaciones, por esto es común encontrar apellidos que predominan como Petro, Hernández, Doria y Cantero, considerados en los estudios antropológicos como los primeros pobladores de esta localidad y determinantes para el predominio del color de piel de estos pobladores (Fortich, 2014). La ausencia del equilibrio de Hardy Weinberg obtenida en esta investigación, fue similar a los resultados reportados en poblaciones amerindias colombianas para marcadores Alu (Novick et al., 1998).
Los valores obtenidos para el índice de fijación mostraron que las dos subpoblaciones presentaron un aislamiento genético. Respecto a los estadísticos F se observó que en los valores de FST mostraron valores cercanos a cero en casi todos los marcadores, indicando que las dos sub-poblaciones se comportan como una sola población; estos resultados son menores a lo reportado en poblaciones de Europa, África y afrocolombianas (Gómez et al., 2010, Terreros et al., 2009). Además, la diversidad genética obtenida en el presente estudio puede atribuirse a variaciones individuales dentro de las poblaciones, ya que solo el 7,4% de la diversidad genética total proviene de la variación entre las poblaciones (FST).
Al mismo tiempo, las FIS Y FIT ratifican el déficit de heterocigoto, estos resultados se dan por consecuencia de apareamientos consanguíneos. Estos valores obtenidos no poseen similitud con las poblaciones brasileñas (Cotrim et al., 2004).
Conclusiones
La población de San Pelayo, Córdoba (Colombia) presentó una baja diversidad genética, y un alto nivel polimórfico en todos los elementos Alu. El exceso de homocigotos que hay en toda la población es producido por la endogamia, lo cual aumenta la posibilidad de pérdida de los alelos para las siguientes generaciones, en consecuencia, esto llevaría a la disminución de la diversidad genética a lo largo del tiempo.