INTRODUCCIÓN
Physalis peruviana, conocida como uchuva o baya dorada, es un frutal tropical exótico de la familia Solanaceae, nativa de la región Andina, principalmente Colombia, Perú y Ecuador. Su fruto presenta alto niveles de vitaminas (A, B y C) y minerales (hierro y fósforo) y ácidos grasos polinsaturados que le confieren propiedades antioxidantes, anti-inflamatorias y anticancerígenas [1,2]. Actualmente es el segundo frutal de exportación de Colombia, con alta demanda en los mercados Europeos, debido a su atractivo sabor, color y forma como por su potencial medicinal. Para el año 2014 fueron cultivadas un total de 932 ha con una producción de 13.260 ton y un rendimiento de 9,81 t/ha, concentrada en los departamentos de Boyacá (58%), Antioquia 817%) y Cundinamarca (15%) [3[. A pesar de su importancia económica sigue siendo un cultivo semidomesticado y no se conocen cultivares resistentes ni variedades mejoradas en Colombia [4].
Estudios de colecta, conservación y caracterización son necesarios para el planteamiento de estrategias de mejoramiento genético de esta especie, en este sentido, el país cuenta con colecciones de germoplasma cultivado y silvestre procedente de los principales municipios productores manejado por centros de investigación y universidades [4], las cuales han sido evaluadas por características biológicas, morfológicas, agronómicas, moleculares y de resistencia al principal problema fitosanitario que actualmente enfrentan los agricultores en Colombia, Fusarium oxysporum [4,5,6,7,8,9]. Se ha avanzado en el conocimiento de la variabilidad y de taxones relacionados usando tecnologías de secuenciación de última generación a partir de los cuales se han desarrollado diferentes marcadores moleculares [5,7]. Estudios de caracterización de la diversidad y estructura genética de poblaciones de Physalis han desarrollados por Enciso et al. (2013) [6], quienes detectaron SNPs asociados a la resistencia a F. oxysporum; Osorio (2016) [8], usando marcadores SNPs concluyeron que la estructura genética de 100 accesiones de uchuva estaba asociada principalmente con estado del cultivo (silvestre o cultivado) y no por el sitio geográfico de donde proceden los materiales, resultados similares fueron obtenidos por Garzón et al. (2015) [9 ], quienes evaluaron 47 accesiones de P. peruviana con 642 SNP y 24 loci InDels (Inserción o Deleción).
Entre los marcadores moleculares más utilizados para evaluar la diversidad genética en plantas están los microsatélites o SSR (Secuencias Simples Repetidas) que son altamente polimórficos y de herencia codominante [10]. En uchuva, Martínez et al. (2015) [5] caracterizaron 222 individuos de Corpoica usando marcadores COSII, IRGs y SNPs, en donde se estableció la estructura y la diversidad genética de estos materiales. A partir de estas secuencias, se han desarrollado marcadores SSR en regiones codificantes y no codificantes tanto en P. peruviana como P. floridana Rydb [11]. Chacón et al. (2016) [4], estudiaron la estructura genética de 85 accesiones cultivadas y no cultivadas de uchuva procedentes de las cordilleras central, occidental y Oriental usando 15 loci SSRs, encontrando una baja diversidad genética y que las cordilleras Andinas juegan un papel importante en la estructuración genética de la uchuva Colombiana más que el estado biológico (silvestre o cultivado).
Los marcadores Microsatélites Amplificados al Azar (RAMs) también conocidos como ISSR (Intersimple Sequence Repeat) son útiles para medir la diversidad genética en plantas, animales y microorganismos y en identificar las diferencias existentes entre familias, especies y al interior de las especies, su metodología es factible para pequeños laboratorios y en estudios de exploratorios de la diversidad genética; además han sido utilizados en la caracterización de la diversidad genética en especies de frutales andinos permitiendo la discriminación de los materiales evaluados [12,13].
Teniendo en cuenta que la uchuva es considerada un frutal promisorio para Colombia y que Boyacá es uno de los departamentos pioneros en la producción nacional, se plantea el presente trabajo de investigación que tiene como objetivo caracterizar la variabilidad genética de los materiales de P. peruviana en los principales municipios productores de Boyacá como una primera aproximación hacia el conocimiento del germoplasma y el planteamiento de estrategias de conservación y mejoramiento genético de esta especie en la región.
MÉTODO
Material vegetal
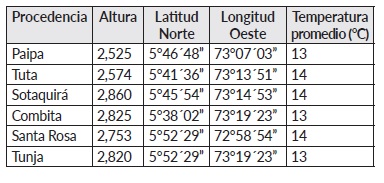
Se colectaron hojas jóvenes de 15 materiales de uchuva con características rústicas que se encontraban en huertos caseros, bordes de carretera o de cultivos comerciales en los municipios de Paipa (3), Sotaquirá (1), Combita(2,), Tuta (2), Santa Rosa (3) de Viterbo y Tunja(4) (Cuadro 1 ).
Caracterización molecular
La caracterización molecular se hizo en los laboratorios de investigación en Biología Molecular, BIOPLASMA y GEBIMOL, de la Universidad Pedagógica de Colombia, Tunja. Para la extracción de ADN se utilizó el protocolo de Dellaporta et al. (1983) [14]. Los ADN totales se visualizaron en geles de agarosa al 0,8%, en una cámara Maxicell Primo EC-340 Electroforesis Gel System. Para determinar la concentración de ADN se utilizó el fluorómetro Hoefer Dyna Quant 200 y se diluyó en agua tipo HPLC a un volumen total de 100 μL a 10 ng/μL y se almacenó a -20ºC. Para el análisis RAMs se utilizaron siete cebadores sintetizados por Technologies Inc. Bioneer (Cuadro 2 ). Para la reacción de amplificación con RAMs se preparó el cóctel en un tubo estéril de microcentrífuga (1,5 mL) para un volumen final de 25 μL. La mezcla de reacción se preparó con buffer 1X, MgCl2 1;5 mM, dNTPs 0,2 mM, Taq Polimerasa 1U, cebador 2 μM y ADN genómico 10 ng.
Cuadro 2 Cebadores utilizados en la técnica Microsatélites RAMs.

Las siguientes designaciones se usan para los sitios degenerados: H (A ó T ó C); B (G ó T ó C); V (G ó A ó C) y D (G ó A ó T).
La amplificación se llevó a cabo en un termociclador PTC 100 Programmable Termal Controller (MJ. Research, Inc), en el laboratorio de molecular. La desnaturalización inicial fue a 95ºC durante 5 min; desnaturalización a 95ºC por 30 seg, hibridación a una temperatura de 50ºC (cebador AG y CA), 55ºC (cebador CCA-TG-CT) y 58ºC (cebador CGA) durante 45 seg, una extensión de 72ºC por 2 min, 37 ciclos desde la desnaturalización a extensión y por último una extensión a 72ºC durante 7 min. Los productos amplificados fueron separados por electroforesis en geles de poliacrilamida 37:1 (acrilamida: bisacrilamida) al 7% a 150 v por 1 h en una cámara pequeña de DNA Sequencing System, FB-SEQ-3545 de Fisher Biotechnologies. La tinción se realizó usando sales de plata.
Análisis Estadístico
Se generó una matriz binaria de ausencia (cero) y presencia (uno). La similitud genética entre los individuos se calculó utilizando el coeficiente de similitud de Nei y Li (1979) [15]. El análisis de agrupamiento se realizó por el método UPGMA y se generó un dendrograma utilizando el paquete estadístico NTSYS (Numerical Taxonomy System for personal Computer, versión 2.02 PC). Para evaluar la diversidad genética se estimó la heterocigosidad insesgada y el porcentaje de loci polimórficos utilizando el paquete estadístico TFPGA (Tools For Population Genetic Analices, versión 1.3, 1997). Se determinó el f estadístico insesgado con un intervalo de confianza del 95 % y se realizó el Análisis de Varianza Molecular (AMOVA) con el programa GEnALex 6.5.
RESULTADOS
El análisis mediante el coeficiente de Nei-Li a una similitud de 0,65 diferenció a la población en tres grandes grupos (Figura 1). El grupo I formado a una similitud genética de 0,70 está conformado por materiales de uchuva procedentes de los municipios de Paipa, Tunja y Combita. Se pude observar que los materiales de Paipa tiene una mayor homogeneidad (0,85) y se presenta una distribución más laxa de los materiales de Tunja y Combita; hay que considerar que estos sitios se encuentran a menos de 100 km de distancia por lo cual el flujo genético y el intercambio de material de siembra pueden ocurrir con mayor frecuencia.
En el grupo II se encuentran a un nivel de similitud de 0,70 los materiales de Santa Rosa y Combita, los cuales comparten algunas características morfológicas y agronómicas interesantes (González, 2016 datos sin publicar). Una vez se evidencia el flujo de material que existe entre las diferentes zonas productoras del departamento. A un nivel de similitud de 0,63 se encuentran los materiales procedentes de Santa Rosa de Viterbo (Grupo III) en donde se encuentra el material genético más diverso de todos los evaluados en este estudio el cual debe ser evaluado por sus características morfológicas, agronómicas, nutricionales. Chacón et al. (2016) [4] estudiaron la estructura genética de germoplasma de uchuva colombiano encontraron un valor promedio fue de 0,13, lo cual puede ser debido a la naturaleza de los materiales evaluados y el agrupamiento determinado por las presiones de selección existentes en las cordilleras.
En general, los agrupamientos en este estudio se presentaron de acuerdo al origen geográfico de donde proceden los materiales y no por su estado biológico como había sido reportado por Garzón et al. (2015) [9] y Osorio (2016) [8], quienes no encontraron ninguna relación entre la estructura genética y la geografía pero si con el estado biológico. Osorio (2016) [8], encontró tres grupos de accesiones, un grupo formado principalmente por accesiones silvestres, otro grupo con accesiones cultivadas de fincas tradicionales y un tercer grupo con accesiones cultivadas en campos comerciales, siendo los departamentos de Cundinamarca, Boyacá, Santander y Norte de Santander (Oriente de los Andes) en donde se encontraron la mayoría de los materiales silvestres por lo tanto son áreas potenciales para trabajos de conservación y mejoramiento genético de Physalis.
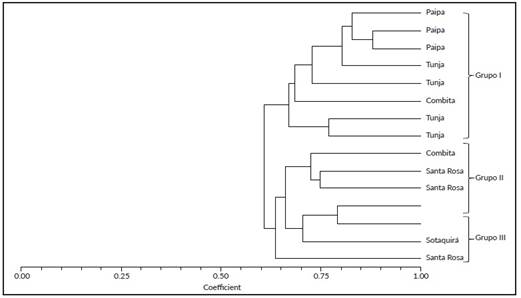
Figura 1 Dendrograma de los materiales de uchuva (P. peruviana L.) basado en el Coeficiente de similitud de Nei-Li.
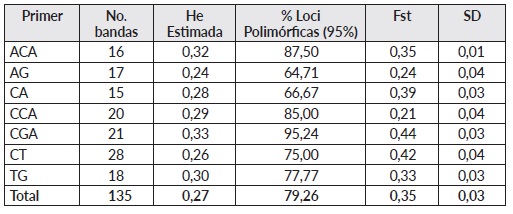
Los siete cebadores RAMs generaron un total de 135 bandas, 15 para el cebador CA y 28 para el CT, con pesos moleculares entre 250 y 1,200 Kb, el número de bandas obtenidas en este estudio se considera adecuado para la estimación de los parámetros genéticos [5,8,12]. El cebador CGA fue el que mayor aporte hizo a la variación genética observada con un Fst de 0.44, lo cual muestra que es un marcador molecular que debe ser tenido en cuenta a la hora de evaluar germoplasma de Physalis o especies relacionadas (Cuadro 3 ).
Los valores de heterocigosidad promedio estimada estuvieron comprendidos en un rango entre 0,24 y 0,33 para los cebadores Ag y CGA respectivamente, con porcentajes de loci polimórficos de 65 al 95%. El valor promedio para la población total fue de 0,27 con una desviación estándar de 0,03 (Cuadro 3), menor a reportado por Morillo et al. (2011) [12], quienes evaluaron morfológica y molecularmente, con RAMs, 18 introducciones de uchuva P. peruviana de la colección de la Universidad de Nariño encontrando valores de loci polimórficos del 100% y una heterocigosidad estimada de 0,44. Otros estudios de diversidad y estructura genética en Uchuva colombiana como los realizados por Chacón et al. (2016) [4] usando marcadores SSRs, reportan también un valor de heterocigosidad bajo (He= 0,22) atribuido al tamaño de la muestra, el origen de las accesiones y el tipo de marcador molecular utilizado. Osorio (2016) [8] evaluó la diversidad genética en un grupo de 100 accesiones de uchuva procedentes de diferentes regiones de Colombia con más de 5000 SNPs y encontró valores de heterocigosidad casi tres veces más alta que en este estudio. Garzón et al. (2015) [9] usando marcadores COSII e IRGs en germoplasma colombiano encontraron valores de He= 0,30, manifestando la existencia de alta variabilidad genética debido a la naturaleza alógama de la especie que cuenta con 54% de polinización cruzada lo cual favorece el flujo genético y la aparición de nuevos recombinantes ciclo tras ciclo.
El coeficiente de diferenciación genética fue de 0,35 según Wright (1978) [16], valores iguales o mayores a 0,35 muestran una gran diferenciación genética, la cual está asociada con el nivel de estructuración y la dinámica espacio-temporal a la cual están sometidos los materiales de uchuva en su entorno natural. El Análisis de Varianza Molecular AMOVA, muestra que el 76% de la variabilidad genética observada en los materiales de uchuva colectados en el departamento de Boyacá es debida al componente dentro de grupos y que el 24% (Cuadro 4) restante se atribuye a las diferencias existentes entre los grupos considerados, lo cual indica que se deben hacer estudios microgeográficos que incluyan todo ese componente de variación y que se pueda usar efectivamente en la identificación de materiales adaptados, con buen rendimiento, color, sabor y con tolerancia a F. oxysporum, principal problema fitosanitario, que respondan a las necesidades del agricultor, productor y consumidor. Resultados similares han sido reportados por Osorio (2016) [8], mostrando que el grupo formado por los materiales silvestres es el que presenta mayor diferenciación y tienen un gran potencial para el mejoramiento genético. Chacón et al. (2016) [4] encontraron niveles de diferenciación significativos entre los materiales y las cordilleras de los Andes (FST = 0,174, P = 0,001) concluyendo que la diversidad genética está más asociada al componente geográfico que al biológico (silvestre o cultivado).
En Colombia la uchuva crece como una planta silvestre que está en un proceso de domesticación por lo cual más conocimiento y conservación de germoplasma nativo son necesarios para superar los actuales problemas agronómicos presentes en las principales regiones productoras. Los diferentes estudios de diversidad y estructura genética de germoplasma de P. peruviana y otras especies relacionadas usando diferentes herramientas biotecnológicas (marcados moleculares, secuenciación, bioinformática) han mostrado la existencia de variabilidad genética asociada tanto al estado biológico del material como al sitio geográfico de donde proceden los materiales. Lo anterior sugiere que en el germoplasma Colombiano de uchuva hay potencial para muchas características de interés comercial, agronómico y nutricional que todavía no ha sido explorado.
CONCLUSIONES
Se determinó la existencia de una variabilidad genética importante en los materiales de uchuva colectados, en el departamento de Boyacá, a pesar de que el número es bajo se encontró que ésta está principalmente influenciada por el sitio geográfico y a nivel intraespecífico lo cual sugiere niveles de jerarquización y subdivisión mayores a los considerados en este estudio. Los agrupamientos en este estudio se presentaron de acuerdo al origen geográfico de donde proceden los materiales y no por su estado biológico como había sido reportado en investigaciones en Physalis. Hay potencial en los materiales que debe ser conservado y aprovechado en el planteamiento de estrategias que conduzcan al mejoramiento genético de esta especie.