Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Química
Print version ISSN 0120-2804On-line version ISSN 2357-3791
Rev.Colomb.Quim. vol.37 no.1 Bogotá Jan./Apr. 2008
CARACTERIZACIÓN DEL TALLO ACEPTOR DEL tARN MEDIANTE DESCRIPTORES LOCALES BASADOS EN CARGAS PARCIALES
tRNA ACCEPTOR STEM CHARACTERIZATION BY MEANS OF LOCAL DESCRIPTORS BASED ON PARTIAL CHARGES
CARACTERIZAÇÃO DO TALO ACEITADOR DO tRNA MEDIANTE DESCRITORES LOCAIS BASEADOS EM CARGAS PARCIAIS
M. Marín Ray1, Édgar E. Daza1
1 Departamento de Química, Facultad de Ciencias, Universidad Nacional de Colombia, sede Bogotá, Bogotá, D. C., Colombia. eedazac@unal.edu.co
Recibido: 04/12/07 – Aceptado: 15/04/08
RESUMEN
En este trabajo se caracteriza la distribución de carga del tallo aceptor del tARN, considerando todas las posibles combinaciones de pares Watson-Crick. El estudio se realizó con 256 fragmentos moleculares de 10 nucleótidos que modelan los tres primeros pares del tallo aceptor, la base diferenciadora y el extremo CCA. Para caracterizar los nucleótidos se proponen dos descriptores locales basados en la distribución de carga de la base nitrogenada de cada nucleótido, los cuales se calculan a partir de las cargas parciales de Mulliken obtenidas de cálculos HF/6-31G. La caracterización y clasificación de los tallos según estos descriptores mostró cómo la base diferenciadora tiene un comportamiento particular respecto a los demás nucleótidos del tallo y una fuerte influencia sobre el extremo CCA. La clasificación de nueve variaciones del tallo aceptor del tARNAla mostró una buena relación estructura-actividad que pone en evidencia la bondad de los descriptores propuestos para caracterizar de manera local la distribución de carga de estas biomoléculas.
Palabras clave: tallo aceptor del tARN, base diferenciadora, aminoacilación, elementos de identidad, distribución de carga, cargas parciales, descriptores locales de carga.
ABSTRACT
In this work the charge distribution of the tRNA acceptor stem is characterized, considering all the possible Watson- Crick base pair combinations. 256 RNA fragments modeled by 10 nucleotides were used in order to model the first three pairs of the acceptor stem, the discriminator base and the CCA end. We propose two local charge descriptors based on the charge distribution of the nitrogenated base to characterize each nucleotide. These descriptors were computed from atomic partial charges derived from HF/6-31G calculations. From the characterization and classification of the stems according to the proposed descriptors, we found a special behavior for the discriminator base (in contrast to the other positions) and a strong effect of this position on the CCA end. The classification of nine variations of the tRNAAla acceptor stem showed a good structure-activity relationship that makes evident the usefulness of the proposed descriptors to characterize the local charge distributions of these biomolecules.
Key words: tRNA acceptor stem, discriminator base, aminoacylation, identity elements, charge distribution, partial charges, local charge descriptors.
RESUMO
Nesse estudo é caracterizada a distribuição da carga do talo aceitador considerando- se todas as combinações possíveis dos pares Watson-Crick. O estudo realizouse com 256 fragmentos moleculares dos 10 nucleotídeos que modelam os três primeiros pares do talo aceitador, a base diferenciadora e o extremo CCA. Com o intuito de caracterizar cada nucleotídeo, foram propostos dois descritores locais baseados na distribuição de carga da base nitrogenada de cada nucleotídeo, os quais se calculam a partir das cargas parciais de Mulliken obtidas de cálculos HF/6-31G. A caracterização e classificação dos talos segundo esses descritores demonstrou um particular comportamento da base diferenciadora em relação aos demais nucleotídeos do talo e uma forte influência sobre o extremo CCA. A classificação de nove variações do talo aceitador do tRNA mostrou uma boa relação estrutura-atividade que colocam em evidência a utilidade dos descritores propostos para caracterizar de maneira local a distribuição de carga dessas bio-moléculas.
Palavras-chave: Talo aceitador tRNA, base diferenciadora, aminoacilação, elementos de identidade, distribuição de carga, cargas parciais, descritores locais de carga.
INTRODUCCIÓN
Es bien conocido que la tripleta del anticodón no es la única parte del tARN responsable del reconocimiento entre la aminoacil- tARN sintetasa (aaRS) y el tARN necesario para el proceso de aminoacilación. De hecho, hay otras partes de la molécula involucradas, las cuales se conocen como elementos de identidad. Estos elementos son un conjunto de nucleótidos ubicados sobre los brazos del anticodón y aceptor que se han establecido como fundamentales para expresar la identidad química de los tARN a la enzima (1-3). De particular importancia resulta el nucleótido de la posición 73 (el primero desapareado en el extremo 3’), que aparece como elemento de identidad en la mayoría de los sistemas de aminoacilación, y se conoce como base diferenciadora (1, 4).
Se han hecho diversos estudios experimentales, así como simulaciones empleando dinámica molecular, para establecer cómo se afecta el tallo aceptor por modificaciones sobre sus elementos de identidad. Así, por ejemplo, se han evaluado algunos parámetros geométricos de la doble hélice (5-9), las interacciones de apilamiento (10-12), la disposición del extremo CCA (12-15) y los procesos de hidratación (7, 16). En algunos casos se han podido establecer algunas correlaciones entre la disposición espacial de la base diferenciadora y la actividad de aminoacilación (10).
Para comprender más detalladamente las bases moleculares del proceso de reconocimiento aaRS-tARN, recientemente se han empleado diversas aproximaciones al Potencial Electrostático Molecular (PEM) para caracterizar la aaRS y el tARN o fragmentos del mismo (17-20). En estos estudios se ha encontrado que la distribución de carga, caracterizada mediante el PEM, determina en gran medida un reconocimiento inicial de largo alcance entre la enzima y el tARN (17, 18). También se ha encontrado que las propiedades electrostáticas del tallo aceptor del tARNAla pueden ser más determinantes en el reconocimiento que las variaciones geométricas que pueda inducir un cierto par de bases (19).
Motivados por las buenas correlaciones establecidas a partir de propiedades electrostáticas (17-19) y que, hasta donde sabemos, no existe un estudio sistemático de todos los posibles tallos aceptores con sus respectivas bases diferenciadoras, en este trabajo monitoreamos los cambios en la distribución de carga del tallo aceptor de dos conjuntos de tARNs. De una parte consideramos el tallo aceptor del tARNAla y ocho variaciones del mismo, las cuales han sido estudiadas experimentalmente, y de otra parte el total de los 256 tallos aceptores que se pueden construir empleando los pares Watson-Crick (WC) estándar. Para comparar y clasificar los elementos de cada uno de los dos conjuntos hemos propuesto algunos descriptores locales establecidos a partir de los resultados de cálculos cuánticos realizados sobre tallos aceptores modelo, cada uno compuesto por diez nucleótidos.
Los resultados se presentan en tres partes. En primer lugar, mostramos una relación estructura-actividad (SAR por sus siglas en inglés) para nueve variaciones del tallo aceptor del tARNAla cuya actividad ha sido reportada. En segundo lugar, analizamos el poder discriminante de los descriptores propuestos para cada una de las posiciones del tallo aceptor, y cómo los descriptores corroboran un comportamiento particular para la base diferenciadora y, por último, presentamos un análisis de agrupamiento que muestra una fuerte influencia de la base diferenciadora sobre el extremo CCA.
MÉTODOS
Los sistemas moleculares
El tallo aceptor de los tARN se modeló mediante motivos estructurales compuestos por 10 nucleótidos (Figura 1). Para el conjunto de 256 tARNs, los nucleótidos en las posiciones 1-6 se escogieron de manera que se tuviesen todos los posibles arreglos con pares WC (A-U, U-A, G-C, y C-G), para la posición 7, la correspondiente a la base diferenciadora, se consideraron los cuatro ribonucleótidos (A, C, G y U), y en las posiciones 8-10 se dejó la secuencia constante CCA típica de los tARNs. Para el conjunto de tARNAla todos los tallos presentan los pares 1G-C6, 2G-C5 y 3G-U4, correspondientes a la secuencia nativa, para la posición 7 se mutó la base diferenciadora nativa A, por cada una de las otras tres bases nitrogenadas estándar y por cinco bases modificadas: 2AA, 2AP, Neb, 7DAA y I; en el trabajo de Fischer y cols. (11) se puede encontrar la fórmula estructural de las nueve bases empleadas.
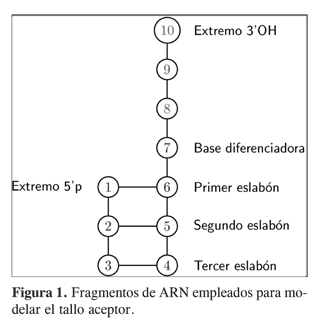
Para la modelación de las geometrías nucleares se tomaron posiciones estándar para tARNs en disposición A, azúcar 3-endo, y como grupos de cierre usamos PO4 3- para las posiciones 5’ y OH- para las 3’. El esqueleto fosfodiester, lo mismo que las bases modificadas, fueron optimizados con mecánica molecular empleando el campo de fuerzas Amber (21). Con geometrías establecidas de esta manera realizamos cálculos cuánticos ab initio HF/6-31G con el programa Gaussian 98 (22). Para monitorear el comportamiento de la distribución de cargas electrónica empleamos las cargas parciales de Mulliken.
Descriptores locales
Un problema esencialmente abierto en química es comparar moléculas de manera cuantitativa (23, 24), éste se hace aún más complejo cuando el sistema molecular es de gran tamaño. Además, desde el punto de vista cuántico, la comparación de cantidades fundamentales como la densidad de carga o el potencial electrostático, e incluso el análisis de la función de onda, en términos de orbitales, se hace casi impracticable cuando se quieren considerar grandes conjuntos de moléculas (24). De manera que es necesario proponer descriptores de fácil cálculo y comparación. De hecho es deseable, para el caso de biopolímeros como los ácidos nucleicos y las proteínas, poder tener caracterizaciones locales (por aminoácido o por nucleótido) con el fin de comparar y explicar distintos comportamientos debidos a cambios puntuales en sus estructuras primarias. En este trabajo presentamos dos descriptores basados en las cargas parciales de Mulliken para caracterizar, comparar y clasificar los tallos aceptores del tARN:


donde b es el total de los átomos de la base nitrogenada de un nucleótido dado.
Hemos escogido las cargas de la base en lugar de las del nucleótido porque están menos influenciadas por los cierres empleados para los extremos 5’ y 3’ (posiciones 1, 3, 4 y 10) los cuales hacen que la carga para algunos átomos tome valores extremos, y la escala sea por ende menos discriminante. El primer descriptor (Q) asigna una carga a cada base, por lo que se puede considerar como la carga de la base; el segundo (D) da una idea de la dispersión de las cargas parciales de cada base y podría de manera muy aproximada dar una idea de su polarización, pues valores muy altos indican la presencia de cargas muy positivas y muy negativas, y por tanto, de enlaces muy polarizados.
Los descriptores propuestos se ensayaron primero en un estudio tipo SAR con el conjunto de nueve tallos asociados al tARN de alanita, y luego la distribución de sus valores se estudió para el conjunto de 256 posibles tallos W-C en función de la posición en el tallo aceptor. Para ello empleamos diagramas de cajas y bigotes (25), en los que se indican los valores de los cuartiles primero y tercero, la mediana y la media. Los extremos de los bigotes fueron definidos de manera estándar ±1,5 X RIC (RIC: Rango Intercuartil) a partir del tercer y primer cuartil, respectivamente.
RESULTADOS Y DISCUSIÓN
Una aplicación SAR
Hemos comparado los fragmentos moleculares que representan el tallo aceptor del tARNAla y ocho variaciones de éste, en las cuales sólo se han hecho cambios en la base diferenciadora. Para cada uno de estos tallos se ha reportado la actividad de aminoacilación in vitro (kcat/KM), la cual se encuentra normalizada respecto al tallo nativo, cuyo valor es 1,0. Los valores de actividad relativos son los siguientes: 1,3 (2AA); 1,0 (A); 0,67 (2AP); 0,48 (Neb); 0,070 (7DAA); 0,029 (C); 0,015 (U); 0,0097 (G); 0,0049 (I) . Los valores de actividad se presentan con las cifras significativas con que los presentó el respectivo autor (11).
Empleando los valores de los descriptores de cada posición como variables para caracterizar cada uno de los sistemas (D1, D2,…, D10, Q1 , Q2,…,Q10) hicimos un análisis de agrupamiento empleando el método de agrupamiento sencillo y la métrica de Pearson, esta medida métrica la hemos convertido en una medida de similitud normalizada de acuerdo con:
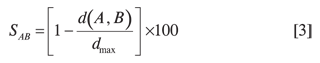
donde: d(A,B) es la distancia métrica entre los tallos A y B, y dmax es la distancia máxima entre un par de tallos cualquiera en todo el conjunto de estudio. Los resultados se muestran en el dendrograma (Figura 2).

En el dendrograma se puede observar cómo el tallo nativo se asocia con el de la base modificada 2AA, la cual presenta la mayor actividad (130%). A la rama definida por estos dos tallos se une otra que agrupa a las bases con actividades del 67 y 48% de la nativa: 2AP y Neb respectivamente. A ellas se agrega la base con la siguiente actividad 7DAA (7%). En una rama aparte se unen las dos con menor actividad G (0,97%) e I (0,49%). Por último, se agregan al dendrograma los tallos de las bases C y U, que tienen actividades de 2,9 y 1,5% respectivamente. Es claro que en el dendrograma se pueden identificar dos zonas importantes: la zona de la izquierda en donde se agrupan los cinco tallos con la actividad más alta, y la zona de la derecha donde se encuentran los tallos con las actividades más bajas. Si el agrupamiento se hace con el conjunto de los descriptores por separado, la clasificación es igualmente buena, como puede observarse en las Figuras 3 y 4. La calidad de estos agrupamientos la corroboramos mediante el método de selección de nivel óptimo de agrupamiento Point Biseral (29), el cual arrojó como niveles de corte los siguientes porcentajes de similitud: 47,52% (Pointbis=0,6282) para el primero; 70,48% (Pointbis=0,6360) para el segundo, y para el tercero 45,78% (Pointbis=0,7528). Entre paréntesis anotamos el valor óptimo del parámetro que se emplea en esta prueba.

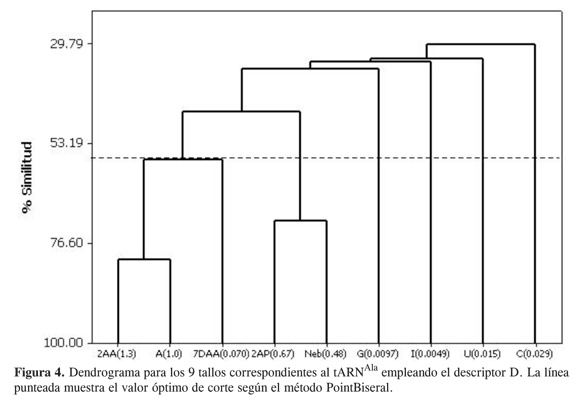
Como puede verse, los descriptores propuestos recogen de buena manera algunas de las características moleculares de estos tallos, al punto de poder correlacionar la descripción que de ellos se logra con la actividad de aminoacilación reportada experimentalmente, de manera que parecen caracterizar apropiadamente los tallos aceptores del tARN, por lo que los emplearemos para establecer clasificaciones y tendencias en el conjunto de los 256 posibles tallos con bases W-C.
Distribución de los descriptores
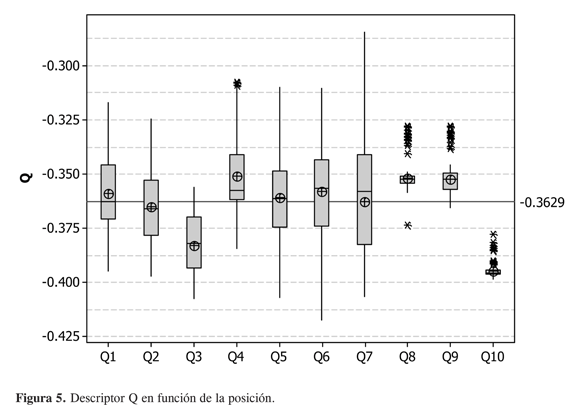
Antes de presentar la clasificación que logramos para el conjunto de 256 tallos presentaremos un análisis de la distribución de sus valores para este conjunto. En la Figura 5 se muestra la distribución de la carga de la base (Q) para cada una de las posiciones del tallo. Puede apreciarse cómo la posición que presenta mayor dispersión en los datos, y por ende mayor variabilidad es la 7, toda vez que el RIC es el mayor de todos (RIC=0,04), seguido por el de la posición 6 (RIC=0,03). Los bigotes para la posición 7 alcanzan el valor más alto (aprox. -2,9), y el límite inferior es sólo superado por el de la posición 6. La distribución de los datos correspondientes a las posiciones 1 y 4 muestra cierta desviación positiva debida al grupo PO43-, empleado como cierre, cuya electronegatividad hace que en conjunto la carga de la base sea menos negativa para más estructuras (26). Resulta interesante que la posición 4 muestra cinco outliers, cuatro de los cuales corresponden a la secuencia C4C5C6X7, donde X es cualquiera de las cuatro bases diferenciadoras. Esto se debe posiblemente a que en los pares G-C la citosina se comporta como un extremo electropositivo debido al grupo amino expuesto hacia el surco mayor. Este efecto se ve reforzado cuando varios pares G-C se encuentran apilados, lo cual podría explicar la presencia de estos casos aislados cuya carga es la menos negativa para esta posición (27). A su vez, las bases de los nucleótidos en los extremos 3’ (posiciones 3 y 10) presentan los valores promedios más bajos, así el 75% de las bases en 3 y todas las adeninas de la posición 10 tienen valores inferiores a la media global. Esto último refleja el efecto del OH- empleado como cierre (). En la misma Figura 5 es posible observar que el extremo constante CCA muestra muy poca variabilidad; llama la atención que la citosina C9 tenga un rango intercuartil mayor que la C8 que debería, en razón de su vecino cambiante –la base diferenciadora– mostrar mayor variabilidad. Para el extremo CCA hay una presencia relativamente grande de outliers con desviación positiva, la mayoría de los cuales tienen en común un par A-U o U-A en las posiciones 1-6.
La Figura 6, correspondiente al descriptor D, muestra mayor similitud en el comportamiento para las posiciones 1 a 7, todas con rangos intercuartiles similares y una desviación negativa. También se observa una reducción muy fuerte del rango intercuartil para la posición 8, y un rango mínimo para 9 y 10. Para este descriptor el comportamiento del extremo CCA sí refleja el efecto de la variación en la posición 7, el cual va decreciendo entre las posiciones 8 a 10, en contraste con el descriptor anterior. Una manera de interpretar este efecto es considerar el extremo CCA como una extensión que reproduce con menor intensidad el efecto de la variación en la base diferenciadora, es decir, el comportamiento electrostático del extremo CCA, al verse afectado sólo por los cambios en dicha posición, actúa como una extensión de la posición 7.

Al hacer la gráfica de cajas para el descriptor D considerando cada una de las cuatro bases por separado, sin importar la posición en que se encuentren en el tallo, encontramos un comportamiento interesante (Figura 7). De una parte, si bien en todos los casos los rangos intercuartiles son de nuevo muy similares, notamos la presencia de outliers para cada base. Para la adenina se presentan 61 outliers suaves (entre ± 1,5 X RIC y 3,0 X RIC), todos correspondientes a adeninas en la posición 7 (la de la base diferenciadora). Ante este comportamiento buscamos la localización de las tres moléculas restantes con adenina en la posición 7, y encontramos que se hallan en la parte inferior del bigote. En el caso de la citosina el comportamiento de este descriptor es aún más contundente que en la adenina, pues las 64 estructuras que tienen C en la posición 7 se muestran como outliers. Para la guanina sólo se presentan 11 outliers y, al igual que en el primer caso, al buscar las otras moléculas con base diferenciadora G, las encontramos muy cerca del extremo del bigote inferior. Los valores de D para el uracilo tienen un comportamiento distinto; ahora los outliers (32 en total) se encuentran por encima de la caja y no son, como en los casos anteriores, correspondientes a la posición 7 sino a uracilos en las posiciones 5 y 6. Cuando buscamos la distribución de los uracilos en la posición 7, encontramos que éstos se hallan concentrados fundamentalmente en la región definida por el bigote inferior, con 16 excepciones que se hallan sobre el extremo superior de la caja (entre el segundo y tercer cuartil).

Podemos así concluir que el descriptor D, cuando se analiza para cada base, permite diferenciar la posición 7 como una posición en el tallo con características particulares, ya que para A, C y G esta posición aparece como outlier o por debajo del primer cuartil (25%) de moléculas con el valor más bajo, sin que se presenten outliers extremos (esto es, 3,0 X RIC por debajo del primer cuartil). Sólo para U se presentan algunas excepciones. Esto resulta acorde con el tipo de interacciones al que están sometidas las bases en las distintas posiciones del tallo aceptor. Debido a que la posición 7 es la única que se encuentra desapareada, es de esperarse que presente enlaces menos polarizados (valores más bajos de D), ya que aquellas bases que se encuentran apareadas deben presentar grupos altamente cargados (lo cual se refleja en enlaces más polarizados) para poder establecer las interacciones por puentes de hidrógeno.
Clasificación de los 256 tallos aceptores
A partir de la distribución que presentan los dos descriptores propuestos podemos deducir que el descriptor D resulta más apropiado para intentar una clasificación de los 256 tallos según su base diferenciadora. Para ello, empleando como descripción de cada tallo el vector formado por los valores de D para cada posición (D1,D2,…,D10), hicimos un agrupamiento empleando también el método de agrupamiento simple y la métrica de Pearson para establecer la relación entre cada par tallos (28). Al igual que en el trabajo de Marín y cols. (24), para la interpretación de los dendrogramas convertimos la medida métrica en una medida de similitud (ver ecuación. 3) Los resultados de este agrupamiento se pueden ver en la Figura 8.
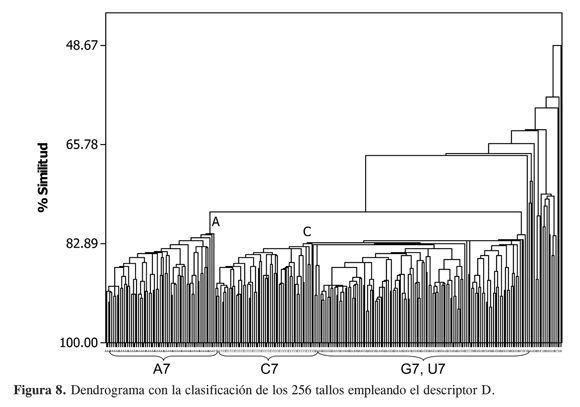
Allí se observa cómo los tallos con base diferenciadora adenina se hallan agrupados mayoritariamente en una rama, lo mismo ocurre con la C. Los otros dos nucleótidos, G y U, resultan mezclados. Cabe destacar que a pesar de estar empleando los descriptores de las 10 posiciones, la clasificación refleja la presencia de la base en la posición 7, i.e. la base diferenciadora. Esto indica que buena parte de la identidad del tallo aceptor está determinada por la base diferenciadora lo cual justifica, desde el punto de vista estructural, su importancia como elemento de identidad según se ha establecido experimentalmente, y que un cambio en ella resulte tan drástico en la actividad de aminoacilación, como sucede por ejemplo para Escherichia coli (1). El comportamiento de los descriptores para el extremo terminal CCA permite pensar que si bien el extremo es constante, alcanza a amplificar la identidad de la base diferenciadora, como puede inferirse de la distribución que se observa en las Figuras 2 y 3. La importancia del extremo CCA como extensión de la identidad de la base diferenciadora se hace aún más evidente al hacer el mismo análisis de agrupamiento pero sin considerar las posiciones 8-10, es decir, (D1,D2,…,D7). En este caso se pierde cualquier tipo de discriminación entre los tallos con distinta base diferenciadora (información no mostrada).
CONCLUSIONES
Con el fin de caracterizar la distribución de carga del tallo aceptor del tARN, hemos definido dos descriptores locales construidos a partir de las cargas parciales de Mulliken de los átomos que conforman las bases nitrogenadas. Estos descriptores locales nos permiten representar cada tallo por medio de una variable asociada a cada uno de los nucleótidos que lo conforman. Al hacer una caracterización de los 256 tallos obtenidos de todas las posibles combinaciones de pares Watson-Crick, y probando las cuatro bases en la posición 7, logramos establecer que la base diferenciadora es una posición especial desde el punto de vista de la distribución de carga por dos razones principalmente: por un lado, al ser una posición desapareada presenta una distribución de valores distinta a la de las otras seis posiciones para los dos descriptores debido a que no está sometida a establecer interacciones por puentes de hidrógeno; y por otro, al ser la posición más cercana al extremo CCA, influencia completamente la distribución de carga de estos nucleótidos, convirtiéndolos en una extensión de los cambios de la base diferenciadora.
Además, al clasificar nueve tallos aceptores correspondientes al tARNAla nativo, y ocho variaciones, se observó que los tallos que presentan una actividad de aminoacilación más alta quedan agrupados en una rama del dendrograma, mostrando así la bondad de los descriptores propuestos como descriptores locales útiles para caracterizar la distribución de carga de estas biomoléculas.
AGRADECIMIENTOS
Los autores expresan su agradecimiento a Colciencias por el apoyo económico brindado para el desarrollo de este trabajo.
REFERENCIAS BIBLIOGRÁFICAS
1. Giege, R.; Sissler, M.; Florentz, C. Universal rules and idiosyncratic features in tRNA identity. Nucleic Acids Res. 1998. 26: 5017-5035. [ Links ]
2. Fender, A.; Sissler, M; Florentz, C.; Giegé, R. Functional idiosyncrasies of tRNA isoacceptors in cognate and noncognate aminoacylation systems. Biochemie. 2004. 86: 21-29. [ Links ]
3. Giegé, R.; Florentz, C.; Kern, D.; Gangloff, J.; Eriani, G.; Moras, D. Aspartate identity of transfer RNAs. Biochimie.1996. 78: 605-623. [ Links ]
4. Crothers, D. M.; Seno, T.; Söll, D. G. Is there a discriminator site in transfer RNA? Proc. Natl. Acad. Sci. USA. 1972. 69: 3063-3067. [ Links ]
5. Alexander, R. W.; Nordin, B. E.; Schimmel, P. Activation of microhelix charging by localized helix destabilization. Proc. Natl. Acad. Sci. USA. 1998. 95: 12214-12219. [ Links ]
6. Ramos, A.; Varani, G. Structure of the acceptor stem of Escherichia coli tRNAAla role of the G3·U70 base pair in synthetase recognition. Nucleic Acids Res. 1997. 25: 2083-2090. [ Links ]
7. Mueller, U.; Schübel, H.; Sprinzl, M.; Heinemann, U. Crystal structure of acceptor stem of tRNAAla from Escherichia coli shows unique G·U wobble base pair at 1,16Å resolution. RNA. 1999. 5: 670-677. [ Links ]
8. Limmer, S.; Reif, B.; Ott, G.; Arnold, L.; Sprinzl, M. NMR evidence for helix geometry modifications by a G-U wobble base pair in the acceptor arm of E. coli tRNAAla. FEBS Lett. 1996. 385: 15-20. [ Links ]
9. Vogtherr, M.; Schübel, H.; Limmer, S. Structural and dynamic helix geometry alterations induced by mismatch base pairs in double-helical RNA. FEBS Lett. 1998. 429: 21-26. [ Links ]
10. Nagan, M. C.; Beuning, P. J.; Musier- Forsyth, K.; Cramer, C. J. Importante of discriminator base stacking interactions: molecular dynamics analysis of A73 microhelixAla variants. Nucleic Acids Res. 2000. 28: 2527-2534. [ Links ]
11. Fischer, A. E.; Beuning, P. J.; Musier- Forsyth, K. Identification of iscriminator base atomic groups that modulate the alanine aminoacylation reaction. J. Biol. Chem. 1999. 74: 37093-37096. [ Links ]
12. Metzger, A. U.; Heckl, M.; Willbold, D.; Breitschopf, K.; RajBhandary, U. K.; Rösch, P; Gross, H. J. Structural atudies on tRNA acceptor stem microhelices: exchange of discriminator base A73 for G in human tRNALeu switches the acceptor specificity from leucine to serine possibly by decreasing the stability of the terminal G1-C72 base pair. Nucleic Acids Res. 1997. 25: 4551-4556. [ Links ]
13. Seetharaman, M.; Williams, C.; Cramer, C. J.; Musier-Forsyth, K. Effect of G-1 on histidine tRNA microhelix conformation. Nucleic Acids Res. 2003. 31: 7311-7321. [ Links ]
14. Puglisi, E. V.; Puglisi, J. D.; Williamson, J. R.; Rajbhandary, U. L. NMR analysis of tRNA acceptor stem microhelices: Discriminator base change affects tRNA conformation at the 3' end. Proc. Natl. Acad. Sci. USA. 1994. 91: 11467-11471. [ Links ]
15. Limmer, S.; Hofmann, H. P.; Ott, G.; Sprinzl, M. The 3'-terminal end (NCCA) of tRNA determines the structure and stability of the aminoacyl acceptor stem. Proc. Natl. Acad. Sci. USA. 1993. 90: 6199-6202. [ Links ]
16. Nagan, M. C.; Kerimo, S. S.; Musier- Forsyth, K; Cramer, C. J. Wild-type RNA microhelixAla and 3:70 variants: molecular dynamics analysis of local helical structure and tightly bound water. J. Am. Chem. Soc. 1999. 121: 7310-7317. [ Links ]
17. Tworowski, D.; Safro, M. The long-range electrostatic interactions control tRNA-aminoacyl-tRNA synthetase complex formation. Protein Sci. 2003. 12: 1247-1251. [ Links ]
18. Tworowski, D.; Feldman, A. V.; Safro, M. G. Electrostatic potential of aminoacyl-tRNA synthetase navigates tRNA on its pathway to the binding site. J. Mol. Biol. 2005. 350: 866-882. [ Links ]
19. Beuning, P. J.; Nagan, M. C.; Cramer, C. J.; Musier-Forsyth, K.; Gelpi, J.-L.; Bashford, D. Efficient aminoacylation of the tRNAAla acceptor stem: dependence on the 2:71 base pair. RNA. 2002. 8: 659-670. [ Links ]
20. McDowell, J. A.; Turner, D. H. Investigation of the structural basis for thermodynamic stabilities of tandem GU mismatches: solution structure of (rGAGGUCUC)2 by two-dimensional NMR and simulated annealing. Biochemistry. 1996. 35: 14077-14089. [ Links ]
21. Cornell, W. D.; Cieplak, P.; Bayly, C. I.; Gould, I. R.; Merz, K. M.; Ferguson, D. M.; Spellmeyer, D. C.; Fox, T.; Caldwell, J. W.; Kollman, P. A. A second generation force field for the simulation of proteins, nucleic acids and organic molecules. J. Am. Chem. Soc. 1995. 117: 5179-5197. [ Links ]
22. Frisch, M. J. et al., Gaussian 98 Rev. A 11. Gaussian Inc., Pittsburgh PA. 2001. [ Links ]
23. Bultinck, P.; Gironés, X.; Carbó- Dorca, R. Molecular quantum similarity: Theory and applications. En Reviews in Computational Chemistry. New York: John Wiley and Sons, Inc. Publishers. 2005. Volume 21, pp. 127-207. [ Links ]
24. Marín, R. M.; Aguirre, N. F.; Daza, E. E. A graph theoretical similarity approach to compare molecular electrostatic potentials. J. Chem. Inf. Model. 2008. 48: 109-118. [ Links ]
25. Tukey, J. W. Exploratory Data Analysis. Reading, MA; Addison- Wesley. 1977. [ Links ]
26. Marín, R. M. Elementos de identidad del brazo aceptor del tRNA. Una clasificación estructural. Trabajo de pregrado. Departamento de Química, Facultad de Ciencias, Universidad Nacional de Colombia. 2004. p. 24. [ Links ]
27. Marín, R. M. Caracterización de superficies de potencial electrostático molecular (SPEM) para fragmentos del tallo aceptor del tRNAAla. Tesis de Maestría en Ciencias-Química. Departamento de Química, Facultad de Ciencias, Universidad Nacional de Colombia. 2007. p. 37. [ Links ]
28. Johnson, R. A.; Wichern, D. W. Applied Multivariate Statistical Analysis. Upper Saddle River, New Jersey: Prentice Hall. 2002. p. 668. [ Links ]
29. Wild, D. J.; Blankley, C. J. Comparison of 2D Fingerprint Types and Hierarchy Level Selection Methods for Structural Grouping Using Ward's Clustering. J. Chem. Inf. Comput. Sci. 2000; 40: 155-162. [ Links ]














