Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Biológica Colombiana
Print version ISSN 0120-548X
Acta biol.Colomb. vol.12 suppl.1 Bogotá Dec. 2007
DISEÑO DE OLIGONUCLEÓTIDOS PARA EL ESTUDIO DE GENES CELULOLÍTICOS Y SOLVENTOGÉNICOS EN CEPAS COLOMBIANAS DE Clostridiumsp. (CLOSTRIDIACEAE)
Oligonucleotide Probe Design for the Study of Cellulolytic and Solventogenic Genes in Colombian Clostridiumsp. strains (Clostridiaceae)
JOSÉ DAVID MONTOYA SOLANO1, Químico Farmacéutico; ZULMA ROCÍO SUÁREZ MORENO2, M.Sc.; DIEGO MAURICIO RIAÑO PACHÓN3, Biólogo; DOLLY MONTOYA CASTAÑO1, M.Sc., Dr. rer. nat.; FABIO ANCÍZAR ARISTIZÁBAL GUTIÉRREZ4, Ph. D.
1Grupo de Bioprocesos y Bioprospección, Instituto de Biotecnología, Universidad Nacional de Colombia, Sede Bogotá, ciudad universitaria, Carrera 30 No. 4503. AA. 14490. Teléfono: 571 316 5000, exts. 16971 16954. jdmontoyas@unal.edu.co dmontoyac@unal.edu.co
2Grupo de Bacteriología y Bacteriología Vegetal, Laboratorios ICGEB Trieste, Italia. Teléfono: +3940375 73 17. suarez@icgeb.org
3Departamento de Biología Molecular, Universidad de Potsdam. Potsdam, Alemania. Teléfono: +493319772809. diriano@rz.unipotsdam.de
4Departamento de Farmacia, Universidad Nacional de Colombia, Sede Bogotá, ciudad universitaria, Carrera 30 No. 4503. AA. 14490. Telefono: 571 316 50 00, ext. 14643. faaristizabalg@unal.edu.co
Presentado el 2 de octubre de 2006, aceptado el 16 de mayo de 2007, correcciones 3 de octubre de 2007.
RESUMEN
El objetivo de este estudio fue analizar las rutas metabólicas para la producción de solventes y degradación de celulosa en cepas colombianas promisorias del género Clostridium. Para ello se diseñaron sondas de hibridación que sirvieran para posteriores estudios de mejoramiento genético de las cepas. Se construyó la base de datos denominada MULTICLOST en Microsoft Access® con las secuencias de 485 genes involucrados en las rutas metabólicas arriba mencionadas, provenientes de 45 especies bacterianas y 10 especies fúngicas. Los genes fueron agrupados de acuerdo al tipo de enzima y a los dominios catalíticos o de unión a sustrato en el caso de las celulasas. Cada grupo se sometió a alineamiento múltiple en ClustalW 1.83 y con base en los resultados se crearon subgrupos de similitud mayor al 50%. Se localizaron secuencias conservadas de longitud mayor a 19 nucleótidos en GeneDoc 2.6.002 y sus valores termodinámicos fueron estimados con GeneRunner v3.05, mientras que la sensibilidad y especificidad fue verificada por búsquedas en GenBank usando BLASTN 2.2.8. En total se obtuvieron 94 secuencias conservadas con las siguientes características: longitud promedio de 24 nucleótidos, Tm promedio de 65,8 ºC y contenido de (G+C) entre 14,3 y 60,0%. Se determinó que ninguna de las sondas diseñadas forma estructuras secundarias estables con Tm superior a 36,1 ºC. De acuerdo a sus características y valores termodinámicos, todas las sondas podrían ser utilizadas en la construcción de un microarreglo o en reacciones de PCR para la identificación de regiones relevantes en el mejoramiento del proceso por ingeniería metabólica.
Palabras clave: sondas de oligonucleótidos, Clostridium sp., Microarreglos, Bioinformática.
ABSTRACT
The goal of the present study was to analyze the metabolic pathways involved in solvent production and cellulose consumption by promising Colombian native strains of the genus Clostridium. Therefore a set of oligonucleotide probes was designed, with the aim of analyzing potential targets for genetic improvement of the Colombian strains. The database named MULTICLOST was created in Microsoft Access® using the sequences from 485 genes involved in solventogenesis, 1,3propanodiol production and cellulolysis from 45 bacterial and 10 fungal species. The genes were grouped according to their respective enzyme function and to the catalytic domain or the substrate binding domain in the case of cellulases. ClustalW 1.83 was used for multiple alignment of every group. Subgroups of sequences with more than 50% identity among themselves were created. Conserved sequences longer than 19 nucleotides were identified using GeneDoc 2.6.002 and their thermodynamic values were calculated with GeneRunner v3.05, while their sensitivity and specificity were verified by searching in GenBank with BLASTN 2.2.8. Ninetyfour conserved sequences were obtained with an average 24nucleotide length, 65.8ºC average Tm and a (G+C) content between 14.3% and 60.0%. None of these probes forms stable secondary structures at temperatures higher than 36.1ºC. According to the former results, all of the probes could be used in an oligonucleotide microarray or in PCR reactions for the identification of metabolic targets for improvement of the industrial process.
Key words: Oligonucleotide probes, Clostridium sp., Microarrays, Bioinformatics.
INTRODUCCIÓN
Este trabajo parte de la necesidad de una herramienta molecular adecuada para la detección de genes involucrados en los procesos de solventogénesis y celulolisis en cepas colombianas promisorias del género Clostridium. Dicho género incluye especies solventogénicas y no patógenas empleadas extensamente desde principios del siglo XX para la producción de acetona, butanol y etanol (Jones et al., 1986). Algunas de estas especies tienen la capacidad de utilizar sustratos celulósicos para la fermentación acetobutílica, los cuales reducen significativamente el costo total del proceso. Tradicionalmente la rentabilidad industrial de la biosíntesis de estos solventes y la degradación de celulosa depende de los rendimientos de la cepa y del costo de la fuente de carbono (Lynd et al., 2002; Pachauri et al., 2006). En el ámbito internacional se han patentado varios procesos consolidados para la producción biotecnológica de solventes con cepas bacterianas transformadas, alcanzando altos rendimientos por medio del uso de sustratos económicos y enzimas de alta eficiencia (Nakamura et al., 2003; Zverlov et al., 2006).
El Instituto de Biotecnología de la Universidad Nacional de Colombia (IBUN) ha trabajado en el empleo de residuos agroindustriales y en la búsqueda de cepas nativas con rendimientos superiores al de las cepas patrón. En la década de 1990 se aislaron 178 cepas de Clostridium a partir de suelos colombianos con el ánimo de encontrar cepas hiperproductoras de solventes. En uno de los estudios realizados se seleccionaron 13 cepas teniendo como base la concentración de solventes totales acetona, butanol y etanol a partir de glucosa (Montoya et al., 2000). Estudios posteriores evidenciaron que las cepas nativas de Clostridium pueden degradar sustratos celulósicos como pectina, xilano, carboximetilcelulosa y celulosa microcristalina (Montoya et al., 2001), mientras que cinco de las cepas producen 1,3propanodiol (1,3PD) a partir de glicerol en mayor concentración que Clostridium butyricum DSM2478 (Cárdenas et al., 2006). Adicionalmente los análisis de variabilidad molecular por AFLP Amplified Fragment Lenght Polymorfism, PFGE Pulsed Field Gel Electrophoresise hibridización DNADNA sugieren que 10 de las cepas promisorias corresponderían a una nueva especie del género Clostridium filogenéticamente muy cercana a C. butyricum (Arévalo, 2001; Suárez, 2004; Jaimes et al., 2006). Este conjunto de resultados permite considerar a las cepas nativas como candidatas para el diseño de bioprocesos innovadores de producción de solventes a nivel industrial. Los rendimientos de las cepas promisorias podrían ser mejorados por ingeniería metabólica, lo cual requiere del conocimiento de los genes de las rutas metabólicas involucradas en los procesos de solventogénesis y celulolisis. Los primeros estudios genéticos de las cepas promisorias incluyen la construcción de una librería genómica a partir de la cepa IBUN 22A, en la cuál se encontraron ocho clones con actividades exoglucanasa, endoglucanasa y‚ glucosidasa (Vargas et al., 2002). Estudios posteriores permitieron predecir genes homólogos a dhaB1 y dhaT de C. butyricum en las cepas nativas IBUN 22A, IBUN 13A e IBUN 158B; dichos genes codifican para las proteínas Glicerol Deshidratasa y 1,3Propanodiol Óxidorreductasa involucradas en la producción de 1,3PD a partir de glicerol (Montoya et al., 2006; Quilaguy et al., comunicación personal).
Un estudio completo de los genes involucrados en las rutas metabólicas de interés requiere de una estrategia más eficiente, basada en la información genómica que ha sido obtenida a nivel mundial para el género Clostridium. Sin embargo, hasta la fecha Clostridium acetobutylicum, Clostridium beijerinckii, Clostridium kluyveri y Clostridium thermocellum son las únicas especies solventogénicas y/o celulolíticas no patógenas del género cuyo genoma ha sido completamente secuenciado (Nölling et al., 2001; Copeland et al., 2007a; Copeland et al., 2007b; Seedorf et al., 2007). Un ensayo de hibridación basado en sondas universales constituye una buena alternativa, puesto que permitiría analizar la presencia y expresión de los genes involucrados en todas las rutas metabólicas sin necesidad de conocer previamente la secuencia genómica exacta de las cepas promisorias. En este trabajo se plantea el uso de un microarreglo como ensayo de hibridación, puesto que los microarreglos poseen alta capacidad de procesamiento de muestras y su costo es comparable con el de otras técnicas para el análisis de perfiles de expresión como SAGE Serial Analysis of Gene Expresión, SSH Suppression Substractive Hibridizationy MPSS Massively Parallel Signature Sequencing(Velvescu et al., 1995; Bunney et al., 2003; Stoughton, 2005). Los microarreglos de DNA son ensayos dirigidos a la secuencia genómica de un organismo que permiten analizar la concentración relativa de una gran variedad de moléculas específicas de DNA o RNA simultáneamente. Las muestras analizadas usualmente corresponden a DNA o RNA total extraído del cultivo bajo las condiciones de estudio, el cuál es hibridizado con las sondas del microarreglo para determinar la presencia y nivel de expresión de genes específicos (Stoughton, 2005). Por medio de los microarreglos se pueden identificar fenotipos bacterianos específicos, asignarle función a genes desconocidos, agrupar genes en rutas metabólicas de interés, identificar los genes reguladores de dichas rutas y otras funciones que resultan útiles en la comprensión del metabolismo fermentativo de las cepas nativas de Clostridium sp. Por ejemplo, estudios previos de cepas mutadas de C. acetobutylicum usando un microarreglo genómico han demostrado la presencia de genes reguladores necesarios para la solventogénesis y la esporulación en el megaplásmido pSOL1 (Tomas et al., 2003; Alsaker et al., 2005). El objetivo de este artículo es proponer una metodología para el diseño de sondas de DNA útiles en la construcción de un microarreglo de oligonucleótidos o en otros ensayos de hibridación enfocados al estudio y mejoramiento de las rutas metabólicas anteriormente mencionadas. Las sondas para microarreglos pueden corresponder a cDNAs preidentificados (RNA total retrotranscrito) o bien a una colección de oligonucleótidos diseñados para genes específicos. El uso de un microarreglo de oligonucleótidos supone ventajas en nuestro caso, puesto que los oligonucleótidos basados en secuencias consenso permiten detectar la expresión de genes conservados en especies cuyo genoma aún no ha sido secuenciado, como es el caso de las cepas colombianas de Clostridium sp. La utilidad de esta metodología ha sido demostrada en trabajos como la patente de Graham y Wollweber (1990), quienes diseñaron oligonucleótidos consenso para la detección de ·amilasas en diferentes especies del género Bacillus, o bien en el trabajo de Matveeva et al. (2004), quienes diseñaron oligonucleótidos consenso dirigidos al genoma del VIH1 a partir de regiones conservadas en los alineamientos múltiples de sus variantes. El uso de microarreglos de oligonucleótidos también ha sido reportado para el género Clostridium por Paredes et al. (2007), quienes diseñaron un microarreglo con oligómeros de 60 nucleótidos para el análisis metabólico en cepas mutadas de C. acetobutylicum. Este artículo presenta una metodología para el diseño de oligonucleótidos a partir de secuencias genéticas publicadas en GenBank para especies bacterianas y fúngicas representativas de las rutas metabólicas de interés, con la cual se busca compensar la baja cantidad de secuencias publicadas hasta la fecha para especies del género Clostridium. El diseño está basado en la identificación de secuencias conservadas en la mayor cantidad de especies posible, de forma que cada sonda resultante sea útil para detectar la presencia del mismo gen tanto en dichas especies como en las cepas nativas de Clostridium sp. (partiendo de su cercanía evolutiva y/o metabólica). En la metodología propuesta se han tenido en cuenta aspectos generales del diseño de oligonucleótidos para microarreglos, tales como su especificidad con respecto al gen objetivo, su incapacidad para formar homodímeros o heterodímeros con otras sondas, la ausencia de estructuras secundarias y el control de parámetros como longitud y Tm (punto de fusión) dentro de rangos previamente establecidos (Tolstrup et al., 2003).
MATERIALES Y MÉTODOS
CONSTRUCCIÓN DE LA BASE DE DATOS LOCAL MULTICLOST
A partir de una revisión bibliográfica se identificaron las enzimas que participan en la producción de acetato, butirato, etanol, acetona y butanol a partir de piruvato (fig 1) así como en la producción de 1,3propanodiol y dihidroxiacetonaP a partir de glicerol (fig 2) en clostridios solventogénicos no patógenos. También se identificaron las enzimas involucradas en la degradación de celulosa hasta glucosa, incluyendo las proteínas estructurales del celulosoma (complejo de degradación de sustratos celulósicos), y en la degradación de polisacáridos de la hemicelulosa hasta los monosacáridos correspondientes (fig 3; Schwarz, 2001). Las secuencias de los genes correspondientes fueron obtenidas en GenBank (Benson et al., 2007) y UniProt (Bairoch et al., 2005) para 16 especies del género Clostridium y 29 géneros de microorganismos relacionados ( Tabla 1). Los registros de dichas secuencias fueron almacenados y categorizados en una base de datos construida en Microsoft Access XP. Para cada registro se recopiló la siguiente información: nombre del gen, número de acceso de GenBank, nombre de la proteína codificada, número de acceso de Uniprot, especie, conglomerado (si el gen pertenece a uno), longitud del gen y longitud de la proteína.


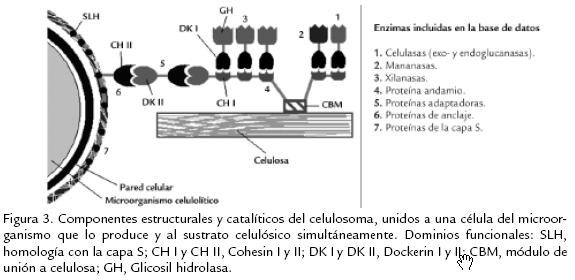

Para esta base de datos local se establecieron las siguientes categorías: (1) 'Solventogénesis', con las enzimas fosfotranscarboxilasas, carboxilato kinasas, CoA transferasas, acetoacetato descarboxilasas, aldehído deshidrogenasas y alcohol deshidrogenasas, necesarias para la producción de acetato, butirato, etanol, butanol y acetona (Jones y Woods, 1986; Biebl et al., 1999; Nölling et al., 2001); (2) '1,3Propanodiol', donde se almacenaron registros para las enzimas glicerol deshidratasa, 1,3PD deshidrogenasa, glicerol deshidrogenasa, dihidroxiacetona kinasa y glicerol3fosfatasa, necesarias para la producción de 1,3propanodiol y glicerol (Biebl et al., 1999); (3) 'Celulasas', donde se incluyeron las proteínas estructurales que forman el andamio y el sistema de anclaje del celulosoma (Bayer et al., 1998), así como celulasas entre las que se cuentan exoglucanasas, endoglucanasas, ‚glucosidasas, xilanasas, xilosidasas y mananasas (Coughlan y Mayer, 1992; Lucas et al., 2001). La subcategoría 'Celulasas' fue a su vez separada por familias de glicosil hidrolasas, dominios de unión a celulosa y dominios de homología con la capa S considerando la composición multidominio de estas enzimas (Rabinovich et al., 2002). Adicionalmente se decidió incluir la categoría 'Factor de transcripción Spo0A', dado que esta proteína de esporulación ha sido reconocida como factor de transcripción de los operones sol, adc y bdh de producción de solventes (Harris et al., 2002). La lista completa de categorías y subcategorías se presenta en la Tabla 2.

CONSTRUCCIÓN DE ALIMENTOS MÚLTIPLES CON ALTA CONSERVACIÓN DE SECUENCIAS
Para cada tipo de enzima las secuencias de los genes registrados en MULTICLOST fueron recuperadas a partir de GenBank, tomando siempre la cadena positiva de cada gen. En el caso de la categoría 'Celulasas' los grupos fueron formados por familias de dominios catalíticos o de unión a sustrato dentro de cada tipo de enzima (Bayer et al., 1998). Las secuencias de cada grupo fueron alineadas con el programa de alineamiento múltiple ClustalW 1.83 (Thompson et al., 1994) usando la matriz de respectivamente. La calidad de los alineamientos múltiples fue mejorada por eliminación de los genes con secuencia menos conservada, hasta alcanzar un valor alineamiento ClustalW1.6 (peso de 1 para las concordancias y 0 en los demás casos; Aiyar, 2000) y los valores de penalización por apertura y extensión de gaps 15 y 6,66 superior a 65 en el promedio del Puntaje de Alineamientos Pareados (PAP) calculado por ClustalW entre todos los genes de cada alineamiento (este valor mínimo se redujo a 50 en las familias enzimáticas con alta variabilidad de secuencia, como es el caso de las celulasas multidominio). Los alineamientos múltiples definitivos fueron construidos con los genes restantes, utilizando los valores de penalización 15 y 6,66 para apertura y extensión de gaps (11 y 1 para las familias enzimáticas con alta variabilidad de secuencia).
DISEÑO DE LAS SONDAS DE OLIGONUCLEÓTIDOS
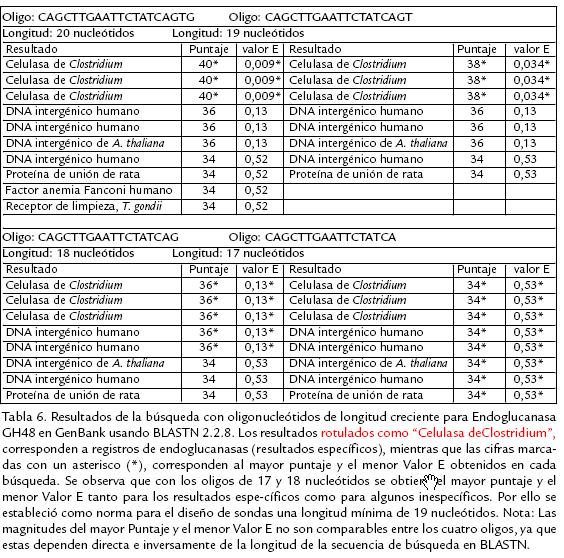
A partir de los alineamientos definitivos se extrajeron secuencias con longitud mínima de 19 nucleótidos conservadas en más del 50% de los genes de cada alineamiento, utilizando para ello el editor de alineamientos GeneDoc 2.6.002 (Nicholas y Nicholas, 2001). La longitud mínima requerida para las sondas fue establecida a partir de los resultados arrojados por BLASTN para sondas de 17 a 20 nucleótidos dirigidas a un mismo gen de Endoglucanasa ( Tabla 6). Para aumentar la probabilidad de que los oligonucleótidos diseñados puedan reconocer sus genes objetivo en las cepas promisorias de Clostridium sp., se seleccionaron siempre regiones conservadas en al menos una especie de los órdenes Clostridiales o Bacillales. Las propiedades termodinámicas de las sondas diseñadas fueron calculadas con GeneRunner v3.05 (Hastings Software, NY) y se aceptaron solo aquellas cuya Tm según la fórmula termodinámica de SantaLucia (1998) estuviera en el rango de 44 a 90 ºC y que no formaran estructuras secundarias con Tm superior a 37 ºC. Estos criterios se establecieron considerando las temperaturas de desnaturalización e hibridación recomendadas usualmente para microarreglos (42 ºC y 95 ºC respectivamente; Hedge et al., 2000; Schuchhardt et al., 2000; Tomas et al., 2003; Tummala et al., 2003; Rybicki, 2005). Para comprobar la sensibilidad y especificidad de los oligonucleótidos diseñados, éstos fueron sometidos a búsquedas en GenBank 141.0 (Benson et al., 2007) usando la aplicación BLASTN 2.2.8 (Altschul et al., 1997). Se seleccionaron aquellos oligonucleótidos para los cuales todos los registros reportados por BLASTN con Valor E (Expected Value) inferior a 0,05 correspondieran a genes con función equivalente a los genes del alineamiento original (los resultados de BLASTN son más significativos cuando su valor E es menor, pero el valor E tiende a ser conservativo cuando la secuencia de búsqueda es muy corta, de manera que para secuencias de longitud menor a 40 nucleótidos los resultados con valor E < 0,05 se pueden considerar específicos; Altschul et al., 1997). Por último se estableció la ubicación de cada sonda de oligonucleótidos en sus genes objetivo usando el programa Artemis v5 (Rutherford et al., 2000) y se determinó el dominio funcional correspondiente a cada una por medio de búsquedas de las proteínas codificadas por los genes objetivo en la base de datos CDD v1.65 (Conserved Domain Database) usando RPSBLAST 2.2.6 (Reversed Position Specific BLAST; Altschul et al., 1997).
RESULTADOS
CONSTRUCCIÓN DE LA BASE DE DATOS MULTICLOST
La base de datos construida en Microsoft Access XP fue registrada bajo el nombre MULTICLOST ante la Dirección Nacional de Derecho de Autor de Colombia (Libro 13, Tomo 17, Partida 142 del 20 de noviembre de 2006; disponible en http://www.ibun.unal.edu.co/lineas/bioprocesos/index.htm). Esta base de datos contiene registros de 485 genes que codifican para las enzimas y proteínas estructurales involucradas en la acidogénesis, solventogénesis, degradación de celulosa y producción de 1,3PD en 55 especies bacterianas y fúngicas, permitiendo recuperar las secuencias de dichos genes y proteínas en las bases de datos GenBank (Benson et al., 2007) y UniProt (Bairoch et al., 2005). De estos registros 95 corresponden a la categoría 'Solventogénesis', 94 a la categoría '1,3Propanodiol', 14 a la categoría 'Factor de esporulación Spo0A', y 282 a la categoría 'Celulasas', la más numerosa considerando la subdivisión adicional por familias de dominios funcionales.
Las especies mayoritarias de la categoría 'Celulasas' son C. acetobutylicum y C. thermocellum, las cuales representan el 25% de dicha categoría, mientras que en 'Solventogénesis' el mayor porcentaje (36%) corresponde a las especies C. acetobutylicum y Bacillus subtilis. Por su parte, la categoría '1,3Propanodiol' contiene el 25% de secuencias de las especies Clostridium butyricum, Clostridium perfringens y Citrobacter freundii. En la Tabla 2 se muestra la cantidad de registros que fueron agregados a la base de datos para cada subcategoría, mientras que en la Tabla 3 se enlistan los números de acceso en GenBank para secuencias representativas de cada subcategoría almacenadas en MULTICLOST. Los números de acceso de GenBank y UniProt contenidos en cada registro permiten recuperar la secuencia genética e información de conglomerados a partir de GenBank, así como la secuencia proteica e información biológica a partir de UniProt. Todas las enzimas incluidas en cada categoría fueron utilizadas para realizar los alineamientos múltiples.

CONSTRUCCIÓN DE ALINEAMIENTOS MÚLTIPLES CON ALTA CONSERVACIÓN DE SECUENCIAS
Las secuencias de los genes almacenados en MULTICLOST fueron usadas para crear los alineamientos a partir de los cuales se diseñarían las sondas de oligonucleótidos. Se establecieron como criterios de selección los valores PAP mínimos 65 y 50 de acuerdo con el contenido de secuencias conservadas en cada alineamiento, eliminándose de cada uno los genes que produjeran PAP inferiores a estos valores. En el caso de las celulasas se usaron parámetros más flexibles (apropiados para enzimas multidominio) y sus dominios funcionales se alinearon independientemente. Para los alineamientos de genes de las categorías 'Solventogénesis', '1,3Propanodiol' y 'Factor de esporulación Spo0A' se usaron las penalidades de apertura y extensión de gaps [156,66], establecidas por defecto en ClustalW y apropiadas para genes conservados en toda su extensión a través de diversas especies. En los alineamientos definitivos de celulasas fue necesario flexibilizar dichas penalidades, de forma que se pudieran alinear fácilmente los dominios conservados en cada familia enzimática. En estos casos se utilizaron las penalidades de apertura y extensión [11 1], correspondientes a las penalidades por defecto usadas en algunas aplicaciones de BLAST (Altschul et al., 1997). De esta forma los alineamientos definitivos fueron construidos con tres combinaciones de parámetros: 65 [15 6,66], 50 [15 6,66] y 50 [111] (PAP (apertura extensión de gaps)). Con el uso de dichas combinaciones de parámetros se obtuvieron en total 56 alineamientos definitivos para las cuatro categorías de MULTICLOST. El número de alineamientos obtenidos en cada categoría con las diferentes combinaciones de parámetros se muestra en la Tabla 4.

Dada la dificultad inherente al alineamiento de genes provenientes de especies tan diversas como las analizadas, el 77% de los alineamientos definitivos contiene dos a cuatro genes y el 18% contiene cinco o seis genes. En la figura 4, a manera de ejemplo, se observa el alineamiento múltiple de genes correspondientes a factores de esporulación Spo0A pertenecientes a varias especies de clostridios.

DISEÑO DE LAS SONDAS DE OLIGONUCLEÓTIDOS
La estrategia de diseño de oligonucleótidos en este trabajo está enfocada a la obtención de sondas que permitan identificar cada gen de interés en cualquiera de las especies que componen los alineamientos, así como en las cepas colombianas de Clostridium sp. La longitud mínima de las sondas se estableció a partir de los resultados de búsquedas en GenBank con sondas de longitud creciente diseñadas para un gen Endoglucanasa usando BLASTN. Se encontró que con sondas de longitud inferior a 19 nucleótidos resulta imposible distinguir los resultados específicos de los inespecíficos a partir de los valores de referencia 'Score' y 'Valor E' ( Tabla 6; Altschul et al., 1997). Dicha longitud se encuentra en el rango de valores reportados para el diseño de sondas de oligonucleótidos para microarreglos (Guckenberger et al., 2002, Lee et al., 2004). En total se obtuvieron 94 sondas de oligonucleótidos, algunas de las cuales se enlistan en la Tabla 5 y cuyos resultados en la búsqueda con BLASTN con Valor E inferior a 0,05 correspondieron en cada caso a las enzimas para las cuales fueron diseñadas (la lista completa de sondas está disponible como material suplementario en http://www.ibun.unal.edu.co/lineas/bioprocesos/index.htm). En promedio con cada sonda se pueden reconocer tres genes específicos para la enzima objetivo en GenBank tras las búsquedas correspondientes con BLASTN, lo cual sugiere que existe una buena probabilidad de reconocer los genes homólogos en las cepas promisorias de Clostridium. Los resultados de las búsquedas con RPSBLAST indican que las sondas diseñadas para las rutas de acidogénesis, solventogénesis y producción de 1,3PD reconocen siempre el mismo dominio en cada gen, mientras que en el caso de las celulasas cada sonda puede identificar varias enzimas de diversa función que poseen el mismo dominio funcional (considerando el carácter multidominio de las celulasas).

Las sondas diseñadas se encuentran distribuidas de la siguiente forma: 25 corresponden a la categoría 'Solventogénesis', 48 a la categoría 'Celulasas' (con sondas para varias familias de dominios funcionales en cada tipo de enzima), 19 a '1,3Propanodiol' y dos al factor Spo0A. Los valores característicos promedio de las sondas diseñadas fueron los siguientes: longitud de 24 nucleótidos, Tm termodinámica de 65,8 ºC, contenido de (G+C) de 39,0%, Tm de la horquilla más estable (cuando existe) de 10,4 ºC y ΑG de unión al objetivo de 37,4 kCal/Mol. De esta forma, se concluye que todas las sondas diseñadas pueden ser usadas para la construcción de un microarreglo hibridizado a 42 ºC (Cronin et al., 2001) o bien para un ensayo de Southern Blot realizado en condiciones similares (Harris et al., 2002). Se diseñaron además sondas para control positivo, negativo y de purificación de rRNA utilizando los mismos criterios descritos anteriormente; la sonda de control positivo 5'ATCATCCCTAACTCAACTGGTGCTG3' se diseñó a partir de la gliceraldehído3Pdeshidrogenasa de C. acetobutylicum, mientras que la sonda de control negativo 5'TGACAGATGAATCCGACACATGCAG3' fue diseñada con base en el gen de la opsina, cuyo producto (OPN1SW, GenBank: NM_001708) participa en la transducción de la señal visible en la región azul del espectro electromagnético hacia una señal electroquímica proceso que ocurre en el ojo humano. La sonda de control de purificación 5'TGGTCGGTACAATGAGATGCAACCT3' fue diseñada a partir del gen 16SrRNA de la cepa IBUN 22A (Montoya et al., 2001). Algunos de los oligonucleótidos diseñados también podrían ser utilizados como cebadores para PCR, bien sea en reacciones individuales o en múltiplex PCR. En cada caso uno de los oligonucleótidos diseñados actuaría como cebador forward, mientras que como cebador reverse se podría utilizar un oligonucleótido corto de secuencia aleatoria, como el cebador RAPD#3 (5'GTAGACCCGT3'; Amersham Pharmacia Biotech, Suecia) reportado para RAPD Random Amplified Polymorphic DNAen Clostridium difficile (Peláez et al., 2002). Se seleccionaron 27 oligonucleótidos de los diseñados con la metodología presentada anteriormente, utilizando para ello los criterios de diseño de cebadores de PCR descritos por Rybicki (2005). Estos oligonucleótidos podrían ser utilizados en cuatro reacciones de Múltiplex RTPCR en Tiempo Real separadas de acuerdo a su temperatura de anillado. El primer grupo (sondas I3A, I6A, III3, I16B, III6A) podría ser empleado en una reacción con una temperatura de anillado de 54 ºC; el segundo grupo (sondas I12B, I13, II11, III2A, II17A, II17B, II21A, II22C) en una reacción con temperatura de anillado máxima de 58 ºC; el tercer grupo (sondas II1.2, II7A, III1B, II12A, III2B, III5, II21B) en una reacción con temperatura de anillado de 63 ºC y el cuarto grupo (sondas I9, II8B, II10A, II10B, II15.2, III8A, III9) en una reacción con temperatura de anillado de 67 ºC. Cabe mencionar que los parámetros termodinámicos predichos con herramientas bioinformáticas deben ser ajustados a partir de los resultados de ensayos de hibridación preliminares.
DISCUSIÓN
La metodología seguida en este trabajo para el diseño de las sondas de oligonucleótidos está encaminada a encontrar secuencias conservadas a través de una variedad de microorganismos de los dominios Eubacteria y Eukarya, todos ellos representativos de las rutas metabólicas de interés. Esta estrategia es práctica cuando se recurre a un ensayo de hibridización en el cual la cantidad de sondas es limitada. Sin embargo, en un microarreglo puede incluirse una gran cantidad de sondas, de manera que las secuencias conservadas y sus posibles réplicas dejan muchos espacios libres que pueden ser utilizados para pruebas adicionales. Con el fin de obtener huellas de DNA de cada cepa y dar indicios sobre las relaciones evolutivas de sus genes se podría recurrir a una estrategia adicional: extraer de los alineamientos secuencias con los mismos criterios de longitud, temperatura de fusión y ausencia de estructura secundaria utilizados para los oligonucleótidos diseñados, pero esta vez en regiones que no sean compartidas por ninguno de los genes de cada alineamiento. Al utilizar estas secuencias como sondas se aprovecharía de forma más eficiente el espacio disponible en un microarreglo, y al hallar puntos hibridizados con una muestra podría decirse que la secuencia detectada tiene mayor similitud con el gen de una especie determinada incluida en el microarreglo. La utilización de sondas diseñadas a partir de secuencias conservadas en una variedad de especies constituye un ensayo exploratorio aunque no excluyente, de forma que si una de estas sondas hibridiza con el DNA o el cDNA de la cepa investigada se tiene un indicio de la presencia del gen para el cual ha sido diseñada la sonda. Si no se presenta hibridización con las sondas diseñadas para una familia enzimática, no podría asegurarse que la cepa no posea genes que codifiquen para enzimas de dicha familia.
La clasificación de las cepas nativas a nivel de especie ha presentado dificultades, y sus caracteres taxonómicos únicos podrían conducir a su clasificación como una nueva especie dentro del género. Sin embargo, aún si se llegara a concluir que son cepas de la especie C. butyricum, el genoma de esta última no ha sido completamente secuenciado ni existen por el momento proyectos genoma que la cobijen. Por este motivo se consideró pertinente buscar secuencias de genes que sean característicos del género Clostridium, pero que también se encuentren en otros géneros relacionados.
Como se presentó en la metodología de construcción de la base de datos MULTICLOST, se incluyeron en ella registros especies bacterianas y fúngicas relacionadas con Clostridium, bien fuera por su clasificación taxonómica o por exhibir las mismas rutas metabólicas. Bajo el primer criterio se incluyeron especies de los géneros Bacillus, Oceanobacillus, Geobacillus y Lactobacillus, integrantes todos ellos del Filum Firmicutes (bacterias Gram (+)), así como otras bacterias del orden Clostridiales que exhiben las rutas metabólicas pertinentes, entre las que se cuentan varias especies de los géneros Thermoanaerobacterium, Acetivibrio, Butyrivibrio, Ruminococcus y Caldicellulosiruptor (Lynd et al., 2002; Coughlan y Mayer, 1992; Rabinovich et al., 2002; Tabla 1). Otros microorganismos alejados filogenéticamente del género Clostridium fueron incluidos por ser celulolíticos reconocidos, como es el caso de Cellulomonas fimi, Hypocrea jecorina y varias especies de los géneros bacterianos Bacteroides y Fibrobacter y de los hongos Orpinomyces y Piromyces (Coughlan y Mayer, 1992; Lynd et al., 2002; Rabinovich et al., 2002). Por su parte, las enterobacterias patógenas Klebsiella pneumoniae y Citrobacter freundii fueron incluidas por ser representativas de la producción de 1,3PD a partir de glicerol (Sun et al., 2003). Por último fue necesario incluir a las levaduras Saccharomyces cerevisiae, Schizosaccharomyces pombe y Zygosaccharomyces rouxii, considerando que son los únicos microorganismos que pueden convertir el glicerolP proveniente de la glucólisis en glicerol (Biebl et al., 1999).

Como se mostró en los resultados, las subcategorías de MULTICLOST con mayor número de enzimas condujeron a la obtención de una mayor cantidad de alineamientos y secuencias conservadas. Se escogió ClustalW 1.83 como software de alineamiento con los parámetros establecidos por defecto por ser lo suficientemente bajos para compensar la variación considerable entre los genes de cada familia enzimática a través de diferentes especies (Thompson et al., 1994). La combinación de parámetros de alineamiento 65 [15 6,66] (PAP mínimo [apertura extensión de gaps]) en ClustalW fue aplicable a los grupos de genes que guardan una similitud considerable a lo largo de toda su secuencia y con ella se obtuvieron 16 alineamientos, la mayor parte correspondientes a la categoría 'Solventogénesis'. El PAP promedio en estos alineamientos va de 66 a 94, y se conformaron por grupos de dos a cinco genes. Por su parte, la combinación 50 [15 6,66] fue aplicable a grupos de genes con PAP bajos pero que conservaban similitud significativa a través de toda la secuencia. Con esta combinación de parámetros se construyeron 30 alineamientos, siendo la combinación con mayor aplicabilidad para los genes analizados en este trabajo. Con ella se obtuvieron los mayores alineamientos, que contienen hasta siete genes cada uno, aunque los valores promedio de PAP descienden a un rango de 51 a 71,1. La combinación 50 [111] constituyó la mejor opción en grupos cuyos genes comparten solo ciertos dominios funcionales, como es el caso de la mayoría de las celulasas multidominio (Bayer et al., 1998). Con esta combinación se construyeron 11 alineamientos con PAP promedio que van de 55,9 a 77. A partir de las regiones conservadas en dichos alineamientos se diseñaron 94 sondas de oligonucleótidos. Uno de los principales sustentos teóricos para la búsqueda de secuencias conservadas en los genes estudiados es que en las especies correspondientes se han demostrado eventos de transferencia horizontal, siendo este de hecho el origen de varios operones en el genoma de C. acetobutylicum (Nölling et al., 2001). En este estudio se dedujo la transferencia horizontal de la Endoglucanasa V de Erwinia chrysantemi a Cellulomonas uda, en donde se identifica como Endoglucanasa I, a partir de sus semejanzas en cuanto a secuencia nucleotídica, frecuencia de uso de codones y porcentaje de G+C. Sin embargo, los genes provenientes de especies fúngicas y actinomicetos almacenados en MULTICLOST formaron casi invariablemente alineamientos separados de aquellos en los cuales se concentraban genes de las clases Clostridia y Bacilli, aun con el uso de parámetros de alineamiento poco restrictivos (datos no mostrados). Este resultado contrasta con la hipótesis adelantada por GarcíaVallve et al. (2000), en la que se propone que los genes de celulasas en hongos del rúmen fueron obtenidos mediante transferencia horizontal a partir de bacterias del rúmen (hipótesis reforzada por la ausencia de intrones en dichos genes). La base de datos MULTICLOST y las sondas de oligonucleótidos diseñadas son herramientas muy útiles para el mejoramiento futuro de las cepas nativas de Clostridium sp. por ingeniería metabólica, puesto que permiten analizar y comprender las rutas metabólicas implicadas en el consumo de sustratos de bajo costo y en la producción de metabolitos de alto valor comercial en diferentes especies. Con MULTICLOST se obtiene una visión amplia de las especies que representan cada ruta metabólica, así como de las variaciones enzimáticas que existen entre ellas, mientras que con las sondas de oligonucleótidos se puede analizar el perfil de expresión de los genes implicados en cada ruta. La estrategia propuesta para el diseño de oligonucleótidos guarda semejanza con la estrategia propuesta en la patente de Graham y Wollweber (1990) para la detección de genes de α-Amilasa en diferentes especies del género Bacillus, así como con la estrategia diseñada por Matveeva et al. (2004) para el análisis de variantes del virus VIH1 por ensayos de hibridación. Sin embargo, la estrategia para el análisis de rutas metabólicas en las cepas colombianas de Clostridium sp. muestra ventajas con respecto a la especificidad de detección y a la cantidad de sondas obtenidas. En la estrategia patentada por Graham y Wollweber (1990), por ejemplo, se recurre al uso de nucleótidos ambiguos o bien se escogen nucleótidos arbitrarios en las posiciones sin consenso de los alineamientos múltiples, arriesgando de esta forma la especificidad y capacidad de detección de las sondas resultantes. Por otra parte la estrategia de Matveeva et al. (2004) es muy similar a la de este artículo en cuanto a la selección de sondas a partir de los alineamientos múltiples por parámetros de longitud, valores termodinámicos y formación de estructuras secundarias o dímeros, pero su estructura automatizada y exhaustiva resulta poco práctica para el diseño de un número limitado de sondas que cubran un amplio espectro de genes. La estrategia utilizada en el presente estudio resultó eficaz para el diseño de sondas dirigidas a regiones consenso de cada gen objetivo, con las cuales se espera analizar en el futuro las rutas metabólicas de interés en las cepas colombianas de Clostridium sp. y así mejorar eventualmente su rendimiento industrial.
AGRADECIMIENTOS
Este trabajo fue desarrollado dentro del marco del proyecto "Producción de 1,3 Propanodiol a partir de glicerol generado del proceso productivo de Biodiesel, empleando cepas nativas de Clostridium spp.: estudio del operón condiciones de producción por fermentación y su viabilidad económica", código 11011217848 financiado por el Instituto Colombiano Francisco José de Caldas COLCIENCIAS y la Universidad Nacional de Colombia.
BIBLIOGRAFÍA
AIYAR A. The Use of CLUSTAL W and CLUSTAL X for Multiple Sequence Alignment. En: Misener S, Krawetz S, editors. Bioinfomatics: Methods and protocols. New Jersey: Humana Press Inc.; 2000. p. 221-239. [ Links ]
ALSAKER K, PAPOUTSAKIS E. Transcriptional Program of Early Sporulation and StationaryPhase Events in Clostridium acetobutylicum. J Bacteriol. 2005;187(20):7103-7118. [ Links ]
ALTSCHUL S, MADDEN T, SCHÄFFER A, ZHANG J, ZHANG Z, MILLER W, et al. Gapped BLAST and PSIBLAST: A New Generation of Protein Database Search Programs. Nucleic Acids Res. 1997;25(17):3389-3402. [ Links ]
ARÉVALO C. Contribución a la caracterización molecular de cepas nativas de Clostridium spp. con potencial biotecnológico [tesis de maestría]. Bogotá D.C.: Posgrado Interfacultades en Microbiología, Universidad Nacional de Colombia; 2001. [ Links ]
BAIROCH A, APWEILER R, WU C, BARKER W, BOECKMANN B, FERRO S, et al. The Universal Protein Resource (UniProt). Nucleic Acids Res. 2005;33:D154-D159. [ Links ]
BAYER E, SHIMON L, SHOHAM Y, LAMED R. CellulosomesStructure and Ultrastructure. J Struct Biol. 1998;124(23):221-234. [ Links ]
BENSON D, KARSCHMIZRACHI I, LIPMAN D, OSTELL J, WHEELER D. GenBank. Nucleic Acids Res. 2007;35(Database issue):D21-D25. [ Links ]
BIEBL H, MENZEL K, ZENG A, DECKWER W. Microbial Production of 1,3Propanediol. Appl Microbiol Biotechnol. 1999;52(3):289-297. [ Links ]
BUNNEY W, BUNNEY B, VAWTER M, TOMITA H, LI J, EVANS S, et al. Microarray Technology: A Review of New Strategies to Discover Candidate Vulnerability Genes in Psychiatric Disorders. Am J Psychiatry. 2003;160(4):657-666. [ Links ]
CÁRDENAS D, PULIDO C, ARAGÓN O, ARISTIZÁBAL F, SUÁREZ Z, MONTOYA D. Evaluación de la producción de 1,3propanodiol por cepas nativas de Clostridium sp. mediante fermentación a partir de glicerol USP y glicerol industrial subproducto de la producción de Biodiesel. Rev Col Cienc Quím Farm. 2006;35(1):120-137. [ Links ]
COUGHLAN M, MAYER F. The CelluloseDecomposing Bacteria and their Enzyme Systems. En: Balows A, editor. The prokaryotes: A Handbook on the Biology of Bacteria. 2 ed. New York: SpringerVerlag; 1992. p. 460-502. [ Links ]
CRONIN M, PHO M, DUTTA D, FRUEH F, SCHWARCZ L, BRENNAN T. Utilization of New Technologies in Drug Trials and Discovery. Drug Metab Dispos. 2001;29(4):586-590. [ Links ]
GARCÍAVALLVE S, ROMEU A, PALAU J. Horizontal Gene Transfer of Glycosyl Hydrolases of the Rumen Fungi. Mol Biol Evol. 2000;17(3):352-361. [ Links ]
GRAHAM B, WOLLWEBER K. Oligonucleotide Probes for Detection of AlphaAmylase Genes. Washington D.C.: U.S. Patent and Trademark Office; 1990. U.S. Patent 4917999. [ Links ]
GUCKENBERGER M, KURZ S, AEPINUS C, THEISS S, HALLER S, LEIMBACH T, et al. Analysis of the Heat Shock Response of Neisseria meningitidis with cDNAand OligonucleotideBased DNA Microarrays. J Bacteriol. 2002;184(9):2546-2551. [ Links ]
HARRIS L, WELKER N, PAPOUTSAKIS E. Northern, Morphological, and Fermentation Analysis of spo0A Inactivation and oOverexpression in Clostridium acetobutylicum ATCC 824. J Bacteriol. 2002;184(13):3586-3597. [ Links ]
HEGDE P, QI R, ABERNATHY K, GAY C, DHARAP S, GASPARD R, et al. A Concise Guide to cDNA Microarray Analysis. Biotechniques. 2000;29(3):548-562. [ Links ]
JAIMES C, ARISTIZÁBAL F, BERNAL M, SUÁREZ Z, MONTOYA D. AFLP Fingerprinting of Colombian Clostridium spp. Strains, Multivariate Data Analysis and Its Taxonomical Implications. J Microbiol Methods. 2006;67(1):64-69. [ Links ]
JONES D, WOODS D. AcetoneButanol Fermentation Revisited. Microbiol Rev. 1986;50(4):484-524.
LEE I, DOMBKOWSKI A, ATHEY B. Guidelines for Incorporating NonPerfectly Matched Oligonucleotides Into TargetSpecific Hybridization Probes for a DNA Microarray. Nucleic Acids Res. 2004;32(2):681-690. [ Links ]
LUCAS R, ROBLES A, GÁLVEZ A, GARCÍA T, PÉREZ R, ÁLVAREZ G. Biodegradación de la celulosa y la lignina. Jaén: Junta Andalucia Consejería EDU; 2001. p. 14-62. [ Links ]
LYND L, WEIMER P, VAN ZYL W, PRETORIUS I. Microbial Cellulose Utilization: Fundamentals and Biotechnology. Microbiol Mol Biol Rev. 2002;66(3):506-527. [ Links ]
MATVEEVA O, FOLEY B, NEMTSOV V, GESTELAND R, MATSUFUJI S, ATKINS J, et al. Identification of Regions in Multiple Sequence Alignments Thermodynamically Suitable for Targeting by Consensus Oligonucleotides: Application to HIV Genome. BMC Bioinformatics [serial online] 2004 [citado 5 Sep 2007]; 5(44): [15 pantallas]. Disponible en: URL: http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=419695. [ Links ]
MONTOYA D, SPITIA S, SILVA E, SCHWARZ W. Isolation of Mesophilic SolventProducing Clostridia from Colombian Sources: Physiological Characterization, Solvent Production and Polysaccharide Hydrolysis. J Biotechnol. 2000;79(2):117-126. [ Links ]
MONTOYA D, ARÉVALO C, GONZALES S, ARISTIZÁBAL F, SCHWARZ W. New SolventProducing Clostridium sp. Strains, Hydrolyzing a Wide Range of Polysaccharides, are Closely Related to Clostridium butyricum. J Ind Microbiol Biotechnol. 2001;27(5):329-335. [ Links ]
MONTOYA J, SUÁREZ Z, MONTOYA D, ARISTIZÁBAL F. Análisis bioinformático y predicción de genes en secuencias genómicas de Clostridium sp. IBUN22A. Rev Col Biotecnol. 2006;8(1):57-64. [ Links ]
NAKAMURA C, WHITED G. Metabolic Engineering for the Microbial Production of 1,3Propanediol. Curr Opin Biotechnol. 2003;14(5):454-459. [ Links ]
NICHOLAS K, NICHOLAS H. GeneDoc: A Tool for Editing and Annotating Multiple Sequence Alignments [programa de ordenador online]. Versión 2.6.002. Pittsburg (PA): Pittsburgh Supercomputing Center; 2001. Disponible en: URL: http://www.psc.edu/biomed/genedoc/. [ Links ]
NÖLLING J, BRETON G, OMELCHENKO M, MAKAROVA K, ZENG Q, GIBSON R, et al. Genome Sequence and Comparative Analysis of the SolventProducing Bacterium Clostridium acetobutylicum. J Bacteriol. 2001;183(16):4823-4838. [ Links ]
PACHAURI N, HE B. ValueAdded Utilization of Crude Glycerol from Biodiesel Production: A Survey of Current Research Activities. Portland: 2006 ASABE Annual International Meeting; 2006. Paper Number: 066223. [ Links ]
PAREDES C, SENGER R, SPATH I, BORDEN J, SILLERS R, PAPOUTSAKIS E. A General Framework for Designing and Validating OligomerBased DNA Microarrays and its Application to Clostridium acetobutylicum. Appl Environ Microbiol. 2007;73(14):4631-4638. [ Links ]
PELÁEZ T, ALCALÁ L, ALONSO R, RODRÍGUEZCRÉIXEMS M, GARCÍALECHUZ J, BOUZA E. Reassessment of Clostridium difficile Susceptibility to Metronidazole and Vancomycin. Antimicrob Agents Chemother. 2002;46(6):1647-1650. [ Links ]
RABINOVICH M, MELNICK M, BOLOBOVA A. The Structure andMechanism of Action of Cellulolytic Enzymes. Biochemistry Moscow. 2002;67(8):850-871. [ Links ]
RUTHERFORD K, PARKHILL J, CROOK J, HORSNELL T, RICE P, RAJANDREAM M, et al. Artemis: Sequence Visualisation and Annotation. Bioinformatics. 2000;16(10):944-945. [ Links ]
RYBICKI E. A Manual of Online Molecular Biology Techniques. Cape Town: University of Cape Town; 2005. Disponible en: URL: http://www.mcb.uct.ac.za /manual/MolBiolManual.htm. [ Links ]
SANTALUCIA J. A Unified View of Polymer, Dumbbell, and Oligonucleotide DNA NearestNeighbor Thermodynamics. Proc Natl Acad Sci USA. 1998;95:1460-1465. [ Links ]
SCHUCHHARDT J, BEULE D, MALIK A, WOLSKI E, EICKHOFF H, LEHRACH H, et al. Normalization Strategies for cDNA Microarrays. Nucleic Acids Res. 2000;28(10):e47ie47v. [ Links ]
SCHWARZ, W. The Cellulosome and Cellulose Degradation by Anaerobic Bacteria. Appl Microbiol Biotechnol. 2001;56(56):634-649. [ Links ]
STOUGHTON R. Applications of DNA Microarrays in Biology. Annu Rev Biochem. 2005;74:53-82. [ Links ]
SUÁREZ Z. Contribución a la taxonomía de 13 cepas nativas de Clostridium sp. mediante análisis multivariado de técnicas de caracterización genotípicas y fenotípicas [tesis de maestría]. Bogotá D.C.: Posgrado Interfacultades en Microbiología, Universidad Nacional de Colombia.; 2004. [ Links ]
SUN J, VAN DER HEUVEL J, SOUCAILLE P, QU Y, ZENG A, et al. Comparative Genomic Analysis of Dha Regulon and Related Genes for Anaerobic Glycerol Metabolism in Bacteria. Biotechnol Prog. 2003;19(2):263-272. [ Links ]
THOMPSON J, HIGGINS D, GIBSON T. Clustal W: Improving the Sensitivity of Progressive Multiple Sequence Alignment Through Sequence Weighting, PositionSpecific Gap Penalties and Weight Matrix Choice. Nucleic Acids Res. 1994;22(22):4673-4680. [ Links ]
TOLSTRUP N, NIELSEN P, KOLBERG J, FRANKEL A, VISSING H, KAUPPINEN S. OligoDesign: Optimal Design of LNA (Locked Nucleic Acid) Oligonucleotide Capture Probes for Gene Expression Profiling. Nucleic Acids Res. 2003;31(13):3758-3762. [ Links ]
TOMAS C, ALSAKER K, BONARIUS H, HENDRIKSEN W, YANG H, BEAMISH J, et al. DNA ArrayBased Transcriptional Analysis of Asporogenous, Nonsolventogenic Clostridium acetobutylicum strains SKO1 and M5. J Bacteriol. 2003;185(15):4539-4547. [ Links ]
TUMMALA S, JUNNE S, PAPOUTSAKIS E. Antisense RNA Downregulation of Coenzime A Transferase Combined with AlcoholAldehyde Dehydrogenase Overexpression Leads to Predominantly Alcohologenic Clostridium acetobutylicum Fermentations. J Bacteriol. 2003;185(12):3644-3653. [ Links ]
VELCULESCU V, ZHANG L, VOGELSTEIN B, KINZLER K. Serial Analysis of Gene Expression. Science. 1995;270:484-487. [ Links ]
VARGAS C, MONTOYA D, ARISTIZÁBAL F. Clonación y expresión en Escherichia coli de genes de celulasas de Clostridium IBUN 22A. Rev Col Biotecnol. 2002;4(1):29-35. [ Links ]
ZVERLOV V, BEREZINA O, VELIKODVORSKAYA G, SCHWARZ W, et al. Bacterial Acetone and Butanol Production by Industrial Fermentation in the Soviet Union: Use of Hydrolyzed Agricultural Waste for Biorefinery. Appl Microbiol Biotechnol. 2006;71(5):587-597. [ Links ]














