INTRODUCCIÓN
Staphylococcus aureus es la principal especie patógena de su género que causa infecciones hospitalarias y comunitarias 1,2. La capacidad que presenta en adquirir diferentes mecanismos de resistencia a los antibióticos ha favorecido su presencia en ambientes hospitalarios durante décadas, en especial, el S. aureus resistente a meticilina (SARM) 3-5.
En la comunidad, algunos grupos de personas son más propensos a estar colonizados, como el personal sanitario 7-10. Se ha observado que pueden existir hasta un 50% de portadores nasales en el personal médico de los hospitales 11,12.
S. aureus no solo coloniza el cuerpo humano sino también las superficies de diversos ambientes. Numerosos estudios han demostrado que el ambiente de la habitación del paciente puede estar colonizado por SARM, entre otros patógenos 13-17.
La diseminación de los microorganismos en el ambiente hospitalario tiene causas multifactoriales, a menudo se origina a partir de la contaminación cruzada; el medio más común de transferencia de patógenos se produce a través de las manos de los profesionales de la salud y de la flora de los pacientes 15-17. Sin embargo, el ambiente del hospital puede contribuir con la diseminación de patógenos 15,16. Estudios en ambientes hospitalarios han demostrado que los pacientes admitidos en habitaciones donde habían sido tratados pacientes por infecciones resultaron estar colonizados por el mismo microorganismo 14-16.
Está bien establecido que la adquisición de patógenos nosocomiales depende de una compleja interacción del huésped, el patógeno y el medio ambiente 15,16. La inadecuada asepsia fortalece la diseminación de patógenos entre las salas o equipos biomédicos del hospital, creando un ambiente de riesgo para el personal de salud y para los pacientes o familiares 9,10.
En este estudio se caracterizó fenotípica y molecularmente el S. aureus con resistencia a los antibióticos que se encuentran colonizando al personal de salud y al ambiente hospitalario, como aporte al conocimiento del comportamiento epidemiológico de la bacteria en el hospital.
MATERIALES Y MÉTODOS
Se trató de un estudio analítico realizado en un hospital de cuarto nivel de complejidad durante el año 2013.
Las muestras se tomaron con hisopos de algodón estériles mediante frotis de la mucosa nasal y de piel del personal de salud que aceptó su participación en el estudio mediante consentimiento informado. Los aislamientos se manejaron por códigos, respetando de esta manera la confidencialidad del participante. Las muestras ambientales se obtuvieron frotando las superficies de equipos e instrumentos (barandas de las camas, los monitores de signos vitales, atriles, y demás equipos biomédicos), que se encontraban en las diferentes áreas de las unidades de cuidados intensivos (UCI), unidad de cuidados intermedios (UCIN) y salas de cirugía y se procesaron inmediatamente en el laboratorio de microbiología
Cepas y condiciones de cultivo
El aislamiento de especies del género Staphylococcus se realizó sembrando muestras en el medio de cultivo agar salino manitol rojo de fenol (Oxoid Ltd., Hampshire, United Kingdom) y se incubaron por 24 a 48 horas a 37°C. La identificación de S. aureus se efectuó por la fermentación del manitol en el agar selectivo (coloración amarilla del medio), y por la reacción positiva de las pruebas de la coagulasa y la Dnasa, y se confirmó con la observación al microscopio de cocos Gram positivos en racimos, a partir de un extendido directo con tinción de Gram.
Pruebas de susceptibilidad a los antibióticos
La prueba de susceptibilidad antimicrobiana se realizó empleando el método por difusión en agar de acuerdo con la técnica de Kirby-Bauer 18.
Se inoculó una cantidad estandarizada (estándar 0,5 de McFarland) de S. aureus en un medio de agar Mueller-Hinton; a continuación se colocaron discos de susceptibilidad antibiótica, los cuales correspondieron: Oxacilina (OXA, 1 μg), Cefoxitina (FOX, 30 μg), Cefalexina (CS, 30 µg), Gentamicina (GEN, 10 µg), Ciprofloxacina (CIP, 5 μg), Eritromicina (E, 15 μg), Clindamicina (CC, 2 μg),Trimetoprima-Sulfametoxazol (TMP/ SUL 1,25/23,75 μg), Tetraciclina (TET, 30 μg), Cloranfenicol (CL, 30 μg), Vancomicina (VA, 30 μg), Imipenem (IMP, 10 μg), Penicilina (PEN, 10U). El análisis de susceptibilidad se realizó con la cepa ATCC 25923 de S. aureus para verificar la acción de los sensidiscos.
Para conocer el S. aureus meticilino-resistente se determinó el resultado de susceptibilidad con la prueba screening a cefoxitina de acuerdo con los lineamientos establecidos por el Clinical and Laboratory Standards Institute (CLSI) 19.
Aislamiento del ADN de las cepas de referencia y de los aislamientos bacterianos
La extracción del ADN se realizó empleando el protocolo modificado de Cheng et al., 2006 20 a partir de bacterias cultivadas en caldo (1,5 ml) de Lisogenia “Luria-Bertani” (LB) durante toda la noche.
Para confirmar la resistencia a meticilina se amplificó por PCR una banda 1334 pb del gen mecA con los cebadores MR1 y MR2 siguiendo el protocolo reportado por Tokue et al., 1992 21. Las cepas de S. aureus ATCC® 43300 y ATCC ®25923 fueron empleadas como controles positivo y negativo, respectivamente.
La evaluación de los tipos SCC mec en los aislados SARM se realizó mediante PCR multiplex como lo describió previamente 22.
Los grupos agr se determinaron entre los aislamientos de S. aureus sensibles a meticilina (SASM) y SARM mediante la amplificación
independiente de los fragmentos de 440 bp, 572 bp, 406 bp y 588 pb, empleando un juego de cebadores, pan-agr, agr1, agr2, agr3 y agr4 23,24.
La secuencia de los cebadores empleados para las amplificaciones se muestra en la tabla 1.
Tabla 1 Genes blanco para ser amplificados por PCR y secuencias de cebadores empleados en este estudio.

Todas las reacciones de PCR se realizaron en 50 μl (volumen final) en un termociclador GeneAmp PCR system 2400 (Perkin-Elmer Instruments®, Norwalk, Conn).
Análisis estadístico
Los resultados obtenidos fueron analizados mediante estadística descriptiva con la prueba del Chi-cuadrado (χ2) y un valor de P<0,05 para un valor significativo. El análisis del factor de riesgo para la colonización de SARM y de S. aureus sensibles a meticilina (SASM) en el personal de salud y la contaminación de las superficies hospitalarias se desarrolló empleando el paquete estadístico SPSS Vs 22.0 (Chicago, Inc).
RESULTADOS
Se recolectó un total de 164 muestras, 86 fueron de hisopado nasal y de piel del personal de salud (64 mujeres y 22 hombres) con un promedio de edad de 28 años (DS=12,353; min=21, max=59).
Setenta y ocho muestras se obtuvieron de las superficies de las salas hospitalarias: 24 muestras se tomaron en la UCI, 16 muestras en la UCIN y 38 muestras en la sala de cirugía.
La prevalencia del S. aureus fue del 21,3% (35/164), el 9,1% (15/164) de los aislamientos se determinó en el personal de salud, específicamente los que laboraban en las salas de urgencias (6,7 %) y la UCI (2,4%). La colonización en el personal femenino representó el 6,7% y en los hombres el 2,4%. El 4,8% de estos aislamientos fueron detectados en rastreo nasal y el 4,3% en piel (tabla 2).
Enel ambiente hospitalario, el S. aureus se detectó en el 12,2% (20/167) de los casos, siendo significativa la presencia de esta bacteria en la UCI (6,1%, P=0,031). En esta sala la presencia del S. aureus representó un mayor riesgo (OR) de 3,143 (tabla 2).
Tabla 2 Factores asociados con la presencia del Staphylococcus aureus.

OR= odds Ratio; 95% con un intervalo de confianza (IC) del 95% UCIN: Unidad de Cuidados Intermedios; UC: Unidad de Cuidados Intensivos.
Análisis de las pruebas de susceptibilidad a los antibióticos en aislamientos de S. aureus
Los resultados de la prueba de susceptibilidad a los antibióticos muestran que, en el personal de salud, el mayor número de aislamientos fue resistente a ampicilina (40%), tetraciclina
(37,1%) y clindamicina (25,7%) y en el ambiente hospitalario a tetraciclina (31,4%), ampicilina (28,6%) y ciprofloxacina (22,9%) (Datos no mostrados).
Basado en el análisis de esta prueba se logró determinar tres perfiles de resistencia que se muestran en la tabla 3. El perfil I encontrado en bacterias resistentes a ampicilina y gentamicina fue el que mayor riesgo presentó de estar en el ambiente hospitalario (20%; OR= 3,500; IC95% min= 0,050, max = 16,430; P= 0,147).
Tabla 3 Patrones de resistencia a los antibióticos entre los aislamientos de S. aureus. n=35.

*I (resistencia simultánea a: AMP y GEN)
†II (resistencia simultánea a: AMP, TET, GEN)
‡III (resistencia simultánea a: AMP, TET, GEN, CLI, CIP, CHL, SXT, ERY, OXA/FOX)
AMP, ampicilina; OXA, oxacilina; ERY, eritromicina; GEN, gentamicina; CLI, clindamicina; CHL, chloramfenicol; CIP, ciprofloxacina; TET, tetraciclina; SXT, trimethoprima-sulfamethoxazol, FOX, cefoxitina
El perfil II con resistencia a ampicilina, gentamicina y tetraciclina se determinó en aislamientos obtenidos del personal de salud (11,4%) y en las superficies de las salas hospitalarias (17,1%) con un OR de 1,179.
El perfil de resistencia a ampicilina, gentamicina, tetraciclina, clindamicina, ciprofloxacina, cloranfenicol, eritomicina, oxacilina o cefoxitina, denominado perfil III, fue el más prevalente (45,7%) y se detectó en el 25,7% del personal de salud y en el 20% en el ambiente hospitalario con un OR de 2,786.
El 28,6% (10/35) de los aislamientos fueron SARM y todos presentaron el gen mecA. El 11,4% (4/35) se detectó en el personal de salud y el 17,1% (6/35) en las superficies del ambiente hospitalario.
Entre los aislamientos de SARM solo se detectó el SCCmec tipo II, este tipo es compatible con un origen hospitalario 22,25. Ocho aislamientos se detectaron en los SARM que contaminaron las superficies de las salas del hospital y dos que colonizaron al personal de salud. Todos estos aislamientos se asociaron en forma significativa al perfil de resistencia III (fenotipo con multirresistencia a los antibióticos, incluidos los β-lactámicos, lo que confirma su origen hospitalario) con un OR de 23,143 de presentar este fenotipo.
De acuerdo al análisis del locus agr se encontró que 18 (51,4%) aislamientos pertenecían al grupo agr 1, diez (28,6%) al agr 2 y nueve (25,7%) al agr 3. No se detectó el agr 4 entre los aislamientos. El agr 1 se asocia con cepas de origen comunitario y el agr 2 con un origen hospitalario (tabla 4) (23, 24).
Tabla 4 Distribución de los perfiles de resistencia y grupos agr entre los aislamientos de SARM y SASM obtenidos en este estudio. n=35
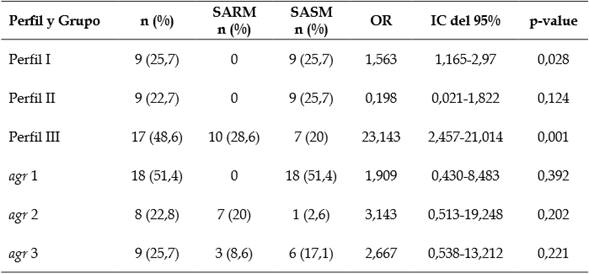
SARM: Staphylococcus aureus resistente a meticilina, SASM: Staphylococcus aureus sensible a meticilina.
El agr 1 se presentó sólo entre los aislamientos SASM y se detectó en igual número en el personal de salud y en el ambiente hospitalario. El agr 2 se detectó en aislamientos que contaminaron las superficies de las salas del hospital, 20% en los aislamientos de SARM y 2,6% en los aislamientos SASM.
El agr 3 se detectó en el 8,6% de los aislamientos del personal de salud (todos aislamientos de SARM) y en el 17,1% de los aislamientos SASM recuperados de las superficies del ambiente hospitalario.
DISCUSIÓN
Nuestros resultados evidencian la presencia de S. aureus en el personal de salud (9,1%) y el ambiente hospitalario (12,2%), lo que evidencia la capacidad de adaptación que tiene esta bacteria a diferentes ambientes, debido quizás en gran medida por el amplio repertorio genético que presenta este microorganismo, que le permite adaptarse rápidamente en el medio que se encuentre 9-17.
Los portadores de S. aureus en el personal de salud y su presencia en el ambiente hospitalario vienen siendo reportados como un factor de gran relevancia en la transmisión del microorganismo 9,11,13. En muchos casos se ha determinado que la colonización nasal en los trabajadores de salud y pacientes, normalmente precede a la infección intrahospitalaria por esta bacteria 26-28.
Sin embargo, en los Estados Unidos se reportaba inicialmente una prevalencia del 2,4%, en el personal de salud, la cual ascendió a 29% para 1992 29. Pero, en el 2004, se estimó que menos del 6% del personal de salud se encontraba colonizado. Esta disminución probablemente se debió a la intensificación en el empleo de medidas de control, como el lavado de manos, el uso de barreras de contención y otras medidas, como se ha venido estableciendo en forma rutinaria en diversos hospitales 30-32, que deben ser aplicadas de forma rigurosa en los hospitales de nuestra región, con el objetivo de disminuir el aumento en la colonización bacteriana.
La caracterización fenotípica basada en la prueba de susceptibilidad a los antibióticos permitió determinar tres perfiles, siendo el perfil III el más frecuente (45,7%) con la característica de multirresistencia a los antibióticos. En concordancia con un estudio realizado en el 2008 en Chile, con 60 aislamientos de S. aureus obtenidos de pacientes y portadores asintomáticos del personal de salud, cuando se determinó que, de seis perfiles, el perfil multirresistente fue el más predominante (82%) 33.
Un estudio realizado por Olarte et al. en Bogotá durante 2010 determinó que en 182 aislamientos de S. aureus obtenidos de rastreo nasal de pacientes, existían siete perfiles de resistencia 34, lo que demuestra que en los portadores asintomáticos de S. aureus existen aislados con resistencia a los antibióticos con variados perfiles de resistencia, debido quizás a la transferencia de bacterias desde el ambiente, del personal de salud, de otros pacientes o por la generación de resistencia en la flora autóctona del individuo por el consumo de antibióticos sin prescripción médica 9,13,15.
En cuanto a los análisis moleculares, se encontró que todos los aislamientos SARM presentaron el gen mecA, lo que evidencia un 100% de concordancia entre los métodos fenotípicos y moleculares. Algunos estudios reportan resultados similares 35,36, aunque otros estudios reportan niveles de concordancia más bajos que van del 96% 37 hasta solo del 51% como el encontrado por Girgis et al. 38.
Los aislamientos SARM fueron detectados en el personal de salud y en las superficies de las salas del hospital, por lo que deben ser considerados como focos de transmisión de este patógeno. Se ha demostrado que el modo principal de transmisión de SARM se da por contacto directo con un individuo colonizado o infectado y con objetos y superficies contaminados 15,31. Este comportamiento quizás sea una de las causas por las que las infecciones causadas por este patógeno, en particular la causada por cepas de SARM, están aumentando en muchos países, tanto en el ámbito sanitario como en el comunitario 2,3. Huang et al. 39 demostraron que la colonización por SARM en los pacientes se relacionó con la infección y el 29% de portadores asintomáticos desarrollaron infecciones invasivas en 18 meses.
Entre los aislamientos SARM todos fueron SCCmec tipo II, estos aislados presentaron el fenotipo multirresistente a los antibióticos del perfil III, que se asocia con aislamientos del ambiente hospitalario (SARM-AH).
Los aislamientos de SARM que se reportaban en Colombia, inicialmente se registraban como hospitalarios, especialmente compatibles con el clon chileno, 40. Sin embargo, en 2006 se reportó el SARM asociado a la comunidad (SARM-AC) 41 y a partir de esta fecha se ha reportado el incremento de este microorganismo 42,43.
Los aislamientos colombianos de SARM-AC son genéticamente compatibles con el clon USA300 44,45, las cepas con este clon no presentan el patrón típico de multirresistencia de los SAMR-AH, sino a un rango más restringido de antibióticos, siendo sensibles a vancomicina y generalmente también a clindamicina, trimetroprima/sulfametoxazol, doxiciclina y rifampicina con sensibilidad variable a quinolonas 46.
Se ha sugerido que el origen del Clon USA 300 se dio quizás de aislamientos de SASM que se encontraban colonizando a la comunidad, y al interactuar con el SARM adquirió características propias 47. Una característica encontrada en este estudio que apoya la hipótesis planteada por Escobar et al. 47, fue que el grupo agr 1, el cual se relaciona con cepas SARM comunitarias 48, fue encontrado en el 51,4% de los aislamientos SASM.
Por otra parte, la distribución de los grupos agr entre los aislamientos de SARM y SASM encontrada en este estudio apoya lo expuesto por Shopsin et al. 45, quienes sostienen que las cepas de S. aureus presentan una capacidad competitiva que las hace crear nichos ecológicos separados para desarrollarse; en este sentido, encontramos que los aislamientos pertenecientes al grupo agr 1 se encontraron sólo en aislamientos de SASM, el grupo agr 2 en aislamientos ambientales de la UCI y el grupo agr 3 en aislamientos SARM que colonizaron al personal de salud (8,6%) y en los aislamientos SASM recuperados del ambiente hospitalario de la sala de urgencias (17,1%). Mientras que el agr 4 no se detectó en algún aislado, quizás esté siendo inhibido competitivamente por los otros grupos.
La diversidad de grupos agr presente entre los 35 aislamientos de S. aureus supone la posibilidad de intercambio entre cepas, mezclando determinantes de resistencia y de patogenicidad, lo que puede favorecer la presencia de cepas más adaptables a diferentes ambientes como lo ha reportado Manago et al. 49.
CONCLUSIÓN
En este estudio se evidenció la presencia de SARM en las diferentes salas del mismo hospital. Sin embargo, es necesario adelantar estudios posteriores de genotipificación para evidenciar transmisión cruzada de estas cepas entre las diferentes salas del hospital, el personal de salud y quizás en pacientes. Esta condición es un factor de riesgo para desarrollar infecciones adquiridas en el hospital. La constante movilidad del personal de salud entre las salas del mismo hospital y probablemente entre hospitales, plantea la necesidad de generar estrategias de prevención y de control de diseminación de SARM y SASM de forma institucional y en red.
Es necesario seguir realizando estudios epidemiológicos para evaluar el comportamiento de esta bacteria y profundizar en el posible origen de los aislamientos SARM.
Una limitación en nuestro estudio fue la evaluación de únicamente tres salas, es necesario ampliar el análisis de todas las salas del hospital y pruebas de genotipificación, con el fin de determinar el patrón de diseminación de los aislamientos SARM y SASM, pues estos aislamientos pueden ser el reservorio de aislamientos más patogénicos y virulentos como se ha demostrado con el clon USA-300.














