Introducción
Colombia posee una enorme diversidad étnica representada en 87 pueblos indígenas con cerca de 21 estirpes lingüísticas (13 familias lingüísticas y 8 lenguas aisladas), más del 36% habitando el occidente colombiano (Arango & Sánchez, 2004 y Departamento Administrativo Nacional de Estadística, 2007). A pesar de esta amplia gama de etnias amerindias, es preocupante la escasa y fragmentada información sobre su origen, además del desconocimiento de las relaciones filogenéticas entre los diferentes grupos. Si se toma en consideración que algunas de estas etnias se encuentran involucradas en procesos de mestizaje debido a desplazamientos de sus territorios ancestrales y la falta de conservación de su identidad cultural. En consecuencia, es necesario generar nuevo conocimiento sobre la diversidad y estructura genética de los pueblos indígenas de Colombia.
La información biológica sobre estos grupos étnicos será muy importante, no solo para explicar la estructura y diversidad genética de la población indígena colombiana, sino también para contribuir a esclarecer la dinámica del poblamiento de Colombia y Suramérica, puesto que, Colombia por su cercanía al istmo de Panamá, tuvo que ver con eventos migratorios y con el flujo genético ocurrido en este subcontinente (Barrantes, et al. , 1990).
Esta diversidad se puede caracterizar mediante la utilización de marcadores moleculares, con los cuales es posible obtener patrones biológicos que ayuden en la construcción de la estructura genética, en la identificación de las posibles rutas migratorias y en el reconocimiento de las dinámicas poblacionales humanas tales como cuellos de botella y deriva génica, entre otros (Sun, et al. , 2003).
Los marcadores genéticos en la región no recombinante del cromosoma Y (NRY), al producir un perfil haploide sin recombinación y transmitirse de padres a hijos varones, permiten observar el legado genético paterno preservado en la especie humana que persiste hoy en día (Gómez, et al. ,2008).
Dentro de estos marcadores, los polimorfismos de un solo nucleótido en el cromosoma Y (Y-SNP) hacen posible la identificación de grupos monofiléticos, pues su lenta tasa de mutación (10-8 mutaciones por generación) permite definir cada evento mutacional como único y, en consecuencia, una mutación en el mismo punto del ADN sería un evento raro en una población (Aitken, et al. , 2004). De esta manera, un conjunto de marcadores Y-SNPs pueden clasificar muestras humanas en haplogrupos, es decir, los linajes NRY definidos por polimorfismos binarios. Entre los grupos indígenas colombianos en general predomina el haplogrupo Q-M3, seguido del haplogrupo Q-M346 (Criollo-Rayo, 2012; Roewer, et al. , 2013; Braga, 2015 y Xavier, et al. , 2015), con trazas de Q-M19 en Ticunas y de P-M45, Q-M19 y C-M217 en Wayuus (Bortolini, et al., 2003 y Zegura, et al. , 2004).
Otro tipo de marcadores en el cromosoma Y son los microsatélites (Y-STR), cuya tasa de mutación (3,17x10-3 mutaciones por generación), mayor que la de los Y-SNPs (Kayser, et al. , 2000), los torna muy útiles en investigación, considerado su alto poder de discriminación, variación poblacional y facilidad de obtención y análisis. Además, por estar localizados en NRY, permiten reconstruir haplotipos que pueden ser rastreados a través de diferentes generaciones.
Dada la baja tasa de mutación en Y-SNP, se espera que los haplogrupos revelados sean grupos de cromosomas relacionados por descendencia, con un antepasado común; por el contario, los haplotipos definidos por Y-STR, al poseer una mayor tasa de mutación, pueden ser idénticos por descendencia o por estado, pudiendo presentar homoplasias (Díaz, 2010). En consecuencia, los haplogrupos al definir afinidad poblacional y los haplotipos al identificar patrones de diversidad dentro de grupos homogéneos, hacen de estos marcadores unas herramientas muy útiles en el estudio de poblaciones particularmente aisladas, étnicamente definidas y con tamaños efectivos pequeños como las etnias indígenas.
No obstante, a pesar de los estudios de diversidad genética encaminados a establecer frecuencias alélicas y haplotípicas de RFLPs en ADNmt y STRs en autosomas y el cromosoma Y para comunidades indígenas del occidente colombiano (Rondón, et al. , 1999; Mesa, et al. , 2000; Keyeux, et al. , 2002; Rodas, et al. , 2003; Carvajal-Carmona, et al. , 2000 y Franco, 2016), el conocimiento sobre la estructura genética de las poblaciones amerindias en esta región sigue siendo escaso y fragmentado, dificultando la definición de patrones integrales de relación filogenética entre las comunidades indígenas de esta región.
Con el objetivo de caracterizar la diversidad del cromo-soma Y en las poblaciones indígenas del occidente colombiano y teniendo en cuenta las características mencionadas para los Y-SNP y Y-STR, el presente estudio utiliza estos marcadores moleculares para incrementar la información genética patrilineal indígena en el Occidente de Colombia, dada la importancia del corredor andino en el poblamiento de Sudamérica, con lo que se logró reconocer sesgos de apareamiento en la población indígena mezclada.
En consecuencia, esta investigación hace una contribución importante a la resolución de preguntas sobre cómo está configurada la estructura genética y las relaciones filogenéticas entre los grupos indígenas del occidente colombiano, y cuáles son los posibles factores que influencian el flujo génico entre estos grupos humanos.
Materiales y métodos
Caracterización de la muestra. Fueron obtenidos 5 mL de sangre periférica de 131 hombres mayores de edad en buen estado de salud y en pleno uso de razón que voluntariamente decidieron participar en el presente estudio, sin parentesco entre ellos hasta la tercera generación, pertenecientes a seis comunidades indígenas en siete resguardos del occidente de Colombia, (Figura 1). A saber, 10 fueron Awakwaiker de Ricaurte-Nariño, 19 Coyaima de Planadas-Tolima, 12 Pijaos de El Guamo (La Luisa)-Tolima, 11 Emberá duma de Bahía Solano-Chocó, 30 Nasa (Páez) de Caldono-Cauca, 36 Nasa (Páez) de Jambaló-Cauca y 13 Pastos de Córdoba-Nariño.
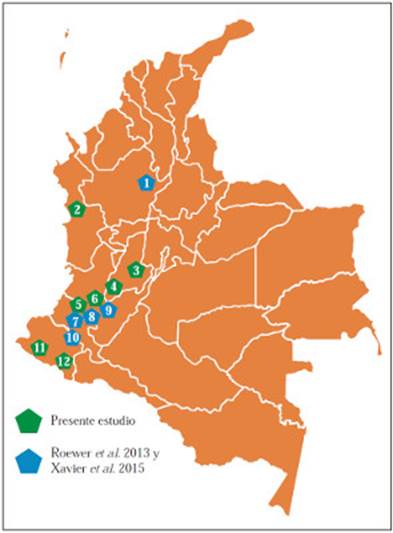
Figura 1 Ubicación geográfica de los centros de recolección de las muestras indígenas. (1) Emberá chamí, Segovia - Antioquia. (2) Emberá duma, Bahía Solano - Chocó. (3) Pijao, El Guamo - Tolima. (4) Coyaima, Planadas - Tolima. (5) Nasa, Caldono - Cauca. (6) Nasa, Jambaló - Cauca. (7) Nasa, Popayán - Cauca. (8) Coconuco, Puracé - Cauca. (9) Guambiano, Silvia - Cauca. (10) Yanacona, La Vega - Cauca. (11) Awakwaiker, Ricaurte - Nariño. (12) Pastos, Córdoba - Nariño.
El presente estudio se rigió por la resolución 8340 de 1993 del Ministerio de la Protección Social y contó con el aval del Comité Institucional de Revisión de Ética Humana (CIREH) de la Facultad de Salud de la Universidad del Valle, protegiendo la confidencialidad de los participantes. Todas las muestras se obtuvieron con consentimiento informado y firmado por cada participante, así como por el gobernador de cada cabildo indígena.
Las muestras de sangre periférica fueron almacenadas a 4 ºC con las respectivas medidas de bioseguridad. El ADN fue extraído utilizando el método de precipitación salina, “salting out”, ( Miller, et al. , 1988), cuantificado por espectrofotometría en un equipo NanoDrop 2000™ y almacenado a -20 °C.
Obtención de datos moleculares Y-SNP e Y-STR. Los datos moleculares utilizados en el presente estudio provienen de dos fuentes: los obtenidos directamente en nuestro laboratorio mediante el muestreo de 131 individuos y los resultados de 73 individuos tomados de la literatura, los cuales fueron comparables debido a la metodología de obtención de datos Y-SNP y Y-STR.
Los Y-STR en las muestras Nasa (Caldono y Jambaló) y Pastos (Córdoba) se obtuvieron utilizando la batería AmpFℓSTR® Yfiler™ (Applied biosystems). Ésta permite amplificar los loci DYS456, DYS389I, DYS390, DYS389II, DYS458, DYS19, DYS385a/b, DYS393, DYS391, DYS439, DYS635, DYS392, Y-GATA-H4, DYS437, DYS438 y DYS448 en una sola reacción de PCR (Pontes, et al. , 2006), la cual se ejecutó en un termociclador Veriti®, los fragmentos obtenidos fueron separados mediante electroforesis capilar en un analizador genético 3130 (Applied biosystems). La asignación alélica se hizo con el software GeneMapper™ V 3.2, comparando el peso de cada fragmento con su respectiva escalera alélica.
Los Y-STR de las muestras Awakwaiker (Ricaurte), Coyaima (Planadas), Pijao (El Guamo-La Luisa) y Emberá duma (Bahía Solano) se tomaron de un estudio previo reali-zado en nuestro laboratorio (Rondón, 2009).
Los haplogrupos Y-cromosómicos se obtuvieron al analizar 8 Y-SNP definidos por el Y Chromosome Consortium (YCC), (Karafet, et al. , 2008) que tipifica los principales linajes indígenas: Q-M242, Q1a2a1a1-M3, Q1a2a1a1a-M19, Q1a2a1a1b-M194, Q1a2a1a1c-M199-P292, Q1a2a1a1e-SA01 del haplogrupo Q y C-M130 del haplogrupo C (ISOGG, 2015). La tipificación de los Y-SNPs se llevó a cabo en las muestras Nasa (Caldono y Jambaló), Pastos (Córdoba), Awakwaiker (Ricaurte), Coyaima (Planadas), Pijao (El Guamo-La Luisa) y Emberá duma (Bahía Solano) mediante minisecuenciación, utilizando la batería ABI PRISM® SNaPshot™ Multiplex System (Applied Biosystems) con los cebadores diseñados por Geppert y colaboradores (2011).
También se utilizaron datos Y-STR obtenidos con Y-filer y datos Y-SNP producto de minisecuenciación de 26 hombres Emberá-Chamí de Segovia-Antioquia, 20 Guambianos (Misak) de Silvia-Cauca, 9 Coconucos de Puracé-Cauca, 12 Nasa (Páez) de Popayán-Cauca, 1 Pasto de Córdoba-Nariño y 5 Yanaconas de La Vega-Cauca reportados en artículos ya publicados de comunidades indígenas colombianas (Roewer, et al. , 2013; Xavier, et al. , 2015), (Figura 1), los datos de referencia y los del presente estudio son complementarios y comparables.
Análisis estadístico. A las muestras negativas para haplo-grupos amerindios se les infirió el haplogrupo a partir de sus haplotipos usando el programa YPredictor versión 1.5.0 (http://www.predictor.yADN.ru/), posteriormente se determinaron sus porcentajes con respecto a las muestras positivas.
La estructura genética de las muestras positivas para haplogrupos amerindios fue calculada a partir de la matriz de datos de microsatélites con el programa Arlequín versión 3.5.1.2 (Excoffier y Lischer, 2010), usando una AMOVA con estadísticos Rst, y un análisis de coordenadas principales (PCoA) a partir de las distancias genéticas corregidas de Nei empleando el programa GenAlEx versión 6.501 (Peakall & Smouse, 2012). Adicionalmente, se infirió la estructura genética espacial con el programa Geneland implementado en R (R Core Team, 2015), utilizando la matriz Y-STR en combinación con las coordenadas geográficas del lugar de nacimiento de cada individuo.
Las relaciones filogenéticas se exploraron con redes de expansión mínima a partir de los haplotipos Y-STR con el programa Network versión 4.6.1.3 (Bandelt, et al. , 1999), empleando el algoritmo de unión promedio (Median-Joining) y ponderándolo con la tasa de mutación de cada Y-STR. Además, se construyeron árboles de inferencia filogenética con el algoritmo Neighbor Joining (NJ) en el programa MEGA versión 6.0 (Tamura, et al. , 2013), utilizando como matriz de distancias los valores pareados RST.
Finalmente, la correlación de matrices se evaluó con la prueba de Mantel en el programa R versión 3.1.2 (R Core Team, 2015), definiendo la matriz de distancias genéticas como los valores pareados de RST linearizados de Slatkin en base a los Y-STRs, la matriz de distancias lingüísticas según la propuesta de Dow y Karunaratna en el 2006 e información cladística disponible (Vidal, 2011; Lewis, et al. , 2014), así como la matriz de distancias geográficas con base en la longitud lineal en Km entre los puntos de muestreo.
Resultados
Diversidad haplogrupal en el cromosoma Y. En las poblaciones indígenas muestreadas en el presente estudio fueron observados los haplogrupos Q1a2a1a1*-M3 y Q1a2*-M346 (M242, xM3), los cuales son los generalmente reportados como los más abundantes entre la población indígena (Tabla 1S, https://www.raccefyn.co/index.php/raccefyn/article/downloadSuppFile/476/2148). Los otros haplogrupos correspondieron a linajes de origen europeo (R), Africano (E) y otros como J, I, F, T y G. La muestra Pijao fue, en términos absolutos, la más pobre en linajes amerindios, seguida de Yanaconas y Coyaima, mientras que las muestras Nasa (Jambaló y Caldono), Emberas y Awa presentaron el mayor número de haplogrupos amerindios (Tabla 1).
Tabla 1 Valores absolutos de los haplogrupos Y cromosómicos en las muestras del presente estudio.
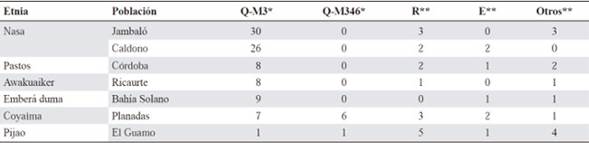
*Los haplogrupos Q1a2a1a1*-M3 (Q-M3) y Q1a2*-M346 (xM3) (Q-M242) se detectaron a través de Y-SNPs. **Los haplogrupos R, E y otros fueron predichos a partir de sus haplotipos Y-STRs.
Cuando se sumaron los resultados de las muestras colectadas en el presente estudio con los reportados en la literatura para el occidente colombiano (Roewer, et al., 2013; Xavier, et al., 2015), se observó que en la mayoría de los grupos indígenas estudiados se presentan porcentajes de haplogrupos no amerindios inferiores al 23%. En este análisis se destacan los Emberá chamí, con solo 8% de haplogrupos no amerindios, mientras que en el otro extremo se ubican los Pijao y Yanaconas, con el 83% y 60%, respectivamente (Figura 2).
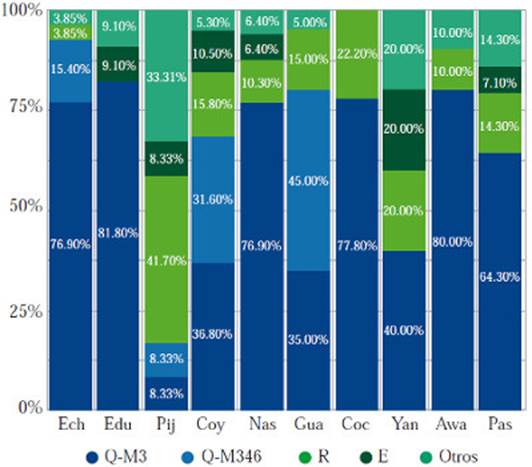
Figura 2 Porcentaje de haplogrupos Y-cromosómicos en los indígenas del occidente colombiano analizados. Los datos de Yanaconas (Yan, n=5), Emberá chamí (Ech, n=26), Coconuco (Coc, n=9) y Guambianos (Gua, n=20), doce datos de Nasa (Nas, n=78) y uno dato de Pastos (Pas, n=14) se tomaron de Roewer, et al. (2013) y Xavier, et al. (2015). 66 datos Nasa, 13 Pastos y las poblaciones Awakuaiker (Awa, n=13), Emberá-duma (Ebm, n=11), Pijao (Pij, n=12) y Coyaima (Coy, n=19), son datos del presente estudio. En la gráfica los etnias están ordenadas de norte a sur.
No se detectaron en ninguna de las poblaciones anali-zadas los haplogrupos Q1a2a1a1a-M19, Q1a2a1a1b-M194, Q1a2a1a1c-M199-P292, Q1a2a1a1e-SA01 ni C-M130 (Figura 2).
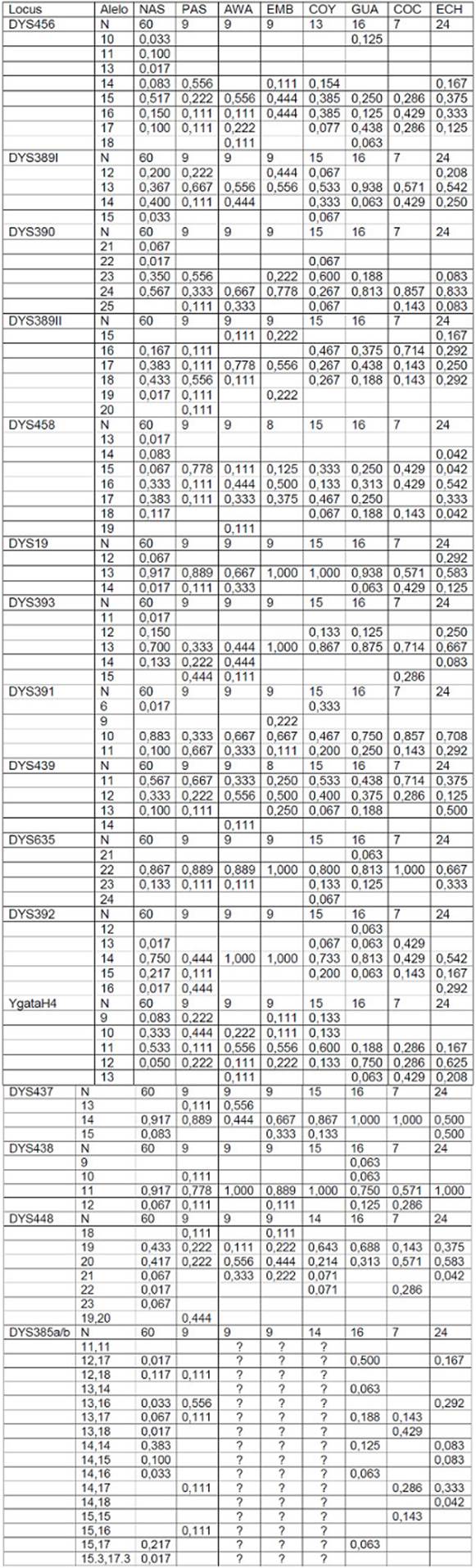
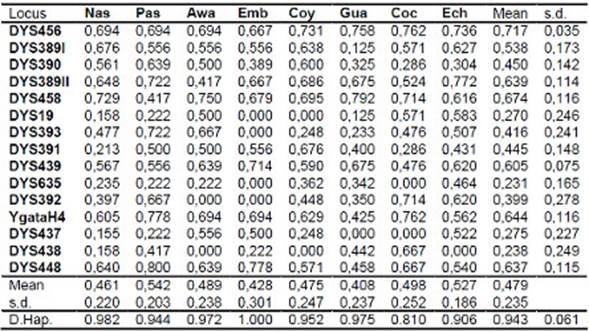
Diversidad genética patrilineal indígena. La distribución de las frecuencias alélicas de las 8 poblaciones amerindias del occidente colombiano es mostrada (Tabla 2S, https://www.raccefyn.co/index.php/raccefyn/article/downloadSuppFile/476/2149). El microsatélite con el mayor número de alelos fue DYS385a/b, los Y-STRs DYS19 y DYS437 fueron los de menor número de alelos. La heterocigosidad esperada promedio más baja fue la del DYS635 (0,231), el cual llegó a valores de cero en las comunidades Emberá duma y Coconuco, otros marcadores que de modo similar presentaron heterocigosidades bajas fueron DYS438 (0,238), DYS19 (0,270), DYS437 (0,275) y DYS392 (0,399) y la heterocigosidad más alta fue DYS456 (0,717), valor que no varió mucho entre las etnias (s.d. 0,035) (Tabla 3S, https://www.raccefyn.co/index.php/raccefyn/article/downloadSuppFile/476/2150).
Entre las muestras del presente estudio, la población de Pastos presentó dos haplotipos del haplogrupo Q1a2a1a1*- M3 con duplicaciones del Y-STR DYS448. A objeto de procesar los datos en los programas estadísticos, estos alelos se tomaron como perdidos.
Adicionalmente, fueron observados los siguientes alelos privados: en Pastos el alelo 20 de DYS389II, en Awakuaiker el 14 de DYS439 y el 19 de DYS458, en Emberá duma el 9 de DYS391, en Guambianos el 21 de DYS635, el 12 de DYS392 y 9 de DYS438, en Coyaimas el 24 de DYS635 y se destacan los Nasa con seis alelos privados, DYS456 alelo 11 y 13, DYS390 alelo 21, DYS458 alelo 13, DYS393 alelo 11 y DYS448 alelo 23 (Tabla 2S). Las muestras Yanaconas con origen amerindio no fueron analizadas, pues presentaron un número muy bajo de individuos como para ser significativos estadísticamente.
Estructura poblacional indígena. Para evaluar la estructura poblacional se propusieron tres hipótesis, en las cuales se agrupaban las poblaciones de muestras en base a su asignación (1) étnica, (2) lingüística y (3) departamental. Cuando se analizó la variación a través de AMOVA basada en la variación de los tamaños alélicos, se observó un mayor porcentaje de variación entre grupos cuando se usó la hipótesis 1 (10,8%), no obstante, ninguno de los valores RCT fue significativo, sin embargo, cuando se calcularon los valores de RST (0,15129) sí fueron significativos (resultados no mostrados).
En cuanto a los valores pareados de RST, estos presentan en general un nivel bajo de estructura. Sin embargo, solo algunos son significativos. Así, los Awakwaiker, Coconuco y Guambiano no presentan diferencias significativas entre ellos; de modo similar, Emberá duma, Guambiano y Coyaima; finalmente, Emberá chamí no presenta diferencias con Guambiano; ni los Nasa con Emberá duma. De esta forma, Pastos es el único grupo en presentar diferencias significati-vas con todos los otros grupos indígenas analizados (Tabla 2).
Tabla 2 Matriz de valores RST pareados y sus valores p con grupos por etnias.
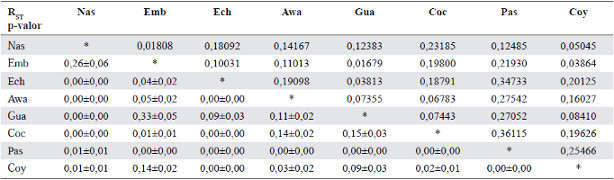
Nas = Nasa, Pas = Pastos, Awa = Awakwaiker, Emb = Emberá duma, Coy = Coyaima, Gua = Guambiano, Coc = Coconuco y Ech = Emberá chamí.
A través de un modelo de frecuencias alélicas no correlacionadas (Tabla 4S, https://www.raccefyn.co/index.php/raccefyn/article/downloadSuppFile/476/2151), se observó en el análisis espacial bayesiano que las muestras se dividen en un número K de poblaciones igual a dos, (Figura 3A), valor que presentó el mayor índice de densidad de iteraciones de cadenas de Makornikov - Monte Carlo (MCMC). En esta división, las muestras Pastos conformaron un grupo aparte de las demás muestras, (Figuras 3B y 3C), confirmando los resultados presentados en la Tabla 2.
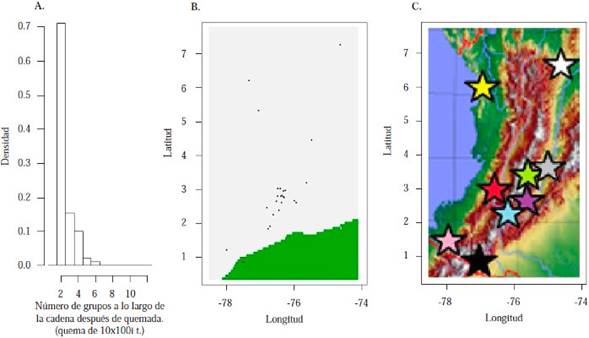
Figura 3 Agrupamiento espacial bayesiano de muestras con haplotipo amerindio. A) Mapa orográfico de Colombia con la ubicación de grupos indígenas: Nasa (rojo), Pastos (negro), Awakwaiker (rosa), Emberá duma (amarillo), Coyaima (verde), Pijao (Gris), Guambiano (violeta), Coconuco (azul) y Emberá chamí (blanco). B) Diagrama de Voronoi, donde cada punto representa el lugar de nacimiento de cada indígena analizado en el occidente de Colombia, algunos puntos pueden superponerse. C) Gráfica de densidad de probabilidad contra número poblaciones por el método Makornikov - Monte Carlo, it = iteraciones.
A partir de las distancias corregidas de Nei entre los grupos étnicos, se elaboró un análisis de coordenadas principales (PCoA), en el cual se visualiza la formación de un grupo conformado por Awakwaiker, Coyaima, Nasa y Emberá duma, y otro por Guambiano, Coconuco y Emberá chamí, la población de Pastos se encuentra aislada. Las dos coordenadas de esta gráfica explican el 73,19% de la variación genética (Figura 4).

Figura 4 Análisis de coordenadas principales (PCoA) de la variación genética (unbiased Nei distance) entre los grupos étnicos. La coordenada 1 explica el 49,86% de la variación, la coordenada 2 el 23,33%, para un total de 73,19% de variación explicada. Nas = Nasa, Pas = Pastos, Awa = Awakuaiker, Emb = Emberá duma, Coy = Coyaima, Gua = Guambiano, Coc = Coconuco y Ech = Emberá chamí.
El árbol NJ inferido a partir de la matriz de RST pareados, presenta a Emberá duma en la parte central de la gráfica en donde se agrupan con Coyaima y Nasa, Emberá duma también se relaciona con otro grupo conformado por dos ramas, una con Awakuaiker y Coconuco y otra con Guambiano y Emberá chamí; los Pastos, por su parte, se sitúan a una distancia mayor del grupo descrito, concordando con los análisis anteriores (Figura 5).
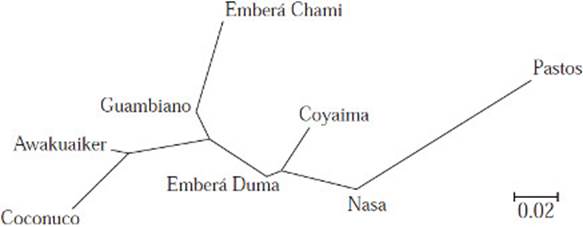
Figura 5 Árbol filogenético NJ a partir de una matriz RST en base a Y-STRs entre los grupos étnicos.
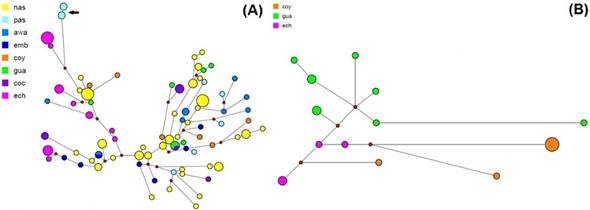
Al examinar la red expansión mínima de las muestras con haplogrupo Q1a2a1a1*-M3, se puede observar que los individuos Pastos que portan una duplicación del Y-STR DYS448 se separan del grupo central por cerca de seis mutaciones. Los haplotipos de Emberá chamí se encuentra principalmente en dos ramas, del mismo modo los Awakwaiker, pero al lado opuesto de la gráfica. Los Coconuco se muestran relacionados a los Guambianos, el resto de las etnias se encuentran dispersos a lo largo de la red (Figura 1S, https://www.raccefyn.co/index.php/raccefyn/article/downloadSuppFile/476/2146).
La red de expansión mínima de haplotipos con haplo-grupo Q1a2*-M346 (M242, xM3), muestra un evento de expansión en los datos Guambianos, los haplotipos Coyaima parecen estar más relacionados con los Emberá chamí que con los Guambianos (Figura 1S).
Correlación de matrices de distancia. El test de Mantel para correlacionar las distancias geográficas y genéticas dio una correlación de r = 0,154 con p-valor = 0,296, y entre las distancias lingüísticas y genéticas un r = 0,004878 y p-valor = 0,445, por lo tanto, la estructura presente en las muestras puede deberse a factores diferentes de los lingüísticos o de la geografía plana (sin considerar la orografía).
Discusión
El presente estudio obtuvo datos de 10 grupos indígenas, sin embargo, dos de estos, los Pijaos y los Yanaconas, presentaron un número absoluto muy bajo de hombres con haplogrupo Y cromosómico amerindio, por lo cual no se analizaron más allá de su diversidad haplogrupal.
Composición haplogrupal indígena. Las altas frecuencias del haplogrupo Q1a2a1a1*-M3 (antes Q1a3a*), evidencia el origen amerindio de las muestras poblacionales en cuanto a la línea paterna se refiere (Tabla 1), pues este haplogrupo es el principalmente hallado en nativos de Sudamérica, comportamiento ya reportado (Roewer, et al. , 2013; Xavier, et al. , 2015). Entre las muestras Coyaima y Pijao siete individuos fueron positivos para Q-M242 y negativos para Q1a2a1a1-M3, estos individuos muy probablemente son Q1a2*-M346 (antes Q1a3*), puesto que no se encontraron reportes de Q*-M242 en Sudamérica y sí de Q1a2*-M346, además de existir un estudio previo en Pijaos de los municipios de Coyaima y Ortega en Tolima que reporta a Q1a2a1a1*-M3 y Q1a2*-M346 como los únicos linajes del haplogrupo Q presentes en estas muestras (Criollo-Rayo, 2012). Sin embargo, no se tiene confirmación molecular, dado que el SNP M346 no formó parte de los SNPs evaluados en el presente estudio, y por tanto nos referimos a estos individuos como Q1a2*-M346 (M242, xM3). En las poblaciones del equipo de Roewer (2013) sí se presenta confirmación molecular de Q1a2*-M346 en Emberá chamí y Guambiano, siendo en este último el haplogrupo más frecuente (Figura 2).
El origen asiático en los indígenas de Colombia. Los reportes de Q1a2*-M346 en diversos grupos indígenas de Colombia (Bortolini, et al., 2003, Roewer, et al., 2013, Criollo-Rayo, 2012 y Braga, 2015), incluido el presente estudio (Q1a2*-M346 (M242, xM3)), México, Perú y Bolivia (Sandoval, et al., 2012), apoyan la hipótesis del origen asiático en una sola migración de la población amerindia con haplogrupo Q, sustentada por la presencia de este haplogrupo en el sur de Siberia pero ausente en poblaciones del este de Asia (Mongoles y Coreanos) y en muy baja frecuencia en poblaciones del este de Europa (Calmucos), según Malyarchuk y colaboradores (2011).
En el presente estudio no se encontró cromosomas con subhaplogrupos del Q1a2a1a1-M3, los cuales están asociados en bajas frecuencias a poblaciones andinas, especialmente en Perú, los Y-SNP M194, M199 y P292 solo se han reportado cada uno en un individuo (Battaglia, et al. , 2013 y Geppert, et al. , 2015), lo que podría explicar la escasa probabilidad de encontrarlos en el presente estudio. El Y-SNP SA01 reportado en poblaciones andinas peruanas (Quechua y Arawak) y bolivianas (Aymara) (Geppert, et al. , 2015), es posiblemente ocasionado por mutaciones muy recientes, por lo que su presencia podría estar restringida a localidades más australes; y puede que el Y-SNP M19 detectado en indígenas colombianos Ticuna y Wayuu (Geppert, et al. , 2015), se encuentre asociado con poblaciones más al oriente de Colombia.
Tampoco se observó en el presente estudio el haplogrupo C-M130, por lo que la presencia C2*-M217 en los indígenas Waonari y Kichwa de Ecuador, los Wayuu de la frontera entre Colombia y Venezuela, y en las poblaciones americanas Navajo y Apache Na-Dené (Zegura, et al. , 2008; Geppert, et al. , 2011), sugeriría que pudo haber arribado de forma independiente a estos sitios, lo que refuerza la hipótesis de una migración marítima de este haplotipo hacia las Américas.
Miscegenación en las muestras indígenas. Haplogrupos no amerindios del cromosoma Y se observaron en todas las etnias evaluadas, sin embargo, poblaciones con pérdida total o parcial de la tradición lingüística como los Pastos, Yanaconas y Pijaos, presentan un incremento en la diversidad haplogrupal con cromosomas no amerindios en mayor medida que aquellas poblaciones indígenas que conservan su tradición lingüística como los Nasa o Emberás que presentan menos mezcla, en donde el aislamiento disminuye los niveles de diversidad y causa diferenciación de las poblaciones. La incursión de los haplogrupos no amerindios en las comunidades indígenas puede deberse a la influencia que tienen en el flujo génico los cascos urbanos asentados en sus cercanías. Un ejemplo ya reportado de esta situación es el de la población mestiza de Ibagué que presenta un flujo génico con los Pijao (Rondón, 2009), efecto que es menos pronunciado en los Coyaimas.
Las poblaciones indígenas evaluadas presentaron porcentajes variables, que van desde el 4% hasta el 20% de cromosomas que no obedecen a los haplogrupos R o E (bajo el predictor de haplogrupos), gran parte de estos se han referenciado en estudios anteriores como haplogrupos J e I de origen euroasiático en poblaciones Nasa, Pijao y Emberás. Se piensa que estos cromosomas incursionaron junto con los R1b en el arribo español a las américas, puesto que en las poblaciones españolas ya se había generado un contacto con poblaciones de musulmanes, moros, judíos y norafricanos durante el medioevo europeo (Criollo-Rayo, 2012).
Estructura genética en el cromosoma Y. La fijación de determinados alelos en estas poblaciones puede ser ocasionada por la deriva génica, en donde los efectos de cuello de botella y efectos de fundador configuran los diversos linajes étnicos, convirtiendo estos alelos en marcadores Y-cromosómicos amerindios en las poblaciones indígenas colombianas en el occidente del país. Esto como posible consecuencia del pequeño tamaño de la población fundadora que colonizó el territorio colombiano y a la reducción de la población indígena durante el contacto español.
Es de tener en cuenta que la diversidad haplotípica de todas las comunidades indígenas fue alta, desde 0,810 en Coconucos hasta 1,0 en Emberá duma, sin variar demasiado, lo cual podría ser efecto de tamaños muestrales pequeños. Sin embargo, no es el caso para poblaciones como los Nasa del presente estudio y los Emberá chamí Roewer, et al. , (2013), cuyas muestras fueron numerosas y aun así presentaron altas diversidades haplotípicas, indicando que las diferencias en el tamaño muestral presentan un efecto bajo en los resultados.
La prueba de AMOVA muestra un nivel bajo de subestructura genética, indicando gran flujo génico entre poblaciones indígenas, una posible consecuencia del colapso total de la población nativa por el contacto europeo, en donde muchos grupos desaparecieron por completo y ocurrieron fusiones de etnias además de la mezcla con no indígenas (Mulligan, et al. , 2004).
Todas las pruebas que intentan inferir el tipo de estructura entre las etnias estudiadas (Tabla 2) (Figuras 3, 4 y 5) muestran que los Pastos se encuentran aislados genéticamente de los demás grupos analizados. Estas diferencias poblacionales parecen no deberse a factores lingüísticos o de distancia geográfica plana (sin orografía), dado que la comparación de matrices no arrojó resultados significativos. Tanto en PCoA como en NJ (Figuras 4 y 5) se forman dos grupos centrales, el primero con Coyaima, Emberá duma y Nasa, en otros trabajos similares ya se había reportado una cercanía genética entre Nasa y Pijaos y estos a su vez con los Emberá katío (Criollo-Rayo, 2012), el segundo grupo con Guambiano, Emberá chamí y Coconuco, este grupo se encuentra más alejado de la costa Pacífica que el primero; La posición de los Awakuaiker cambia de acuerdo al tipo de análisis, en el PCoA está asociado con el primer grupo y en el NJ está asociado con el segundo, por lo que no se da en este punto una conclusión, pero es de tener en cuenta que perteneciendo al segundo concuerda con su cercanía lingüística con Coconuco y Guambiano.
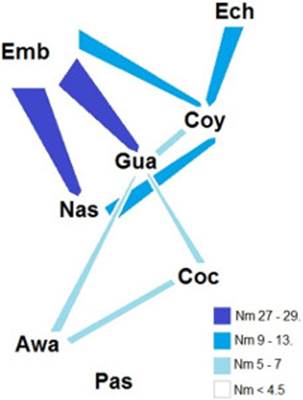
Flujo de genes entre las comunidades del occidente. Se observó una coincidencia entre el flujo génico (número efectivo de migrantes) con la orografía colombiana, lo cual podría explicar las relaciones genéticas observadas en este estudio (Figura 2S, https://www.raccefyn.co/index.php/raccefyn/article/downloadSuppFile/476/2147). De esta forma, se evidencia una tendencia de norte a sur en los números efectivos de migrantes, destacando que el flujo entre los Emberá chamí y los Emberá duma es muy bajo a pesar de estar ubicados en el norte del país y de ser comunidades lingüísticamente cercanas, pues los dialectos de la lengua emberá son ubicados en la familia Chocó; sin embargo, entre sus posiciones geográficas se encuentran las cordilleras occidental y central, que actuarían como una barrera física del flujo génico. El hecho de que el flujo génico de estas poblaciones se encuentre influenciado por esta barrera geográfica, es contrastante con las comunidades indígenas amazónicas colombianas en el valle geográfico, en las cuales la exogamia lingüística tiene un efecto mayor en su flujo génico que la geografía (Braga, et al. , 2012).
Los Emberá duma se comunicarían con las poblaciones Coyaima, Guambiano y Nasa por la costa Pacífica para luego conectar con Tierradentro, los Emberá chamí conectarían con los Coyaima y Guambianos por el valle entre las cordilleras Central y Oriental, resaltando que estas poblaciones son las únicas en presentas el haplogrupo Q1a2*-M346 (M242, xM3), sugiriendo que fue por esta vía su migración hacia el sur. La comunicación de Guambiano, Coconuco y Awakwaiker, sería por las faldas de la cordillera de los Andes del lado occidental, los Pastos estarían aislados dado que se encuentra en el lado opuesto de la cordillera andina, es decir, el oriental.
Este comportamiento de las comunidades indígenas andinas, difiere del de las comunidades amazónicas del Vaupés, en las cuales las uniones maritales se llevan a cabo entre hablantes de lenguas indígenas distintas, incrementando la diversidad genética a través de este particular flujo génico, uno asimétrico, en el cual las mujeres son migrantes y los hombres se asientan en su territorio (Braga, et al. , 2012).
Redes de expansión mínima. Al realizar el análisis de redes de expansión mínima por el algoritmo de unión promedio para los haplotipos del haplogrupo Q1a2a1a1*-M3, parte de la población de Pastos, parte de Nasa y parte de Emberá chamí se alejaron del grupo central en cerca de 6 pasos mutacionales; estos individuos portaban alelos privados diferentes según la etnia (Figura 1S), y se destacan los Pastos con la duplicación de DYS448, donde estas y otras diferencias genéticas que los aíslan de los demás grupos analizados, pueden deberse a contactos precolombinos con grupos indígenas fuera de Colombia, de lo cual existe referencia histórica, asociándolos al imperio Inca, dado que compartían una frontera y estuvieron fuertemente influenciados por estos (Kloosterman, 1997). Por lo tanto, el flujo génico, y quizás el origen del grupo Pastos, no provenga del norte del conti-nente sino del sur.
En la red de expansión mínima de Q1a2*-M346 (M242, xM3) se observó que los haplotipos Coyaima se relacionan más con los Emberá chamí que con los Guambianos (Figura 1S), consecuencia quizás del flujo génico direccionado de norte a sur del que se habló anteriormente.
Contraste del yADN con el mtADN. Finalmente, al comparar los resultados de la diversidad haplogrupal del cromosoma Y en estas poblaciones con los datos mitocondriales sobre estas mismas en otros estudios, se observa que el porcentaje de mitocondrias no amerindias es menor al de cromosomas Y no amerindios, comparando cada población con su par, la excepción a esta observación sería con los Emberá duma en los que fue al contrario y a su vez forma parte del grupo con influencia mitocondrial africana, a saber, Awakuaiker, Pijao del Guamo (La Luisa), Coyaima de Planadas y Emberá duma (Rondón, 2009).
Con respecto a estos, sus contrapartes patrilineales también presentan mezcla de origen africano, excepto Awakuaiker. En los Nasa, Pastos, Emberá chamí y Guambiano, el ADN mitocondrial fue exclusivamente amerindio (Tosse, 2014; Xavier, et al., 2015), en cambio sus contrapartes cromosómicas-Y presentaron mezcla, especialmente los Pastos. En Coconucos y Yanaconas hubo mezcla mitocondrial no amerindia, pero en porcentajes inferiores al 6% (Xavier, et al. , 2015), porcentaje mucho menor que en su contraparte en el Y, con lo cual se podría concluir que en la mayoría de las poblaciones indígenas analizadas se presenta un sesgo de apareamiento, siendo éste más marcado en los Pijaos (Y amerindios 8%; mtADN amerindio 91%), comportamiento de los grupos indígenas americanos previamente reportado (Rodas, et al. , 2003; Novembre & Ramachandran, 2011), la gran excepción fueron los Emberá duma (cromosoma Y amerindio 82%; mtADN amerindio 79%).
El comportamiento migratorio de los linajes masculinos puede diferir de los femeninos, por lo cual se esperaría que al efectuar análisis similares a los del presente estudio en la herencia matrilineal de las muestras, el resultado de las relaciones filogenéticas podría ser diferente al de su contraparte patrilineal.
Conclusiones
El haplogrupo observado con mayor frecuencia (58% en promedio) en la muestra estudiada fue el Q1a2a1a1*-M3. Exceptuando Pijao y Coyaima los demás grupos indígenas presentaron en promedio una frecuencia inferior al 32% de haplogrupos no amerindios.
Los centros urbanos cercanos influencian la composición Y-haplogrupal de los grupos indígenas, lo que podría haber contribuido en el detrimento lingüístico nativo en aquellas comunidades con mayor grado de mezcla, nuevas investigaciones deben ser realizadas para su confirmación.
Variantes alélicas Y-STRs se sugieren como representativas de los haplogrupos indígenas del occidente colombiano para las muestras analizadas dado que en estos marcadores se presenta baja diversidad génica.
Los grupos indígenas del occidente tienen una estructura genética baja debido al flujo génico dado entre ellas, estando los Pastos aislados de los demás grupos indígenas analizados.
La posibilidad que la estructura genética y el flujo génico se encuentren regulados por la orografía en los grupos indígenas del occidente colombiano debe considerarse en futuras investigaciones. Los resultados aquí presentados sugieren la posible acción de las cordilleras como barreras físicas al flujo de migrantes.
Los Pastos, aislados genéticamente de otros indígenas, podrían estar relacionados con descendientes de los incas, dado su contacto precolombino.
Finalmente, se confirma el sesgo de apareamiento que tiende a relacionar cromosomas Y no amerindios con ADN mitocondrial amerindio en la parte mezclada de los grupos indígenas analizados, excepto con Emberá duma donde ocurre lo contrario.