Introducción
En Colombia, el maíz (Zea mays L.) es el tercer cultivo con mayor superficie de siembra después del café y el arroz. En las últimas décadas la producción de maíz en Colombia presentó un incremento de 76 %, entre 1961 y 2016. A pesar de ello, es el país con mayor volumen de importaciones en Suramérica y el séptimo en el mundo, cubriendo alrededor del 74 % de la demanda interna de maíz con importaciones (Govaerts et al., 2019). El cultivo de maíz en Colombia se desarrolla en dos tipos de sistemas productivos que corresponden al sistema productivo tradicional y al tecnificado. El 84.5 % del sistema tecnificado ha adoptado el uso de semilla híbrida. De acuerdo con algunas experiencias en el Centro Internacional de Mejoramiento de Maíz y Trigo (CIMMYT), los híbridos mejorados pueden alcanzar rendimientos entre 37 y 105 % más en relación con las variedades regionales, y en el Valle del Cauca se han alcanzado rendimientos de hasta 8.5 t/ha. Así, con el objetivo de aumentar la producción de maíz en Colombia, los híbridos representan el estándar que se quiere alcanzar en términos de producción y calidad. Esto implica un proceso de mejoramiento genético para la generación de líneas parentales, formación de híbridos experimentales, su evaluación a través de diferentes ambientes y la selección de los mejores híbridos (Govaerts et al., 2019).
La clasificación de grupos heteróticos es importante para los programas de mejoramiento genético del maíz híbrido (Zea mays L.)(Fan et al., 2018), siendo clave para tener una mayor eficiencia ya que permite reducir las probabilidades de evaluar híbridos con características indeseables. La eficiencia en la asignación de grupos heteróticos a las líneas endocriadas es necesario para obtener un patrón heterótico útil para el esquema de mejoramiento genético usado (Oyetunde et al., 2020).
Existen diversos métodos para clasificar líneas parentales en grupos heteróticos, incluyendo el uso de rasgos morfológicos, método de pedigrí, técnicas multivariadas, métodos genéticos incluyendo diseños de apareamientos como los cruzamientos dialélicos y cruzamientos línea x probador, y el uso de marcadores moleculares (Olutayo, 2021). Varios autores han reportado la eficiencia en la determinación del patrón heterótico en una población a partir de datos fenotípicos y de datos genotípicos (Adu et al., 2019). La habilidad combinatoria específica (HCE) para el rendimiento de grano y la habilidad combinatoria general (HCG) son usadas para clasificar líneas parentales en grupos heteróticos, sin embargo, el rendimiento de grano es un carácter complejo y de baja heredabilidad, por lo cual puede ser necesario hacer análisis comparativos que permitan determinar la metodología más eficiente (Mahato et al., 2021). Otros autores han evaluado las dos metodologías en conjunto (marcadores moleculares y cruces dialélicos) para establecer la utilidad de cada una de ellas (Aguiar et al., 2008). Sin embargo, pocos estudios reportan el uso de SNPs y ensayos dialélicos en la determinación de grupos heteróticos. Por lo anterior, aplicar la metodología de clasificación de grupos heteróticos a partir de SNPs en comparación con los resultados obtenidos a partir de ensayos de tipo dialelo, que están basados en las habilidades combinatorias de las líneas evaluadas, puede proveer información importante acerca de la utilidad de estos marcadores con este fin. Por tanto, el objetivo del presente estudio fue comparar la consistencia de la clasificación de grupos heteróticos definidos mediante el ensayo dialélico y marcadores moleculares de tipo SNP para identificar los grupos heteróticos en las líneas tropicales de maíz estudiadas.
Materiales y métodos
Selección de parentales y formación de híbridos
En el ensayo se incluyeron 30 líneas élite del programa de mejoramiento de Semillas Valle S.A. como parentales, incluyendo líneas de grano color blanco y amarillo. Se realizaron cruzamientos entre los 30 parentales, sin incluir los recíprocos, de acuerdo con el método II descrito por Griffing (1956), para un total de 435 cruzamientos [ (P(P-1))/2] y los 30 parentales. La obtención de semilla se realizó en el Centro Experimental de Semillas Valle S.A. ubicado en el municipio de El Cerrito, Valle del Cauca, Colombia.
Evaluación de híbridos y análisis de datos
Los 435 híbridos simples y los 30 parentales fueron evaluados junto con 4 híbridos comerciales como testigos en un diseño de bloques aumentados. Los ensayos fueron llevados a cabo en dos semestres consecutivos, 2018A y 2018B, en dos localidades que correspondían a la finca Estambul, en el municipio de Roldanillo, Valle del Cauca (4°27’05” LN 76°04’53” LO), y al Centro Experimental de Semillas Valle S.A. en el municipio de El Cerrito, Valle del Cauca (3°41’26.3” LN 76°18’35.7” LO). Cada unidad experimental consistió en dos surcos de 5 metros de longitud, con distanciamiento de 0.8 m entre surcos y callejones frontales y posteriores de 2 m y 0.8 m respectivamente. En cada localidad se evaluaron caracteres fenotípicos, incluyendo los componentes de rendimiento y sanidad más importantes para el programa de mejoramiento.
Para cada ensayo se realizó un análisis de varianza, con el fin de usar, posteriormente, las variables ajustadas en un análisis combinado. Para el análisis combinado los datos conforman un conjunto de F1, con parentales, y sin recíprocos. Estos fueron analizados usando el aplicativo de CIMMYT AGD-R (Analysis of Genetic Designs with R for Windows) versión 5.0, de acuerdo con el método 2 propuesto por Griffing, utilizando un modelo mixto (Modelo B) en donde los genotipos son fijos y los bloques aleatorios. Los ensayos se utilizaron como bloques para un total de 4 repeticiones.
Adicionalmente se determinaron: Habilidad Combinatoria General (HCGi), Habilidad Combinatoria Específica (HCEi), Heterosis, Varianza Aditiva (σ2A), Varianza de Dominancia (σ2D), Varianza fenotípica (σ2p) y Heredabilidad en sentido amplio (H2) y estrecho (h2).
Clasificación de líneas en grupos heteróticos
Aplicando la aproximación propuesta por Badu-Apraku et al. (2013), se clasificaron las líneas con base en la HCG de múltiples rasgos (HGCAMT, Heterotic grouping based on GCA of multiple traits). Para esto, se realizó una estandarización de los efectos de la HCG de los rasgos evaluados, para minimizar los efectos de las diferentes escalas de medición. Se incluyeron las variables altura de planta y de mazorca, floración masculina y femenina, aspecto de mazorca, color y tipo de grano, y rendimiento, rasgos que presentaron efectos aditivos significativos en los ambientes evaluados. Una vez estandarizados los valores, se realizó un análisis clúster con el método de Ward de varianza mínima, usando el software estadístico RStudio, y se generó un dendrograma basado en el agrupamiento. Se incluyó el origen de las líneas de acuerdo con la información del programa de mejoramiento, para establecer si el origen geográfico está relacionado con el agrupamiento en grupos heteróticos.
Secuenciación de líneas parentales
66 líneas del programa de Semillas Valle S.A. fueron seleccionadas por ser ampliamente usadas en la formación de híbridos. 16 de estas líneas, parentales de híbridos sobresalientes, fueron secuenciadas por duplicado, obteniendo datos para 82 genotipos, incluyendo 47 líneas amarillas y 35 líneas blancas. La genotipificación se realizó mediante el arreglo de genotipificación MaizeSNP50 BeadChip. Para ello, muestras de semillas fueron enviadas al proveedor Eurofins BioDiagnostics para la extracción de ADN y su genotipificación.
Análisis de diversidad genética
Se seleccionó la fracción de SNPs informativos (segregantes), es decir los SNPs con una frecuencia de alelo menor (MFA)>0.05 y con datos faltantes en menos del 20 % de la población, después de realizar las imputaciones plausibles en la población, usando el software NGSEP (v. 4.1.0.)(Duitama et al., 2014). Los datos genotípicos filtrados se utilizaron para realizar los análisis de diversidad genética.
La matriz de SNPs se utilizó para evaluar la estructura genética en la población de líneas por medio de un análisis de componentes principales (PCA) empleando el software TASSEL (v. 5.2.73) (Bradbury et al., 2007), y posteriormente se realizó una gráfica de tres dimensiones implementando la herramienta XLSTAT (v. 2018) (Addinsoft) para mostrar gráficamente la distribución de los grupos, asignando colores a los puntos de acuerdo con el origen de las líneas. La estructura de la población fue estimada usando un método de asignación poblacional basado en un modelo Bayesiano de Cadenas de Markov Montecarlo (MCMC), implementando el modelo de frecuencias alélicas independientes en el software STRUCTURE (v. 2.3.4). Se realizaron 10 corridas para cada número de poblaciones (K) establecido de 1 a 10. El tiempo de calentamiento y el número de replicaciones de MCMC se fijaron en 10 000 para cada ejecución. El valor K más probable fue determinado con la herramienta en línea Structure Harvester (Earl y vonHoldt, 2012), utilizando la probabilidad logarítmica de los datos [LnP(D) ] y delta K (ΔK) en función de la tasa de cambio en [LnP(D) ] entre valores K sucesivos. Adicionalmente, se calculó el estadístico FST como medida de la heterogeneidad de la población y el grado de separación que ha ocurrido dentro de la población. A partir de los grupos definidos en STRUCTURE se calcularon los valores de FST entre los pares de grupos, usando las librerías Hierfstat (v. 0.5-11) ) (Weir y Goudet, 2017) y Adegenet (v.1.3-1) )(Jombart y Ahmed, 2011) en R, así como la heterocigosidad observada (HO), la diversidad genética promedio (HS), y el índice de fijación FIS para cada subpoblación (Wright, 1965). También se construyó un árbol de unión de vecinos no enraizado en la herramienta en línea iTol (v. 5), con base en la matriz de distancias obtenida con TASSEL (v. 5.2.73), que calcula la distancia como 1-Similitud de Identidad por Estado (IBS, Identity by State), con IBS definido como la probabilidad de que los alelos extraídos al azar de dos individuos sean iguales en el mismo locus.
Comparación de las metodologías evaluadas
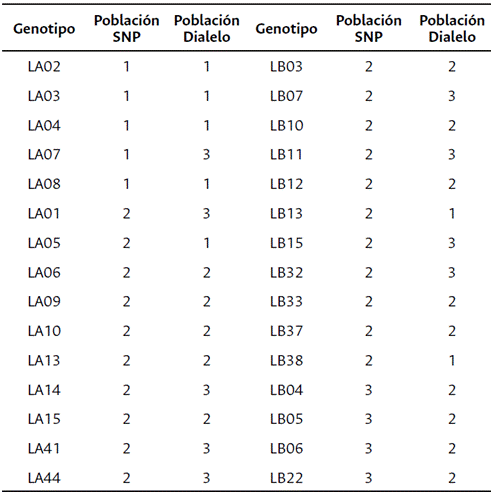
Una vez establecidos los grupos heteróticos, se realizó una comparación de la asignación de genotipos en las metodologías propuestas, con el fin de establecer si hay coincidencia en la formación de grupos. La eficiencia del agrupamiento fue determinada como el porcentaje de líneas clasificadas de forma diferencial en cada metodología (Oyetunde et al., 2020).
Resultados
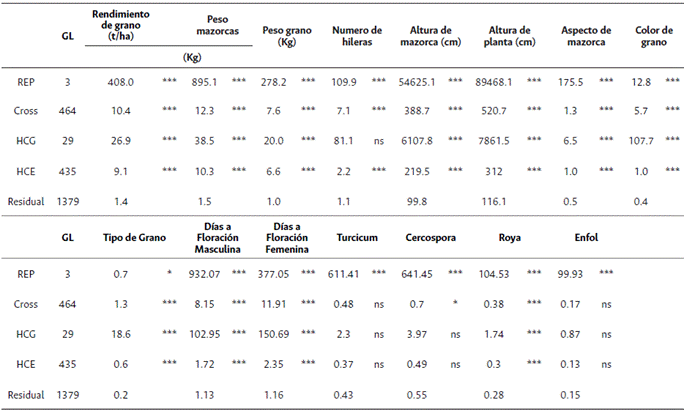
En el análisis de varianza combinado (Tabla suplementaria 1) del ensayo de dialelo se observó que los factores de variación, repetición y cruzamiento fueron significativos para todas las variables evaluadas, excepto para las enfermedades foliares en prefloración y tizón foliar, indicando que el comportamiento de los híbridos fue altamente influenciado por las condiciones ambientales, y que la respuesta a este es diferente según el genotipo. También se observaron cuadrados medios significativos para la HCG y la HCE para los componentes de rendimiento y las variables de planta, excepto para el número de hileras (Tablas suplementarias 2 y 3).
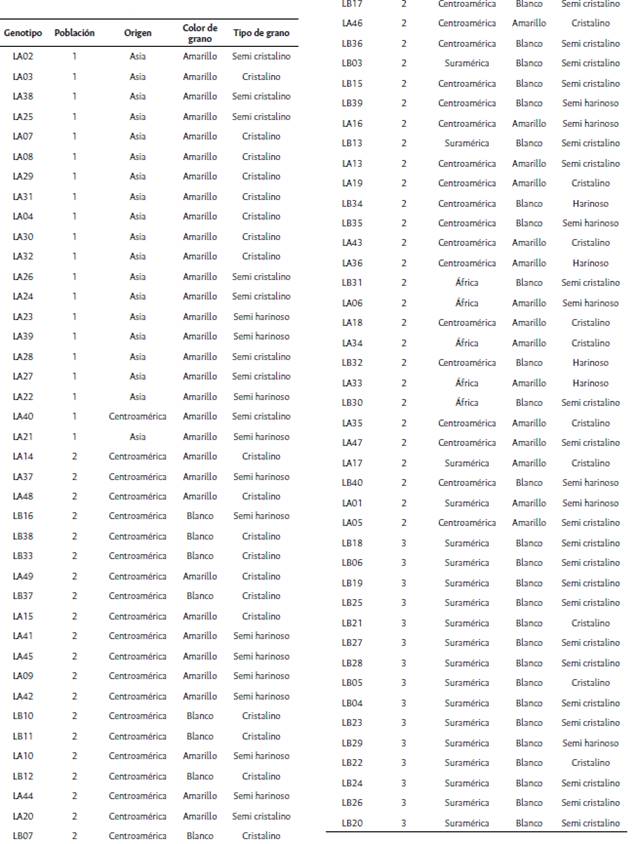
A partir de la metodología de HGCAMT, aplicando el método de agrupación jerárquica de Ward, que permite formar grupos mutuamente excluyentes con base en la similaridad en las características especificadas (Ward, 1963), se obtuvo el dendrograma presentando en la Figura 1. De acuerdo con esta clasificación, se observan 3 grupos heteróticos compuestos por 7, 14 y 9 líneas, respectivamente. En el primer clúster se encuentran agrupadas líneas de origen asiático; en el clúster 2 las líneas de Suramérica, excepto una, junto con líneas de Centroamérica y África; y en el tercer clúster se encuentran líneas predominantemente de Centroamérica.
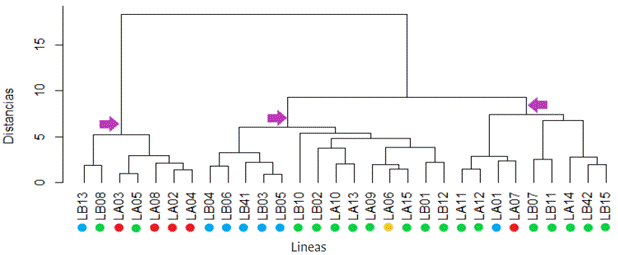
Figura 1 Dendrograma de 30 líneas de maíz tropical a partir de la HCG del rendimiento de grano y otros rasgos, usando el análisis clúster de mínima varianza de Ward. Los puntos de colores representan el origen de las líneas: azul=Suramérica; verde=Centroamérica; rojo=Asia; amarillo=África.
A partir del chip de Ilumina MaizeSNP50 BeadChip se observaron un total de 55 542 SNPs en las 66 líneas endocriadas. Luego de filtrar los datos, 12 209 SNPs resultaron informativos para el análisis de estructura poblacional. Con base en las distancias genéticas de Nei (1972) se realizó un análisis de coordenadas principales con el fin de ilustrar los ejes principales de la variabilidad entre las líneas evaluadas, plasmándola en una ordenación espacial (Figura 2). Las tres primeras coordenadas principales explicaron el 22.17 % de la variabilidad genética entre las subpoblaciones. Los componentes uno, dos y tres presentaron unos porcentajes de 14.57 %, 4.0 % y 3.61 %, respectivamente.
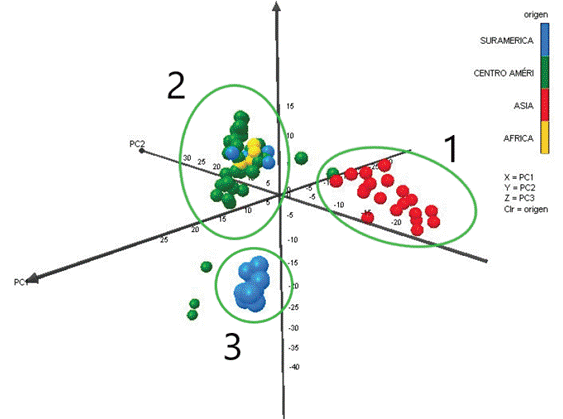
Figura 2 Representación gráfica en tres dimensiones de la ubicación espacial de las líneas evaluadas a partir del análisis de coordenadas principales (PCoA). Los colores corresponden al origen de los genotipos, de acuerdo con la información del banco de germoplasma de Semillas Valle S.A.
De acuerdo con los resultados obtenidos se estableció una separación entre poblaciones, observando tres grupos diferenciados en la representación gráfica, donde se asignaron colores de acuerdo con el origen de las líneas, definido a partir de la información de procedencia y pedigrí del banco de germoplasma de Semillas Valle S.A., para observar si las agrupaciones de puntos corresponden con los orígenes de las líneas (Figura 2), evidenciando que el grupo 2 está compuesto por líneas principalmente de Centroamérica, África y algunos de Suramérica; el grupo 1 está compuesto por materiales de origen asiático y un genotipo de Centroamérica; y el grupo 3 está compuesto únicamente por materiales de Suramérica.
En el análisis de estructura poblacional realizado con el programa Structure, a partir de la metodología propuesta por Evanno et al., (2005), se graficó el número de clústeres (K) con el delta K (ΔK), donde se evidenció un pico agudo en K=3 y otro en K=7. Al visualizar el diagrama de barras en Structure, la diferenciación entre las poblaciones con K=7 no es clara, por tanto, se seleccionó como valor óptimo K=3 (Figura 3; Tabla suplementaria 4). El valor K óptimo indica que tres subpoblaciones tienen una alta probabilidad para el agrupamiento de la población, y cada subpoblación consistió en 20, 47 y 15 genotipos, respectivamente (Tabla 1). Por otra parte, los valores para diversidad genética promedio (HS) para las tres subpoblaciones son relativamente altos, 0.291, 0.367 y 0.248, respectivamente, e indican que la población 2 corresponde al grupo más diverso, siendo este el grupo con mayor número de genotipos. Los valores de heterocigosidad observada (HO) (0.098, 0.027 y 0.073, respectivamente), son bajos lo cual corresponde con líneas homocigotas, así como los valores altos de FIS (Tabla 2).

Figura 3 Estructura poblacional estimada de 82 genotipos de 66 líneas endocriadas de maíz trópica. Diferentes colores indican la agrupación en una subpoblación diferente.
Tabla 1 Resultados de Structure para la proporción de genotipos inferidos en cada clúster, número de genotipos asignados, diversidad genética promedio (Hs), heterocigosidad observada (Ho) e índice de fijación (FIS) para las subpoblaciones inferidas de las líneas de maíz
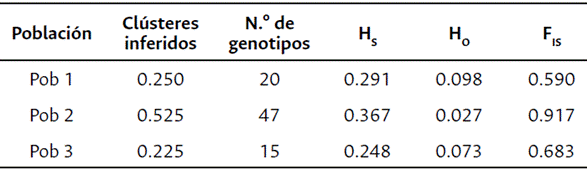
Tabla 2 Divergencia de frecuencia alélica entre las poblaciones inferidas de las líneas de maíz e intervalos de confianza superior e inferior para los FST calculados entre las poblaciones
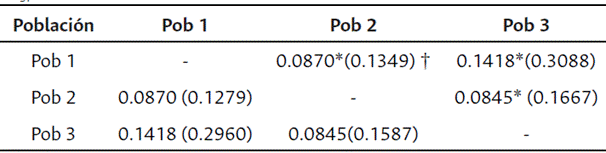
*FST significativamente diferente de 0 (P<0.05). †Intervalos de confianza: límite superior por encima de la diagonal; límite inferior por debajo de la diagonal.
La clasificación de los genotipos corresponde, en general, con los tres grupos observados en el análisis de coordenadas principales. El primer grupo está compuesto por 19 genotipos, el segundo por 52 genotipos, y el tercero por 11 genotipos, de acuerdo con la representación gráfica.
Para las 30 líneas parentales del ensayo de dialelo que también fueron analizadas con marcadores SNPs, la Tabla 3 compara su clasificación en grupos heteróticos mediante ambas metodologías. De los 30 genotipos evaluados, se observa una concordancia en la clasificación del 46.6 %, lo que significa que 14 de las 30 líneas evaluadas se clasificaron en la misma población en las dos metodologías.
Discusión
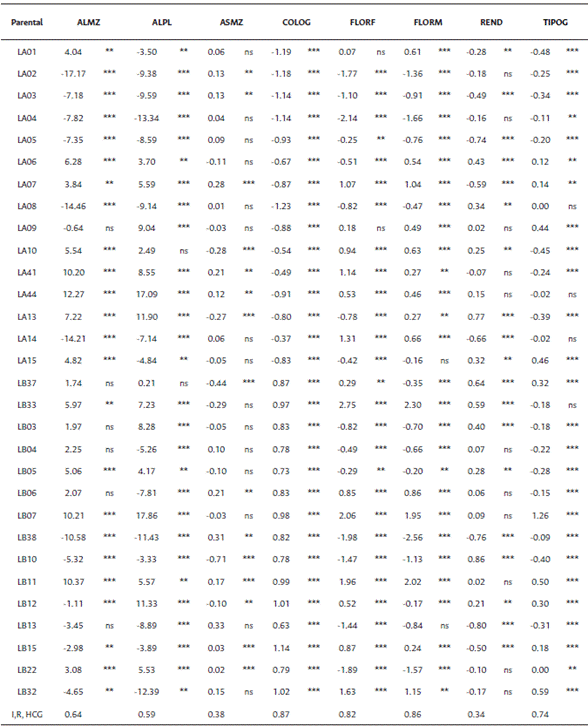
La significancia en la HCG sugiere que la variabilidad observada puede ser atribuida a efectos génicos aditivos, mientras que la significancia en la HCE indica que los parentales de los híbridos evaluados presentan cierta complementariedad entre ellos indicando efectos génicos no aditivos (Lu et al., 2020; Murtadha et al., 2018). Para rasgos como altura de mazorca y de planta, color y tipo de grano, y días de floración femenina y masculina, la magnitud de la HCG con relación a la HCE es de al menos 30 veces, lo que indica que, para el conjunto de líneas evaluadas, los efectos genéticos aditivos son mucho más importantes que los no aditivos para estos rasgos. Este patrón contrasta con los componentes de rendimiento, donde se observó que para las variables rendimiento de grano, peso de mazorcas y peso del grano, la HCG es solo 3 veces mayor que la HCE, indicando que para estas características predominan los efectos no aditivos sobre los aditivos.
A partir de la información de la HCG, es posible seleccionar líneas con potencial para ser usadas como probadores para clasificar nuevas líneas en grupos heteróticos. De acuerdo con Mhike et al. (2011), las líneas con potencial como probadores deben tener efectos de HCG positivos y significativos para el rendimiento de grano, deben estar claramente clasificadas en un grupo heterótico, presentar buen rendimiento de grano por sí mismas y tener efectos de HCE negativos y significativos con líneas de su mismo grupo. De acuerdo con estos criterios, las líneas LB10 y LA13 presentaron los mayores rendimientos en las localidades evaluadas, además de un efecto de HCG altamente significativo, por lo que estas líneas se consideran candidatas para ser usadas como probadores heteróticos del grupo 2. De las líneas definidas en el grupo 1 por las dos metodologías (fenotípica y genotípica), la línea LA08 fue la única línea con efectos de HCG significativamente positivos, y presentó un rendimiento alto respecto a las demás líneas del grupo, por lo que se considera un parental candidato para probador del grupo 1; además esta línea LA08 mostró buen desempeño en rendimiento de grano en cruza con las líneas LB15, LB37 y LA41, y de estas 3 líneas la LB37 se identifica definida en el grupo 2 por ambas metodologías, por lo que podría considerarse a la cruza LA08 x LB37 como Patrón Heterótico. Ninguna de las líneas clasificadas en el grupo heterótico 3 cumplió con los criterios para ser seleccionada como probador de este grupo, y se podría considerar no bien identificado este grupo al no tener concordancia con las dos metodologías.
La evaluación de los componentes principales a partir del análisis de SNPs muestra la existencia de estructura poblacional en el germoplasma evaluado, con una diferenciación moderada. Los resultados permitieron identificar que las poblaciones, en general, corresponden al origen de las líneas. El análisis de la diferencia de frecuencias alélicas entre las poblaciones inferidas permite establecer un valor de FST global alto de 0.17, y una mayor divergencia entre la subpoblación 1 y la subpoblación 3 (Tabla 2). Por otra parte, la subpoblación 2 presenta una divergencia significativa, respecto a las subpoblaciones 1 y 3, sin embargo, valores pequeños indican que existe una baja divergencia nucleotídica entre estas subpoblaciones (Romay et al., 2013).
Las dos metodologías evaluadas permiten clasificar los 30 genotipos en dos grupos genéticamente contrastantes o grupos heteróticos. La clasificación de las líneas endocriadas de maíz está influenciada por la metodología utilizada por los investigadores (Fan et al., 2009). Por tanto, algunos autores han realizado análisis comparativos entre la clasificación de grupos heteróticos a partir de datos fenotípicos y de marcadores moleculares con el fin de establecer la eficiencia de ambas metodologías (Santos De Oliveira et al., 2021; Silva et al., 2020).
Badu-Apraku et al. (2015), compararon la eficiencia de la clasificación de 14 líneas de maíz mediante la evaluación de SNPs y la HCG y HCE en ensayos de campo, encontrando que se tiene una correspondencia cercana entre las clasificaciones de las metodologías evaluadas en términos de la asignación de líneas en los grupos heteróticos. Similar a lo reportado por (Santos De Oliveira et al., 2021), quienes clasificaron 293 líneas endocriadas de grupo heterótico conocido, mediante 5252 SNPs, encontrando que se presenta consistencia entre el agrupamiento basado en las distancias genéticas y los grupos previamente establecidos para las líneas en estudio, con 3.4 % de líneas con una clasificación diferente a la esperada.
Los resultados de la presente investigación soportan hallazgos previos respecto a la utilidad de los estudios de diversidad genética a partir de marcadores moleculares SNPs en la clasificación de líneas de maíz en grupos genéticos, permitiendo la selección de parentales para la formación de híbridos con características genéticas contrastantes, evitando la formación de híbridos entre líneas genéticamente similares o dentro de los grupos heteróticos (Santos De Oliveira et al., 2021; Silva et al., 2020).
La implementación del dialelo es 8.3 veces más costoso respecto a la genotipificación con el Maize50SNP BeadChip y el tiempo requerido es 3 veces más largo, debido al requerimiento de producción de semilla y varias observaciones de híbridos en campo. Esta observación está en concordancia con lo reportado por (Fan et al., 2009), indicando que los métodos moleculares para clasificar líneas endocriadas en grupos heteróticos usualmente son más rápidos y pueden ser más económicos, ya que no requieren la evaluación de gran cantidad de materiales en campo. En estados avanzados de mejoramiento, los métodos basados en datos genéticos y marcadores moleculares son ampliamente utilizados ya que tienen un alto nivel de precisión, dado que sus resultados están mínimamente influenciados por el ambiente (Olutayo, 2021). Silva et al. (2020), indican que la metodología de SNPs puede proveer información importante para la selección de parentales y servir como base para el planteamiento de esquemas de cruzamiento adecuados. Debido a que los resultados entre los agrupamientos con marcadores y con los datos de campo presentan algunas inconsistencias, algunos autores concluyen que los datos de ensayos de campo en diferentes ambientes continúan siendo esenciales para clasificar líneas no relacionadas en grupos heteróticos (Aguiar et al., 2008).
Conclusiones
Tanto la metodología del dialelo como los marcadores moleculares tipo SNP fueron eficientes en la clasificación de líneas endocriadas en grupos genéticos contrastantes, obteniendo dos grupos heteróticos para el carácter rendimiento de grano, con una divergencia en la clasificación del 53.3 %. El uso de marcadores moleculares resulta ser una metodología más fácil de implementar, con un costo más bajo y menor tiempo requerido para su implementación, por lo cual permite la inclusión de mayor cantidad de líneas en un mismo ensayo. Aunque el ensayo dialelo es útil para clasificar las líneas en grupos heteróticos, es una metodología restrictiva ya que requiere una alta inversión de recursos para la producción de semilla y posterior evaluación de híbridos en campo. Sin embargo, su aplicación contribuye en la selección de parentales probadores representativos para cada grupo heterótico identificado, que permitan realizar una clasificación de líneas nuevas mediante cruzamientos de línea x probador, y la identificación de patrones heteróticos cuando se analiza la HCG y la HCE. La separación de las líneas endocriadas en grupos heteróticos constituye la base para el direccionamiento en la formación de híbridos nuevos en el programa de mejoramiento de Semillas Valle S.A., contribuyendo a mejorar la eficiencia debido a la posibilidad de evitar la formación de híbridos entre líneas genéticamente similares, que no expresarán en campo la heterosis requerida en el desarrollo de híbridos de maíz.