Introducción
El síndrome respiratorio agudo severo causado por el coronavirus tipo 2 SARS CoV-2, cuyo mecanismo de patogénesis es poco conocido pero similar a los virus SARS-CoV y MERS, brinda información sobre la genética y el diseño diagnóstico para COVID19 1. Se han Identificado cuatro géneros de coronavirus: α, β, γ, δ. Al género β pertenecen los ya mencionados; tienen genoma de ARN monocatenario de sentido positivo, tamaño de 26-32Kb y longitud de 100 nm 2.
Las espículas que los caracterizan están compuestas por una glucoproteína (maleable, alta tasa de mutaciones y recombinación) que les ayuda en su adaptación evolutiva y conversión a un patobionte en el ser humano 3. La similitud de genomas con CoV y MERS evidencia que el origen del virus SARS CoV-2 es natural y surgió por la alta tasa de mutaciones que presentan. Los típicos CoV contienen al menos diez marcos abiertos de lectura (ORF). Los primeros se traducen en dos poliproteínas grandes y los otros ORF de SARS-CoV-2 codifican para cuatro proteínas estructurales principales: proteínas de espícula (S), envoltura (E), nucleocápside (N) y membrana (M), así como varias proteínas accesorias como orf 3, 6, 7a, 7b, 8 y 9b, que codifican proteínas capaces de evadir la respuesta inmune 4.
Cuando el virus introduce su ARN a las células, crea millones de copias iguales. Cada virus puede generar entre 10 000 y 100 000 copias; de esta manera, cada gota respiratoria producida por una persona con infección activa puede tener hasta 100 millones de viriones, que permiten una fácil diseminación viral y, por lo tanto, alto contagio 2.
El diagnóstico de COVID-19 se basa en la historia epidemiológica, las manifestaciones clínicas y pruebas de laboratorio como la RT-qPCR, tomografía computarizada, identificación de IgM /IgG por Elisa 5. Sin embargo, los síntomas y signos clínicos de los pacientes infectados son altamente atípicos y, por lo tanto, las pruebas moleculares son indispensables para el diagnóstico. Este se lleva a cabo en laboratorios nivel BSL II, con cámaras de flujo laminar tipo II y filtros EPA. Asimismo, las muestras deben estar en triple envase en contenedores homologados bajo normativa de “Sustancia biológica clase B (UN3373)” y se transportan a 4 °C 6; las áreas de trabajo que aseguran el correcto manejo de las muestras, está el área sucia para recepción, registro, desembalaje e inactivación de las muestras y el área limpia, para la extracción del ARN y realización de RT-qPCR 7.
La toma y el tipo de muestra dependen de la procedencia y especificaciones clínicas del paciente. Para el ambulatorio se recomienda el hisopado nasofaríngeo y orofaríngeo, recolectados en un mismo tubo con medio de transporte viral, que incrementa la carga viral (108- 109 copias/hisopado). Si son pacientes hospitalizados, lo adecuado son los aspirados endotraqueal y nasofaríngeo, además el lavado nasal o broncoalveolar, aunque diluye la muestra. El esputo tomado de vías respiratorias bajas contiene más carga viral (108-1011 /ml) y más virus vivos, por lo cual se recomienda después de las 2 semanas de sintomatología 8. Los blancos moleculares de detección más usados para el ensayo de tamizaje de primera línea, está el gen E(envoltura), usado para el diseño de primers y sonda Taqman; si da positivo, se continua con las pruebas confirmatorias usando el gen RdRP (RNA polimerasa dependiente de RNA). Otro blanco usado es el gen Orf1ab (tabla 1) 9.
Tabla 1 Análisis de resultados de las pruebas moleculares RT-qPCR sonda Taqman.

Nota: los genes blanco molecular, deben ser detectados para considerar la positividad viral, mientras que el control endógeno respalda la interpretación, ya que confirma la adecuada toma de muestra, integridad del ARN obtenido y ausencia de inhibidores de la PCR.
Para el diagnóstico de confirmación en zonas sin circulación del virus se necesita la positividad frente a dos genes distintos de COVID-19, uno de ellos específico, o positivo frente a un betacoronavirus, más una identificación al menos parcial del genoma del virus COVID-19. En zonas de transmisión comunitaria, se considera suficiente la positividad de la RT-qPCR para un único gen que sea discriminatorio de COVID-19.
La especificidad de las sondas Taqman permite seguir el rastro de varias reacciones de amplificación independientes en un mismo tubo. Cada sonda se puede diseñar con un fluoróforo que emita a una longitud de onda distinta y el equipo debe tener diferentes filtros para cada uno de estos fluoróforos: FAM (emisión a 520 nm) y ROX (623 nm).
Los protocolos más usados son el de París, Centers for Disease Control (CDC) y el de Berlín, recomendado por la Organización Mundial de la Salud (OMS), los cuales se basan en extracción de ARN mediante columnas de afinidad o usando perlas magnéticas; retrotranscripción en un solo paso, es decir junto con la mezcla maestra de qPCR Taqman simple con una sonda, un amplicón por tubo, o múltiple, con dos o más sondas, y varios amplicones por tubo. El análisis de datos se hace determinando el ciclo umbral de detección o Ct de muestras por comparación con controles positivos, negativos y endógenos (figura 1). El tiempo total del análisis desde la llegada al laboratorio de las muestras hasta el reporte es de 12 horas, si es método manual para 94 muestras, y 8 horas aproximadamente, si es automatizado 11,12.
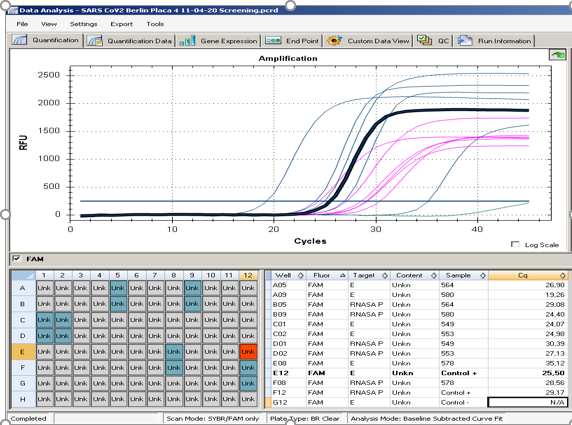
Fuente: Autores.
Figura 1 Amplificación por RT qPCR de los genes E (SARS-CoV-2) y RNAsa P (control endógeno). En azul se observa el gen E y en rosado RNAsa Fuente: Eliana P. Calvo. Grupo de Virología. Universidad El Bosque
En cuanto a los falsos negativos, estos son debidos a la calidad y cantidad de la muestra, debido al manejo o envío inadecuados (sin refrigeración). El virus es termolábil y se degrada con facilidad; de hecho, una vez el RNA se extrae, se degrada progresivamente y afecta la sensibilidad. Muestras recogidas durante el pico, muy temprano o fase tardía de la infección; mutaciones del virus, y presencia de inhibidores de la PCR. En estos casos se recomienda una nueva toma de muestra, especialmente de vías respiratorias bajas, para aumentar la carga viral, teniendo en cuenta que la sensibilidad analítica de la RTqPCR es 5,2 copias de ARN/reacción y que estos factores podrían disminuirla.
En cuanto a la especificidad (95 % a 100 %), esta depende de las secuencias de los primers, los cuales se deberían diseñar de acuerdo con la secuenciación de los aislamientos según distribución geográfica 13. La PCR es muy sensible durante las dos primeras semanas de la infección, pero a partir de la tercera semana se va negativizando; por eso, en este punto, se recomienda el apoyo con pruebas rápidas y la detección de anticuerpos. Asimismo, personas positivas deben tener seguimiento y tomarse dos muestras en intervalo de 14 días, las cuales deben ser negativas, para que se consideren no portadoras del virus.
Finalmente, para garantizar resultados confiables se requiere validar y controlar la calidad de pruebas in house” y kits comerciales. Estas pruebas diagnósticas, consideradas en la actualidad como de referencia y de elección para COVID-19, deben seguir las recomendaciones de la FDA 14,15.














