Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Ciencias Pecuarias
Print version ISSN 0120-0690On-line version ISSN 2256-2958
Rev Colom Cienc Pecua vol.25 no.2 Medellín Apr./June 2012
ARTÍCULO ORIGINAL
Genetic variability of the bovine prolactin–Rsal loci in Holstein cattle in Antioquia province (Colombia) ¤
Variabilidad genética del loci RsaI de la prolactina bovina en ganado Holstein de Antioquia, Colombia
Variabilidade genética do loci RsaI da prolactina bovina em gado Holandês de Antioquia, Colombia
Juan C Rincón1*, Zoot, MS; Albeiro López, Zoot1,2, MV, MS, PhD; Julián Echeverri1,3, Zoot, MS, PhD
* Autor para correspondencia: Juan C Rincón. Universidad Nacional de Colombia, Sede Medellín. E–mail: jcrincon@unal.edu.co
1 Grupo BIOGEM: Biodiversidad y Genética Molecular. Universidad Nacional de Colombia, Sede Medellín. Facultad de Ciencias Agropecuarias.
2 Profesor Asociado. Universidad Nacional de Colombia, Sede Medellín. Facultad de Ciencias Agropecuarias.
3 Profesor Auxiliar. Universidad Nacional de Colombia, Sede Medellín. Facultad de Ciencias Agropecuarias.
(Recibido: 27 mayo, 2011; aceptado: 22 noviembre, 2011)
Summary
The Holstein breed, widely used in Antioquia's dairy industry, has undergone genetic changes due to selection that have affected the frequencies of some polimorphisms within important genes. Objective: to identify polymorphism changes in the prolactin RsaI (PRL–Rsal) locus and to characterize the structure and allelic distribution within the Holstein population in Antioquia. Methods: a total of 1.462 Holstein cows from 11 subpopulations (municipalities) of Antioquia were used. The ADN was extracted from leucocytes using the salting–out method and genotyping was performed by PCR–RFLP. Genetic diversity was determined by heterozygosity. The Hardy–Weinberg equilibrium (HW) and the genetic differentiation among populations were performed using the Arlequin 2.0 software. Allelic and genotypic frequencies were assessed with the SAS 9.2 statistical software. Results: the genotypic frequencies found were 0.695 (AA), 0.276 (AB), and 0.029 (BB), while the frequencies of the A and B alleles were 0.833 and 0.167, respectively. There were no deviations from HW equilibrium in any population. Genetic diversity among populations, expressed in terms of heterozygosity, showed a medium value (Ho=0.276). The FST value of the entire population was significant, indicating genetic differentiation. In addition, some matched FST showed high differentiation. The FIT and FIS parameters were not significant, suggesting that population endogamy or exogamy is not occurring. Conclusions: the bovine PRL–RsaI polymorphism is a suitable marker to evaluate characteristics of economic importance and apparently it has not been influenced by selection pressure. In fact, a significantly high variability (p<0.05) in allele frequencies was observed within the Antioquian Holstein cattle subpopulations.
Key words: genetic differentiation, Hardy–Weinberg equilibrium, PCR–RFLP, single nucleotide polymorphism.
Resumen
La raza Holstein en Antioquia ha sido utilizada ampliamente por la industria de lechería especializada, lo cual ha generado cambios genéticos en las diferentes poblaciones debido a algunas fuerzas de selección que han afectado las frecuencias de algunos alelos en genes de importancia económica. Objetivo: determinar las frecuencias alélicas y genotípicas de un polimorfismo del exón 4 del gen de prolactina bovino (PRL) en el cual se genera un sitio polimórfico para la endonucleasa de restricción RsaI y a partir de estas frecuencias estimar algunos parámetros de estructura poblacional en ganado Holstein. Métodos: el estudio se llevó a cabo en 1.462 vacas Holstein de 11 subpoblaciones (municipios) del departamento de Antioquia. Se extrajo ADN de leucocitos de sangre periférica por el método de Salting out y se realizó la genotipificación mediante la técnica de PCR–RFLPs. La diversidad genética se expresó mediante las heterocigosidades. Se determinó adicionalmente el equilibrio de Hardy–Weinberg y la estructuración genética poblacional mediante el software Arlequin 2.0. Las frecuencias alélicas y genotípicas se evaluaron mediante el paquete estadístico SAS 9.2. Resultados: las frecuencias genotípicas encontradas fueron 0.695 (AA), 0.276 (AB) y 0.029 (BB) y las frecuencias alélicas 0.833 (A) y 0.167 (B). No se encontraron desviaciones del Equilibrio de Hardy–weinberg en ninguna población. La heterocigosidad fue media entre poblaciones (Ho=0.276). El valor de FST de toda la población fue significativo, indicando estructuración genética. Los estadísticos FIT y FIS no fueron significativos, por tanto no es posible asumir endogamia o exogamia en las poblaciones teniendo como referencia este polimorfismo. Conclusiones: el polimorfismo en el exón 4 del gen de la prolactina bovina es un marcador interesante para evaluar características de importancia económica, ya que este no parece haber sido sometido a selección directa, presenta frecuencias alélicas muy variables entre poblaciones, con diferenciación significativa e incluso muy alta entre algunas subpoblaciones (p<0.05).
Palabras clave: diferenciaci n genética, equilibrio de Hardy–Weinberg, PCR–RFLP, polimorfismo de nucle tido simple.
Resumo
A raça Holandesa em Antioquia tem sido muito utilizada pela indústria leiteira especializada, isto tem gerado mudanças genéticas nas diferentes populações devido a algumas forças de seleção que tem afetado as frequências de alguns alelos em genes de importância econômica. Objetivo: determinar as frequências alélicas e genotípicas de um polimorfismo no exon 4 do gene da prolactina bovina (PRL) no qual se gera um sitio polimórfico para a endonuclease de restrição RsaI e com essas frequências estimar alguns parâmetros da estrutura populacional no gado Holandês. Métodos: o estudo foi realizado em 1.462 vacas da raça Holandesa de 11 subpopulações (municípios) de Antioquia. A extração do ADN se fez pelo método de Salting Out e a genotipagem foi realizada pela técnica PCR–RFLP. A diversidade genética foi determinada mediante as heterozigosidades, e o equilíbrio de Hardy–Weinberg (HW) e a diferenciação genética entre as populações foram realizadas usando o software Arlequin v. 2.0. As frequências alélicas e genotípicas foram analisadas com o programa estatístico SAS v. 9.2. Resultados: as frequências genotípicas encontradas foram 0.695 (AA), 0.276 (AB) e 0.029 (BB) e as frequências alélicas 0.833 (A) e 0.167 (B). Não se encontraram desvios no equilíbrio de Hardy–Weinberg nas subpopulações. A diversidade genética expressada como a heterozigosidade observada foi media entre toda a população (Ho=0.276). O valor de FST de toda a população foi significativo indicando estruturação genética. Os estatísticos FIT e FIS não foram significativos, por tal razão não foi possível assumir endogamia ou exogamia nas subpopulações. Conclusões: o SNP no exon 4 do gene da prolactina bovina é um marcador interessante para avaliar características de importância econômica, já que este não parece ter tido um processo de seleção direta, alem, tem frequências alélicas muito variáveis entre as diferentes subpopulações, com diferenciação significativa e ainda muito elevada entre as subpopulações avaliadas (p<0.05).
Palavras chave: diferenciaão genética, Hardy–Weinberg, PCR–RFLP, polimorfismo de nucleot deo simples.
Introducción
La raza Holstein ha sido ampliamente utilizada por la industria lechera de Colombia, lo que ha generado cambios genéticos en las subpoblaciones, debido a las diferentes presiones de selección propias de los distintos sistemas de producción y sus condiciones técnicas. En la actualidad se conocen un amplio número de marcadores genéticos que pueden ser usados para evaluar la diferenciación poblacional (Lirón et al., 2002). Entre estos marcadores se encuentran algunos relacionados con parámetros productivos en ganado de leche (Bedoya et al., 2001), como es el caso del polimorfismo de un sólo nucleótido o SNP que se encuentra localizado en el gen de la prolactina Bovina (Khatami et al., 2005).
El gen de la prolactina bovina está localizado en el cromosoma 23, conformado por 5 exones y 4 intrones (Brym et al., 2007), que codifican para una proteína madura de 199 aminoácidos con un peso molecular de 23 KDa (Cao et al., 2002). Este gen presenta un polimorfismo que consiste en una transición silenciosa de Adenina–Guanina en el codón para el aminoácido 103 en el exón 4 del gen, que da lugar a un sitio polimórfico para la endonucleasa de restricción RsaI (Brym et al., 2005), permitiendo la identificación de dos alelos (A y B) y 3 genotipos diferentes (AA, AB y BB) (Dybus, 2002).
Los parámetros de estructura poblacional describen el grado de reducción de heterocigosidad en una población, con base en el equilibrio de Hardy–Weinberg, lo cual faculta la comparación entre grupos a diferentes escalas jerárquicas y determina el grado de diferenciación entre ellos (Wright, 1969).
La determinación de algunos parámetros poblacionales como las frecuencias, alélicas, genotípicas y el flujo de genes entre las poblaciones usando el gen de prolactina, permite determinar su grado de diversidad y diferenciación (Lirón et al., 2002). Por tal razón la determinación del equilibrio de Hardy–Weinberg (HW), las frecuencias y los parámetros de estructura, permiten no sólo determinar el comportamiento poblacional, sino que de acuerdo a las frecuencias de los alelos en la población, permiten predecir la efectividad de un programa de mejoramiento genético basado en este gen, ya que aquellos alelos que se encuentran casi fijados en una población tendrían diferente respuesta a la selección que aquellos alelos que se encuentran en proporciones intermedias. El objetivo de la presente investigación fue determinar los parámetros de estructura poblacional y las frecuencias alélicas y genotípicas del gen de la prolactina bovina en vacas Holstein del departamento de Antioquia, como aporte para la implementación de programas de selección asistida por marcadores moleculares (MAS).
Materiales y métodos
Área y poblaciones de estudio
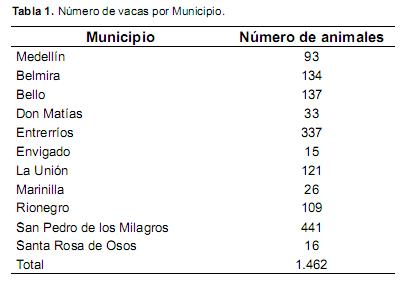
En el estudio se incluyeron 1.462 vacas de la raza Holstein, distribuidas en 120 hatos, pertenecientes a 11 municipios del departamento de Antioquia. Los municipios incluidos fueron San Pedro de los Milagros (Altura: 2.475 msnm; Tº: 14 ºC), Belmira (Altura: 2.550 msnm; Tº: 14 ºC), Entrerríos (Altura: 2.300 msnm; Tº: 16 ºC), La Unión (Altura: 2.500 msnm; Tº: 13 ºC), Envigado (zonas entre los 2.000 a 2.300 msnm; Tº: 16 ºC), Bello (Zonas entre los 2.000 a 2.400 msnm; Tº: 16 ºC), Medellín (Zonas entre los 2.000 a 2.500 msnm; Tº: 15 ºC), Santa Rosa (2.581 msnm; Tº: 14 ºC), Don Matías (2.200 msnm; Tº: 16 ºC), Marinilla (2.150 msnm; Tº: 17 ºC) y Rionegro (2.150 msnm; Tº: 17 ºC). Cada municipio evaluado fue tomado como una subpoblación de Antioquia y el número de individuos para cada subpoblación se presenta en la tabla 1.
Toma de muestras
Se colectaron muestras de sangre de la vena coccígea de los animales seleccionados utilizando para ello BD vacutainer de 5 ml con EDTA (Ácido Etilendiaminotetraacético) como anticoagulante y con agujas número 18 (BD Vacutainer TM, Franklin Lakes, NJ). Los tubos fueron homogenizados por inversión, rotulados para su identificación y refrigerados con hielo durante su transporte hasta el Laboratorio de Biología Celular y Molecular de la Universidad Nacional de Colombia sede Medellín, para realizar la extracción del ADN.
Extracción del ADN de células de sangre
Se realizó el protocolo de extracción de ADN basado en el método de ''salting out'', modificado por Miller et al. (1988) y se almacenó a 4 ºC hasta el momento del análisis. La cantidad y calidad del ADN extraído se evaluó en gel de agarosa al 0.8% teñido con Bromuro de Etidio, y se semicuantificó usando el transiluminador (Biometra®, Goettingen, Germany). La pureza del ADN genómico se determinó mediante un análisis de relación de absorbancia a dos longitudes de onda (260/280 nm), usando para tal fin el Nanodrop 2000c (Thermo scientific , Wilmington, USA). Sólo se usó el ADN genómico con una pureza mayor de 1.8 y menor de 2, lo cual es recomendado para obtener un ADN puro.
Amplificación mediante PCR de la región especifica del gen de la prolactina bovino PRL
Con base en la secuencia del gen de la prolactina bovina y los reportes generados por Brym et al. (2005), se sintetizaron dos oligonucleótidos de 23 y 22 pares de bases (pb), respectivamente, que permiten amplificar un fragmento de 294 pb que se encuentra localizado en el cuarto Exón del gen de prolactina y además presenta un polimorfismo de nucleótido simple (SNP) reconocible por la endonucleasa de restricción RsaI. Los cebadores sintetizados se muestran en la tabla 2.
Se realizó una amplificación mediante la reacción en cadena de la polimerasa (PCR) para la región específica en un volumen final de 25 μL que contiene 2.5 L de buffer PCR 10X (1.0 –1.5 mM de MgCl2, 50 mM de KCl, 10 mM de Tris–HCl, pH de 8.3), 2 mM de MgCl2, 0.4 mM de cada dNTP , 300 nM de cebadores, 0.5–1 unidad de taq polimerasa (Bioline USA, Randolph, MA) y 5 μl de ADN genómico con una concentración de 50 a 300 ng/ μl.
La PCR se desarrolló en un termociclador (Biometra®, Goettingen, Germany) bajo las siguientes condiciones: 1) desnaturalización a 95 ºC por 7 minutos, 2) desnaturalización a 95 ºC por 1 min, 3) temperatura de alineamiento (annealing) de 60 ºC por 30 segundos, 4) extensión a 72 ºC por 40 segundos, 5) se repitió desde el paso 2 hasta el 4 durante 39 ciclos, 6. Se finalizó con una extensión por 5 minutos a 72 ºC.
Los productos de PCR fueron visualizados mediante electroforesis en gel de agarosa al 2% (Amresco, Cochran Road, OH), teñido con bromuro de etidio (Promega, Madison, WI). En cada pozo se sirvieron 5 μl del producto de PCR diluidos en 2 μl de buffer carga 1X (fermentas, Glen Burnie, MD) y se utilizaron 2 μl de marcador de peso molecular de 100 pb (low range Fermentas, Glen Burnie, MD). Los geles fueron visualizados mediante un fotodocumentador(BiometraTM,Goettingen, Germany) para tomar evidencia fotográfica. En todos los casos en la PCR se uso un control negativo sin ADN y un control positivo que poseía un ADN previamente amplificado con éxito.
Digestión con enzimas de restricción del fragmento amplificado
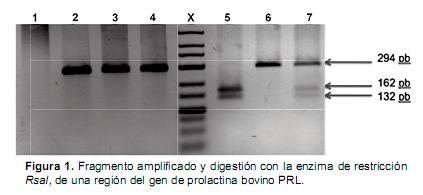
Una vez se comprobó la amplificación del fragmento de 294 pb, los productos de PCR fueron sometidos a digestión con la endonucleasa de restricción RsaI. Dicha enzima generó corte en presencia de la transición Adenina–Guanina, lo que permitió la identificación del alelo B debido a la producción de dos bandas de 162 y 132 pb. El alelo A ante la ausencia del sitio de restricción específico sólo genera bandas del tamaño inicial (294 pb). Teniendo en cuenta lo anterior se presentaron los tres genotipos posibles, el homocigoto AA que presentó sólo un fragmento de 294 pb, el heterocigoto AB que presentó fragmentos de 294, 162 y 132 pb; y el homocigoto BB que presentó dos fragmentos de 162 y 132 pb.
Para la digestión se utilizaron 2 μl de buffer Tango 10X para obtener una concentración final de 1X y un volumen de reacción de 20 μl, se adicionaron cinco unidades de enzima RsaI (Fermentas, Glen Burnie, MD ) y se completó con agua desionizada hasta 15 μl, para utilizar 5 μl del producto de PCR obtenido. Una vez realizada la mezcla esta se sometió a digestión durante 3 horas a 37 ºC. La enzima en esta reacción se utilizó en exceso para evitar la formación de falsos heterocigotos, debido a digestiones parciales del producto de PCR.
En todas las digestiones se realizó un control positivo que presentaba el sitio de restricción en ambos alelos (genotipo BB), y de esta manera se procuró evitar la generación de falsos heterocigotos
Los productos de la digestión se resolvieron por electroforesis en gel de agarosa al 2.5% (Amresco, Cochran Road, OH) en buffer TBE 1X (EDTA 0.05 M, Tris base 0.089 M y Acido Bórico 0.089 M), al cual se le adicionó Bromuro de Etidio (Promega, Madison, WI). En cada pozo se sirvieron 15 μl de producto de digestión diluidos en 5 μl Buffer de carga 1X (Fermentas, Glen Burnie, MD), se pusieron 2 μL de un marcador de peso molecular con rango 100 a 700 pb en cada línea (Low Range Fermentas, Glen Burnie, MD) (Dybus, 2002). Los geles fueron visualizados bajo luz ultravioleta en un equipo de foto documentación de geles (BiometraTM, Goettingen, Germany) con el cual se obtuvo una fotografía digital.
Análisis estadístico
Frecuencias alélicas y genotípicas. La frecuencia de los diferentes alelos se realizó determinando la proporción de cada forma del gen entre el número de copias totales de la población en estudio. Se identificaron los homocigotos (dos copias del mismo alelo) y los heterocigotos (una copia de cada alelo) y se calculó la frecuencia F de cada alelo contando los homocigotos y añadiendo la mitad de los heterocigotos, con el método descrito por Hartl, (2007). La frecuencia genotípica se calculó como la sumatoria de cada genotipo dividido el total de la población (Hartl, 2007). Ambas frecuencias se determinaron usando el paquete SAS 9.2 (SAS Institute Inc, 2003).
Diversidad genética. Se estimó dentro de las poblaciones y en la población total la heterocigosidad observada (Ho) y la heterocigosidad esperada (He) utilizando la metodología de cadena de Markov (Guo et al., 1992) implementado en el software Arlequín v 2.0 (Excoffier et al., 2005).
Estructura genética. El equilibrio de Hardy– Weinberg se determinó con base en las frecuencias alélicas y genotípicas esperadas y observadas utilizando el software Arlequín v 2.0 (Excoffier et al., 2005). Los parámetros de estructura poblacional, se calcularon utilizando el método propuesto por Wright (1969) que parte el coeficiente de endogamia de una población subdividida. De esta manera se calculó el parámetro FIT = HT–Hi/HT, que corresponde a la endogamia total, el parámetro FIS=HS–HI/HS que mide la subdivisión intrapoblacional y el FST=FIT–FIS/1–FIS que mide la subdivisión poblacional (Wright, 1969); donde HT se refiere a la heterocigosidad esperada en la población total, HI es la heterocigosidad promedio observada en un grupo de poblaciones y HS es la heterocigosidad promedio esperada de cada población. Los cálculos fueron realizados mediante el software Arlequín v 2.0 (Excoffier et al., 2005), utilizando para ello el análisis de varianza molecular (AMOVA) que permite analizar la variación entre y dentro de poblaciones, con su significancia estadística, bajo la hipótesis nula ''los alelos o genotipos tienen la misma distribución en todas las subpoblaciones''.
El flujo génico fue expresado como el número de migrantes (Nm) por generación, y se calculó con base a la siguiente expresión derivada de los estadísticos de Wright (1969).
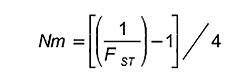
Resultados
Determinación de las frecuencias alélicas y genotípicas
Los fragmentos del gen prolactina de 294 pb fueron amplificados exitosamente en 1.462 muestras de ADN. Los fragmentos de ADN amplificados fueron digeridos usando la enzima de restricción RsaI. Las muestras fueron catalogadas como (AA, AB y BB) de acuerdo a los tamaños de los fragmentos generados (Figura 1).
Carril X, marcador de peso molecular de 100pb. Carril 1 control negativo; carril 2, 3 y 4 tres muestras de ADN amplificadas para el gen bGH; carril 5, patrón de restricción para genotipo homocigoto (BB); carril 6, patrón de restricción para genotipo homocigoto (AA) y carril 7, patrón de restricción para genotipo heterocigoto (AB).
Las frecuencias de los alelos A y B en la población total fueron 0.833 y 0.167, respectivamente. La frecuencia de los genotipos, AA, AB y BB fueron 0.695, 0.276 y 0.029 respectivamente. Las frecuencias alélicas y genotípicas por municipios y la total se presentan en la tabla 3, donde es posible observar una presencia mayor de homocigotos BB en la subpoblación de Marinilla y en Envigado, comparada con las demás poblaciones y el promedio.
Diversidad genética
No se encontró diferencia significativa entre Ho y He; en general Ho estuvo entre 0.133 y 0.394, mientras la esperada estuvo entre 0.217 y 0.382, mostrándose una diferencia considerable pero no significativa (p>0.05).
En ninguno de los municipios evaluados se encontraron evidencias suficientes para suponer desvíos significativos (p>0.05) en el equilibrio de Hardy–Weinberg (Tabla 4), por tanto no es posible evidenciar preferencias de apareamiento, ni cambios en las frecuencias genotípicas entre una generación y la siguiente. La subpoblación de Envigado presentó la mayor diferencia entre Ho y He, encontrándose en el límite de la significancia, lo que parece indicar una tendencia de apareamiento específico en esta población
Estructura y diferenciación genética de las poblaciones
El estadístico FST para la población total presentó un valor de 0.02067 indicando que algunas de las poblaciones se han diferenciado, aunque la magnitud de la diferenciación genética es baja (p<0.05) según los rangos propuestos por Wright (1969).
Los FST pareados por municipios, estuvieron entre 0.0002 y 0.2960, con diferencias significativas (p<0.05) en algunos casos (Tabla 5), por tanto se hallaron valores de diferenciación genética (FST) bajos (FST<0.05), moderados (FST de 0.05–0.15), altos (FST 0.15–0.25) y muy altos (FST>0.25) según los rangos propuestos por Wright (1969).
Los valores de FST pareados entre las poblaciones Marinilla–San Pedro (0.296) y Marinilla–La Unión (0.268) presentaron una diferenciación muy alta y significativa según el mismo rango de comparación. En general la subpoblación de Marinilla presentó una diferenciación alta y significativa con la mayoría de poblaciones, debido principalmente a que esta población presenta la mayor frecuencia de individuos BB (Tabla 3), lo que la hace diferente al resto de poblaciones. Adicionalmente, la alta diferencia entre esta población y las demás puede ser producto de que en Marinilla la presión de selección realizada para volumen de producción ha sido mucho menor que la realizada en los demás municipios, por tanto el proceso de fijación de algunos de los alelos puede llegar a ser más lento y no percibirse en esta población como en las demás en las cuales se ve una alta fijación del alelo A.
Como se observa en la tabla 6, las poblaciones presentaron diferentes valores del estadístico FIS, en algunos casos con valores positivos y en otros negativos. El promedio obtenido fue de –0.01070 y no fue significativo (p>0.05), del mismo modo los valores de FIS por municipio carecieron de significancia (p>0.05), por lo tanto, no es posible asumir tendencias a la endogamia, ni a la exogamia en los municipios analizados. Sin embargo, los Municipios de Marinilla y Envigado presentaron valores de FIS altos, positivos y cercanos a la significancia, lo cual parece indicar una tendencia a la endogamia en estas poblaciones. Dicho planteamiento es reforzado por el resultado planteado en la tabla 3 donde se observa una diferencia alta entre Ho y He, llegando a estar en el límite de la significancia y cercana a la pérdida de equilibrio de Hardy–Weinberg.
El estadístico FIT obtenido fue de 0.01029 y no fue significativo (p>0.05), por tanto, no es posible afirmar que exista una tendencia a la endogamia (Tabla 6).
Flujo génico. El valor medio de flujo de genes representado como Nm (Número de migrantes) fue de 12, un valor determinante, considerando que la tasa efectiva de migración de más de un inmigrante por generación, es suficiente para evitar una diferenciación por efectos de deriva génica. La baja diferenciación genética (FST) y la alta tasa efectiva de migración (Nm) observada en estas poblaciones podría estar relacionada directamente con la utilización de inseminación artificial y la comercialización de vacas entre los diferentes municipios.
Discusión
Los marcadores moleculares han sido ampliamente utilizados en genética de poblaciones para realizar evaluaciones de estructuración y diversidad genética. Entre los marcadores más utilizados se encuentran los polimorfismos en la longitud de fragmentos amplificados (AFLP), que son comúnmente usados para determinar la diversidad genética, pero que por ser marcadores dominantes, no permiten evaluar algunos parámetros de estructuración. Otro marcador de gran importancia son los microsatélites, que poseen la ventaja de ser codominantes y además altamente polimórficos, por lo que se usan frecuentemente en genética de poblaciones para determinar no sólo diversidad sino estructuración. Uno de los marcadores más utilizados para evaluación poblacional son los SNP que poseen la ventaja de ser codominantes, y de tener bajas tasas de mutación, aunque presentan poca información polimórfica (Freeland, 2005). En el presente trabajo se utilizó un solo SNP al interior del gen PRL, ya que este es un gen de importancia económica y se quiso determinar el comportamiento del flujo de este gen en las poblaciones de Colombia. Además, dicho marcador se comportó como un gen neutro en la población evaluada, lo cual lo válida para ser utilizado en análisis poblacionales.
Teniendo en cuenta lo anterior, se realizó el análisis poblacional y se encontró que todas las poblaciones estudiadas del departamento de Antioquia presentaron equilibrio de Hardy– Weinberg para el gen de prolactina, lo que podría sugerir que dichas poblaciones no presentan un proceso fuerte de selección sobre este gen, de tal forma que no se ha logrado todavía la prevalencia de alguna de las formas genotípicas, ni la pérdida del equilibrio por selección. Este resultado concuerda con lo reportado por Mehmannavaz et al. (2009), cuando encontraron equilibrio de Hardy– weinberg al analizar 286 toros Holstein iraníes para el mismo marcador. Del mismo modo, Dybus et al. (2005) concluyeron que había equilibrio cuando realizaron la prueba sobre una población de vacas Jersey. Estudios sobre ganado Creole nativo y Holstein en Argentina no mostraron pérdida del equilibrio al analizar ambas poblaciones diferenciadas (Golijow et al., 1999) ni en las poblaciones de Creole Argentinas y Bolivianas (Lirón et al., 2002). En ninguno de los estudios se encontraron desvíos significativos del equilibrio de Hardy–Weinberg.
En Colombia Echeverri et al. (2010) no encontraron equilibrio de Hardy–weinberg usando el gen PRL en vacas Holstein de 8 hatos diferentes, pero usando un número muy pequeño de animales. Por el contrario Usme et al. (2004) encontraron equilibrio en una población de 145 hembras Holstein de Antioquia (Colombia), pero usando el gen de Kappa–Caseína. Del mismo modo Rosero (2009) llegó a la misma conclusión evaluando una población de 30 animales en el Valle del Cauca (Colombia) usando en gen de Kappa–Caseína y β–Lactoalbúmina.
La heterocigosidad observada en el presente trabajo estuvo entre 0.217 a 0.382 y la esperada entre 0.133 a 0.394, lo cual sugiere una gran variabilidad entre los diferentes municipios. Además no se encontraron diferencias significativas (p>0.05) entre ambas heterocigosidades en ninguno de los municipios, aunque Envigado presentó diferencia considerable entre los valores de Ho y He, con un resultado de significancia aproximado a 0.05, lo cual indica una tendencia de apareamiento endogámico, que puede ser evidenciado por el valor de FIS de 0.6056. Sin embargo en el resto de las poblaciones no es posible afirmar que haya déficit o exceso de heterocigotos. Resultados similares se encontraron en el departamento de Nariño (Colombia) usando el gen de Kappa–Caseína, con
valores de Ho de 0.30 y He de 0.32 (Solarte et al., 2009). En otros estudios sobre Holstein Colombiano se encontró una Ho de 0.567 y He de 0.457 como promedio de las variantes génicas de Kappa Caseína, α–Lactoalbúmina, y β–Lactoalbúmina (Rosero, 2009).
En la presente investigación las frecuencias alélicas variaron entre las diferentes poblaciones desde 0.558 a 0.876 (Tabla 3) para el alelo A, con un valor promedio para la población total de 0.833 y entre 0.124 a 0.442 con un valor medio de 0.167 para el alelo B. Los rangos obtenidos permiten observar una gran variabilidad entre diferentes zonas del departamento de Antioquia (Colombia), sugiriendo gran diversidad genética entre algunos Municipios. Los datos anteriores se encuentran entre los rangos de valores obtenidos en otros estudios, que muestran frecuencias alélicas entre 0.76 (Klauziñska, 2002) y 0.93 (Mehmannavaz et al., 2009) para el alelo A. Sin embargo, el rango de variabilidad encontrado en las poblaciones de Antioquia es grande con respecto a los reportes encontrados en diferentes poblaciones del mundo (Brym et al., 2005; Mehmannavaz et al., 2009; Aijun et al., 2010) y los valores mínimos sólo se asemejan a los encontrados en el ganado Holstein rojo (f(A)=0.58 y f(B)=42) (Wojdak et al., 2008).
Lasfrecuenciasgenotípicasencontradas en los diferentes municipios y en la población total mostraron una tendencia a la prevalencia del genotipo AA con muy poca probabilidad de encontrar individuos homocigotos BB, como se reporta comúnmente en los análisis realizados en la raza Holstein (Dybus, 2002; Brym et al., 2005; Mehmannavaz et al., 2009). En Colombia Echeverri et al. (2010) reportaron valores similares a algunas de las frecuencias obtenidas en este trabajo, con frecuencias de 0.826 para el genotipo AA, 0.153 para el AB y 0.019 para el BB, pero usando una muestra de 153 vacas Holstein.
El Municipio de Marinilla fue el que presentó el valor más bajo para la frecuencia alélica de A, lo cual puede ser posible por el aislamiento que presenta este municipio, ya que no es un municipio reconocido como productor de leche y por el contrario se encuentra en un proceso de transición hacia urbano–industrial más que ganadero, por lo tanto las pequeñas producciones de leche que aún subsisten allí, pudieron haber tenido un proceso de diferenciación por el aislamiento y el uso de monta natural, lo que ha generado la presencia de estas frecuencias alélicas y genotípicas. Marinilla fue uno de los municipios que poseía menor número de animales (n=26), debido a la poca cantidad de hatos lecheros de la raza Holstein en la zona. El municipio de Santa Rosa de Osos presentó una frecuencia de 0.50 para el genotipo AA, lo cual se puede deber a la gran diversidad que se fue generando, teniendo en cuenta que esta es una población lechera importante que cuenta con hatos productores de leche a diferentes alturas. Sin embargo en esta zona no fue posible generar un muestreo más grande (n=16) que permita un mejor estimado del promedio de la población.
La diversidad genética medida en términos de heterocigosidad indica una variabilidad genética media (Tabla 4). Dybus et al. (2005) reportaron heterocigosidades cercanas a la media obtenida en el presente trabajo (Ho=0.2851). Resultados similares fueron encontrados en el Departamento de Nariño (Colombia) para una variante génica en Kappa–Caseína, con valores de Ho de 0.30 (Solarte et al., 2009).
Los valores obtenidos de FIS y FIT no fueron significativos posiblemente por la alta variabilidad encontrada dentro de las subpoblaciones y dentro de la población total, por lo tanto, no es posible alguna preferencia de apareamiento y aún con el uso de inseminación artificial no se ha afectado el equilibrio de Hardy–Weinberg en la población de Antioquia.
La población total presentó una diferenciación muy pequeña pero significativa (FST=0.02067), es decir que por lo menos dos de las poblaciones analizadas son diferentes entre sí, por lo cual Antioquia no se comporta como una única población y por el contrario presenta subpoblaciones entre las que se destaca Marinilla por ser la población más diferente y en la que mayor proporción de individuos BB se encontró. Los valores de FST pareados permitieron corroborar cuales de estos municipios presentan diferenciación entre sí (Tabla 5) y de este modo se pudo identificar aquellos municipios que presentan la mayor diferenciación con respecto a los demás. Entre estas poblaciones sobresale Marinilla que difieren con 5 municipios, además presenta el mayor valor de FST pareado (FST= 0.296) con San Pedro de los Milagros, evidenciando una alta diferenciación entre ambos municipios. Tal diferenciación en Marinilla se pudo haber generado por el aislamiento productivo, por el uso de monta natural y por la baja presión de selección para volumen de producción que se ha presentado en esta zona, por tanto se ha favorecido la mayor frecuencia del alelo B.
Los programas de inseminación, en los que es posible integrar semen de toros de todo el mundo, derriban las barreras geográficas y aumentan el flujo genético, pero en algunos casos los sistemas de producción no permiten que todas las poblaciones tengan acceso a estos programas y se pueden generar barreras de flujo entre poblaciones y por lo tanto una diferenciación genética.
Los resultados obtenidos en el presente trabajo permiten postular al gen de PRL como un importante marcador para la evaluación de características de importancia económica en Colombia, ya que este no parece haber sido sometido a presiones de selección muy fuertes, presenta frecuencias alélicas muy variables, tienen gran potencial por su papel importante en la lactancia y puede generar resultados rápidamente si se encuentra un genotipo superior con respecto a algunas características de importancia económica. Por lo tanto, para el desarrollo de programas de selección asistida por marcadores moleculares en Colombia, es necesario realizar la evaluación a nivel local, debido a que internacionalmente los reportes obtenidos en la raza Holstein son ambiguos, ya que en algunos lugares el genotipo BB es asociado a mejores características lácteas (Mehmannazav et al., 2009) y en otros lugares atribuyen mejores cualidades al genotipo AA (Dybus, 2002; Brym et al., 2005), por lo tanto surge la necesidad de determinar las asociaciones presentes entre los genotipos y algunas características productivas como la producción de leche, el porcentaje de grasa, el porcentaje de proteína y el conteo de células somáticas en Colombia.
Agradecimientos
Se agradece a los propietarios de los hatos en cada municipio por permitir la realización de este estudio y al Laboratorio de Biología Celular y Molecular de la Universidad Nacional de Colombia, sede Medellín.
Referencias
1. Aijun LU, Xiucai H, Hong L, Chen A, Jihong J, Chunlei Z, Haixia X, Xueyuan G. Single nucleotide polymorphisms in bovine PRL gene and their associations with milk production traits in Chinese Holsteins. Mol Biol Rep 2010; 37:547–551. [ Links ]
2. Alipanah M, Kalashnikova L, Rodionov G. Association of prolactin gene variants with milk production traits in Russian Red Pied cattle. Iran J Biotechnol 2007; 5:158–161. [ Links ]
3. Bedoya G, Carvajal LG, Bermúdez NR, Moreno FL, Márquez ME, Davies S, Derr J, Ossa J, Ruiz A. Estructura molecular y poblacional del Ganado criollo Colombiano (GCC). Rev Col Cienc Pec 2001; 14:109–120. [ Links ]
4. Brym P, Kaminski S, Wójcik E. Nucleotide sequence polymorphism within exon 4 of the bovine prolactin gene and its associations with milk performance traits. J Appl Genet 2005; 45:179–185. [ Links ]
5. Brym P, Malewski T, Starzynski R, Flisikowski K, Wojcik E, Rusc A, Zwierzchowski L, Kaminski S. Effect of New SNP Within Bovine Prolactin Gene Enhancer Region on Expression in the Pituitary Gland. Biochem Genet 2007; 45:743–754. [ Links ]
6. Cao X, Wang Q, Yan JB, Yang FK, Huang SZ, Zeng YT. Molecular cloning and analysis of bovine prolactin full–long genomic as well as cDNA sequences. Yi Chuan Xue Bao 2002; 29:768–773. [ Links ]
7. Dybus A. Association of Growth Hormone and Prolactin genes polymorphisms with milk production traits in Polish Black and White Cattle. Anim Sci 2002; 20:203–212. [ Links ]
8. Dybus A, Grzesiak W, Kamieniecki H, Szatkowska I, Sobek Z, Blaszczyk P, Piatkowska E, Zych S, Muszynska M. Association of genetic variants of bovine prolactin with milk production traits of Black–and–White and Jersey cattle. Arch Tierz 2005; 48:149–156. [ Links ]
9. Echeverri J, Vásquez N, Gallo Y. Efecto de la transición Adenina/Guanina del gen de la prolactina bovina sobre características de importancia en producción lechera. Rev Lasallista Investig 2010; 7:16–23. [ Links ]
10. Excoffier L, Laval G, Schneider S. Arlequin ver. 2.0: An integrated software package for population genetics data analysis. Evolutionary Bioinfo Online 2005; 1:47–50. [ Links ]
11. Freeland J. Molecular Ecology. 1st edition. Chichester, West Sussex: John Wiley y sons Ltd; 2005. p.1–60. [ Links ]
12. Freemann ME, Kanyicska B, Lerant A, Nagy G. Prolactin: structure, function and regulation of secretion. Physiol Rev 2000; 80:1523–1631. [ Links ]
13. Golijow CD, Giovambattista G, Rípoli MV, Dulout FN, Lojo MM. Genetic variability and population structure in loci related to milk production traits in native Argentine Creole and commercial Argentine Holstein cattle. Genet Mol Biol 1999; 22:395–398. [ Links ]
14. Guo SW, Thompson EA. Performing the exact test of Hardy– Weinberg proportions for multiple alleles. Biometrics 1992; 48:361–372. [ Links ]
15. Hartl DL, Clark AG. Principles of population genetics. 4th edition. Sunderland (MA): Sinauer Associates Inc Publishers; 2007. p.5–138. [ Links ]
16. Khatami SR, Lazebny OE, Maksimenko VF, SulimovaGE. Association of DNA polymorphisms of the growth hormone and Prolactin genes with productivity in Yaroslavl and Black and White cattle. Russian J Genet 2005; 41:167–173. [ Links ]
17. Klauziñska M. Polimorfizm regionów 5'–flankuj1cych genów GH, GHRH, prolaktyny i miostatyny byd3a. [Polymorphism of 5'–flanking regions of genes GH, GHRH, prolactin, and cattle miostatin.]. PhD thesis. Institute of Animal Genetics and Breeding, Jastrzêbiec, Poland. 2002. [ Links ]
18. Lirón JP, Ripoli MV, De Luca JC, Peral–Garcia P, Giovambattista G. Analysis of genetic diversity and population structure in Argentine and Bolivian Creole cattle using five loci related to milk production. Genet Mol Biol 2002; 25:413–419. [ Links ]
19. Mehmannavaz Y, Amirinia C, Bonyadi M, Vaez–Torshizi R. Effects of bovine prolactin gene polymorphism within exon 4 on milk related traits and genetic trends in Iranian Holstein Bulls. Afr J Biotechnol. 2009; 8:4797–4801. [ Links ]
20. Miller SA, Dykes DD, Polesky HF. A simple salting–out procedure for extracting ADN from human nucleated cells. Nucleic Acid Res 1988; 16:1215. [ Links ]
21. Rosero AJ. Polimorfismo de los genes–Caseína, β–Lactoglobulina y α–Lactoalbúmina en razas bovinas criollas colombianas. Tesis de maestría en Ciencias Agrarias. Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia, Palmira, 2009. [ Links ]
22.. SAS Institute Inc. SAS/STAT User's Guide: Version 9.2 5th edition. SAS Institute Inc., Cary, North Carolina, USA. 2003. [ Links ]
23. Solarte CE, Rosero C, Eraso JM, Zambrano GL, Cárdenas H, Burgos WO. Frecuencias alélicas del gen Kappa Caseína en la raza Holstein del trópico alto de Nariño – Colombia. LRRD 2009, 21: 3. [Fecha de acceso: 6 de diciembre de 2011] URL: http://www.lrrd.org/lrrd21/1/sola21003.htm. [ Links ]
24. Usme J, Restrepo F, Trujillo–Bravo E. Kappa–Caseína Bovina y su asociación con el recuento de células somáticas en ganado Holstein. Actual Biol 2004; 26:17–22. [ Links ]
25. Wojdak K, Kemic M, Strzalaka J. Prolactin Gene Polymorphism and Somatic Cell Count in Dairy Cattle. J Anim Vet Adv 2008; 7:35–40. [ Links ]
26. Wright S. Evolution and Genetics of Populations, The theory of gene frequencies. 2nd volume. Chicago (IL): University of Chicago Press; 1969. p.211–455. [ Links ]
Notas
¤ To cite this article: Rincón JC, López A, Echeverri J. Variabilidad genética del loci RsaI de la prolactina bovina en Ganado Holstein de Antioquia, Colombia. Rev Colomb Cienc Pecu 2012; 25:191–201.