Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Colombia Forestal
Print version ISSN 0120-0739
Colomb. for. vol.14 no.2 Bogotá July/Dec. 2011
CARACTERIZACIÓN DE LA VARIABILIDAD GENÉTICA DE PROGENIES DE Cordia alliodora (R. & P.) Oken1
Genetic variability of Cordia alliodora (R. & P.) Oken progenies
Caracterização da variação genética de progênies de Cordia alliodora (R. & P.) Oken
Marta Leonor Marulanda2, Ana María López3, Marcela Uribe4 & Carlos Mario Ospina5
1 Proyecto financiado por el Ministerio de Agricultura y Desarrollo Rural de la República de Colombia bajo el código 343 derivado del convenio 002 del 2006 y Gobernación de Risaralda.
2Laboratorio de Biotecnología Vegetal. Universidad Tecnológica de Pereira. ubioteve@utp.edu.co. Autora correspondencia.
3Laboratorio de Biotecnología Vegetal. Universidad Tecnológica de Pereira. ubioteve@utp.edu.co.
4Laboratorio de Biotecnología Vegetal. Universidad Tecnológica de Pereira. ubioteve@utp.edu.co.
5Centro Nacional de Investigaciones de café (Cenicafé). carlosmario.ospina@cafedecolombia.com
Recepción: Septiembre 9 de 2010/Aprobación: Julio 25 de 2011
RESUMEN
Cordia alliodora es un árbol muy conocido como productor de madera en zonas tropicales de América Latina y el Caribe; se caracteriza por producir una valiosa madera y presentar crecimiento rápido. En Colombia, es frecuente en sistemas agroforestales con café. Como la mayoría de especies forestales, esta especie presenta problemas biológicos para programas de mejoramiento genético como los largos periodos de regeneración y los altos costos a la hora de mantener una población a largo plazo. Los programas de selección asistidos con marcadores moleculares han tenido un gran impacto en el mejoramiento genético, debido a que minimizan los intervalos de regeneración, incrementan la ganancia genética por generación y permiten la evaluación de la información genética esencial para las especies. En el presente trabajo, se caracterizaron sesenta individuos de C. alliodora pertenecientes a los ensayos de procedencias y progenies establecidos en el programa de mejoramiento genético de Cenicafé. La caracterización se realizó a través de marcadores microsatélites, después del desarrollo de una librería genómica enrriquecida con microsatelites de la especie. Finalmente, se evaluaron veinticuatro microsatélites específicos, de los cuales veinte permitieron detectar veintiocho loci polimórficos y multialélicos. Estos resultados ofrecen una guía para orientar las políticas de producción sostenible y conservación de esta especie de valiosa madera, así como una herramienta para la identificación de clones con interés comercial.
Palabras clave: Boraginaceae, Cordia, ensayo de procedencias y progenies microsatélites, Nogal cafetero, SSR.
ABSTRACT
Cordia alliodora is a well-known wood producer tree of tropical areas of Latin America and the Caribbean characterized for producing valuable wood and by its fast growth rate. In Colombia, it is frequent on agro-forestal systems with coffee. This species, like most forest species have biological problems for genetic improvement programs, such as long regeneration periods and high costs for supporting a population in a long term. The molecular assisted markers in plant breeding programs have had a great impact on genetic improvement, due to the fact they minimize their intervals of regeneration, increase the genetic gain by generation and allow the evaluation of the genetic information essential for the species. In this work, 60 genotypes of C. alliodora were characterized, belonging to the provenance and progenies tests established by the program of genetic improvement of Cenicafé. The characterization was carried out through microsatellite markers, after developing a genomic library enriched with microsatellites of the species. Finally, 24 specific microsatellites were evaluated, 20 of which allowed the detection of 28 polymorphic and multiallelic loci. These results provide a guide for orienting the policies of sustainable production and conservation of this valuable species; also, it provides a useful tool for the identification of clones with commercial interest.
Key words: Boraginaceae, Cordia, provenance and progeny trials, microsatellites, Nogal cafetero, SSR.
RESUMO
Cordia alliodora é uma árvore muito conhecido como produtora de madeira em zonas tropicais da América latina e o Caribe são caracterizados por produzir uma valiosa madeira e ser de crescimento rápido. Na Colômbia é freqüente em sistemas agro florestais com café. Esta espécie como a maioria das espécies florestais, apresenta problemas biológicos para programas de melhoramento genético como os largos períodos de regeneração e os altos custos para manter um numero de espécie num largo prazo. Os programas de seleção assistidos com marcadores moleculares tiveram um grande impacto no melhoramento genético, devido que, minimizam os intervalos de regeneração, incrementam o ganho genético pela geração e também permitem uma avaliação da informação genética essencial para as espécies. No presente trabalho são caracterizados 60 indivíduos de C. alliodora pertencentes aos ensaios de procedências e progênies estabelecidos no programa de melhoramento genético de Cenicafé. A caracterização foi realizada a través de marcadores micro satélites, depois do desenvolvimento de uma livraria genomica enrriquecida com microsatelites da especie. Finalmente foram avaliados 24 microsatélites específicos os quais 20 permitiram detectar 28 loci polimórficos e multialélicos. Estes resultados oferecem uma guía para orientar as políticas de producao sustentável e conservacao desta especie de valiosa madeira, assim como uma ferramenta valiosa para a identificacao dos clones com interesse comercial.
Palavras chave: Boraginaceae, Cordia, ensaio de procedências e progênies, microsatélites, Nogal cafeteiro, SSR.
INTRODUCCIÓN
Las plantaciones forestales están desplazando rápidamente la explotación de los bosques naturales del mundo (Gartland et al. 2002); el área total en bosques naturales es aproximadamente de 3.9 billones de hectáreas (Bha), lo que representa el 30% de la superficie de la tierra y el consumo de madera industrial supera los 1600 billones de m3 en el mundo (Carson et al. 2004). Las Naciones Unidas anticipan que para el año 2030 entre el 50 y el 75% de la producción de madera para uso industrial del mundo procederá de plantaciones forestales; por su parte Gartland et al. (2002) afirman que esta es la mayor oportunidad para la aplicación a gran escala de la biotecnología en especies forestales, investigando sobre las especies de más rápido crecimiento. Los árboles de rotación corta serán los más adaptables al trópico, debido a que las plantaciones forestales se han desplazado desde el hemisferio norte a las zonas tropicales y subtropicales a ambos lados del Ecuador (Carson et al. 2004).
La especie Cordia alliodora (R. & P.) Oken pertenece a la familia Boraginaceae y es un árbol muy conocido como productor de madera en zonas tropicales de América Latina y el Caribe. Se caracteriza por ser una especie de porte alto, hermafrodita y de polinización cruzada. Su distribución en el continente americano es muy amplia: se encuentra desde México (25° N) a lo largo de Centro y Sur América hasta Bolivia, el sur de Brasil y el norte de Argentina (25° S) (Boshier 1995, Boshier et al. 1995a, 1995b, Chase et al. 1996, Boshier & Henson 1997,Sebbenn et al. 2002, 2005, 2007). En Colombia, esta especie se encuentra distribuida en los valles interandinos de las tres cordilleras y la Sierra Nevada de Santa Marta, desde el nivel del mar hasta los 1900 msnm, recibiendo varios nombres según la región.
Hasta el momento, las actividades de mejoramiento en Cordia alliodora han sido realizadas principalmente en Centro América, Colombia y Brasil. En Colombia (Cenicafé 2000), se han empleado marcadores moleculares como apoyo al conocimiento de la diversidad genética presente en poblaciones naturales. Las técnicas de biología molecular, en particular, el uso de marcadores moleculares han permitido conocer y caracterizar el contenido genético de los organismos, así como estimar la diversidad y las relaciones entre grupos de interés (Phillips-Mora 1995). Uno de los usos más generalizados de los marcadores moleculares en los programas de mejoramiento genético es la identificación y la selección de individuos (O’Malley et al. 1996), haciendo la selección más confiable y precisa.
Los estudios en poblaciones de árboles han ido en aumento a partir del desarrollo de los marcadores microsatélites (SSR), gracias a que estos son muy polimórficos y están ampliamente distribuidos en el genoma de especies animales y vegetales (Daynandan et al. 1997, Collevatti et al. 1999). Los loci microsatélites contienen una secuencia simple (mono, di, tri o tetra) que se repite; el número de repeticiones por microsatélite puede ser altamente variable y la naturaleza del marcador es codominante (Buso et al. 2006). En estudios de especies forestales tropicales, la utilización de los microsatelites ha tenido importantes consecuencias para la conservación y uso sostenible de los recursos forestales; así mismo, han sido herramientas importantes para generar información detallada de la diversidad genética y la estructura de la población en especies que probablemente están siendo sobreexplotadas en áreas como la Amazonía, la selva pacífica, los bosques andinos y el cerrado brasileño. A su vez, estos datos ayudan en la elaboración de estrategias para la conservación y aprovechamiento forestal sostenible.
Los objetivos del presente trabajo fueron: identificar marcadores (SSR) que permitan un análisis en los individuos en C. alliodora, para relacionar marcadores con características morfológicas de interés comercial; desarrollar huellas digitales de las diferentes progenies y árboles élite de C. alliodora que permitan su caracterización; y examinar la variación genética entre las progenies de diferentes procedencias.
MATERIALES Y MÉTODOS
MATERIAL VEGETAL
En el año 1997, el Centro Nacional de Investigaciones de Café (Cenicafé) seleccionó en el país 101 árboles “plus”, junto con trece procedencias obtenidas de cuatro países centroamericanos, como base para el programa de selección y mejoramiento genético de especies forestales de interés asociadas al café.
Los criterios de selección de estos árboles plus se basaron en las características fenotípicas que están bajo el mayor control genético aditivo posible, tales como: fuste cilíndrico, recto, libre de rajaduras, sin acanalamientos, sin aletones, ni bifurcaciones. Copa pequeña, angosta y simétrica. Ramas delgadas y distribuidas asimétricamente en verticilos separados. Vigor y dominancia sobre los demás individuos de la población y libre de plagas y enfermedades.
De estos 101 individuos, para el presente estudio, se seleccionaron sesenta, que arrojaron los mejores resultados de las evaluaciones morfológicas, dispuestos en ensayo de procedencias y progenies a lo largo del país. Asi mismo, se muestrearon individuos procedentes de huertos clonales. De los individuos seleccionados, se colectaron hojas jóvenes y sanas. Las muestras de hojas se colectaron y almacenaron en bolsas herméticas con gel de sílice (proporción 1:10, tejido vegetal: gel de sílice); posteriormente, se transportaron al Laboratorio de Biotecnología Vegetal de la Universidad Tecnológica de Pereira. En la tabla 1 se describen los códigos y las procedencias de los arboles estudiados.

EXTRACCIÓN DEL ADN
Para la extracción del ADN se utilizaron dos métodos, el primero con el kit de extracción de ADN de plantas de QIAGEN DNA easy Plant mini kit®, siguiendo las indicaciones del fabricante. El otro método fue el protocolo de extracción de Doyle & Doyle (1990) modificado por Márquez (2003) para la extracción de ADN de Cordia alliodora.
DESARROLLO DE LIBRERÍAS ENRIQUECIDAS PARA LA OBTENCIÓN DE MICROSATÉLITES
Para la construcción de la librería enriquecida, se siguió el procedimiento descrito por Jones et al. (2002). Las secuencias que contenían las zonas microsatélites fueron ensambladas y editadas en Seqman (DNAStar). Finalmente, se diseñaron iniciadores para llevar a cabo la amplificación por PCR, a través del programa DesignerPCR, version 1.03 (Research Genetics, Inc).
AMPLIFICACIÓN DEL ADN
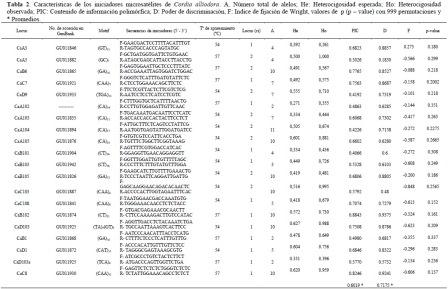
Para la caracterización de las muestras se utilizaron veinte marcadores microsatélites (Tabla 2). Las reacciones se llevaron a cabo en un volumen final 15 µl con 1X de buffer de amplificación (10 mM de Tris HCl, 50 mM de KCl), 200 µM de una mezcla de dNTPs, 2 mM de cloruro de magnesio, 1 µM de cada uno de los primers, 0.5 U de taq polimerasa y 10 ng de ADN genómico. El perfil de amplificación fue de treinta ciclos de 94 °C durante un minuto, un minuto a la temperatura de apareamiento (Tabla 2) y finalmente un minuto a 72 °C, con una extensión final a 72 °C durante 10 minutos. La separación de los productos amplificados se realizó mediante electroforesis vertical en geles de poliacrilamida al 6%, corridos en cámara de electroforesis Sequi-Gen de BioRad, a 110W, los geles fueron teñidos con nitrato de plata siguiendo el protocolo de Benbouza et al. (2006).
ANÁLISIS ESTADÍSTICO
Los análisis estadísticos se realizaron con el programa GENAlEX versión 6.2 (Peakell & Smouse 2006). El análisis comprendió la determinación de medidas de variabilidad genética, diversidad y distancia genética. Además, se determinó el poder de discriminación de los marcadores utilizados (D) a través de la fórmula de Tessier et al. (1999) y de la probabilidad de identidad.
EVALUACIÓN DE CARACTERES MORFOLÓGICOS
Los datos morfológicos de los árboles estudiados fueron tomados por el grupo de investigación en forestales de CENICAFE (Ospina et al. 2008), las variables cuantificadas fueron: longitud de la hoja (HL), ancho de la hoja (HA), número de ramas por verticilo (NRV), diámetro de la copa norte sur (DCNS), diámetro de la copa este oeste (DCEO), altura (medida en metros), diámetro a la altura del pecho (DAP) (medida en metros), supervivencia (medida en porcentaje) y volumen con corteza (VCC). Para esto se utilizó la fórmula que se presenta a continuación (Ospina et al. 2008):
VCC=0.0193+[0.0000361(DAP2*Altura)].
Se calculó la distancia euclidiana (Smith & Smith 1992) entre cada par de genotipos con los datos morfológicos, a partir de la distancia genetica de Nei (1972) y se construyó un dendrograma con el método de agrupación UPGMA (media aritmética no ponderada) utilizando el programa NT-Sys. Versión 2.02 (Rohlf 1989)
RESULTADOS
DESARROLLO DE LIBRERÍAS ENRIQUECIDAS
En total, se identificaron 109 secuencias microsatélites, que fueron dispuestas en la base de datos pública del National Center for Biotechnology Information (NCBI-GenBank). Los datos fueron enviados directamente a través del programa SEQUIN versión 9.50, diligenciando la información pertinente para tal fin. Los números de accesión de las 109 secuencias comprendieron los códigos GU011824 al GU011945. Estas 109 secuencias permitieron el diseño de 74 iniciadores microsatélites; de éstos, veinticuatro fueron sintetizados. Finalmente, para esta caracterización, se seleccionaron veinte iniciadores (Tabla 2), dadas sus características de reproducibilidad, calidad en la amplificación y polimorfismo.
ANÁLISIS DE LA VARIABILIDAD GENÉTICA
Los veinte SSR analizados produjeron amplificación positiva y mostraron polimorfismos en varios alelos. El marcador CaA104 produjo el mayor número de alelos (once), seguido de CaA105, CaB102,CaD103 (Tabla 2);los marcadores que presentaron el menor número de alelos (dos) fueron CaB1, CaD103ay CaA102. De acuerdo con la variabilidad genética presente y el elevado multialelismo que se encontró en los veinte microsatélites, se detectaron veintiocho loci polimórficos.
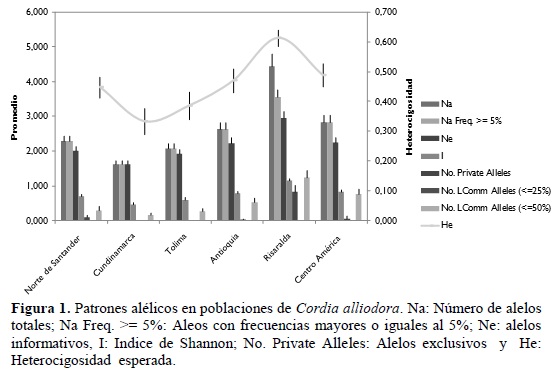
En la figura 1, se observa la distribución de los diferentes alelos detectados en los veintiocho loci microsatélites. Se obtuvieron los parámetros genéticos como el promedio de alelos por locus (Na), el número de alelos informativos (Ne) y la heterocigosidad esperada (He) en las seis poblaciones y en los sesenta individuos. La heterocigosidad esperada (He) fue mayor en la población de Risaralda, seguida por las poblaciones de Centro América y Antioquia (Figura 1). También se observa la presencia de alelos exclusivos en la poblaciones de Risaralda y en menor número en la población de Norte de Santander, la presencia de alelos con frecuencias iguales o inferiores al 50% (alelos de baja frecuencia) se presentaron en mayor número en las poblaciones de Risaralda y Centro América.
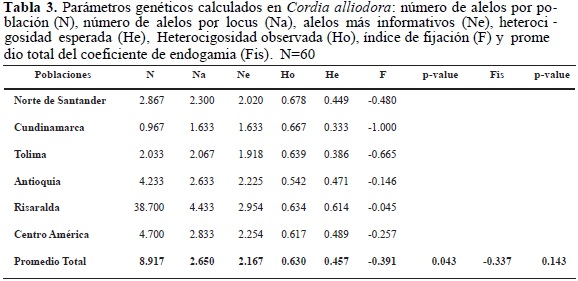
Los parámetros genéticos promedio para todas las poblaciones en todos los loci se observan en la tabla 3. El promedio de alelos por locus osciló entre 1633 para la población de Cundinamarca y Centro América 4.70, mientras las demás poblaciónes presentaron los siguientes promedios de alelos por locus: Antioquia 2633, 4433 para la de Risaralda, Norte de Santander 2867 y Tolima 2033; el promedio de alelos informativos (Ne) fue de 1633 para Cundinamarca y 2954 para Risaralda, siendo estos los menores y mayores valores respectivamente. Los valores de heterocigocidad esperada (He) y observada (Ho) para Risaralda fueron de 0.614 y 0.634, respectivamente; estos fueron los valores más altos con respecto a todas las demás poblaciones. El índice de fijación (F) fue -0.045 para el departamento de Risaralda, este fue el valor más alto, siendo el de menor valor el de la población de Cundinamarca (-1.00), todas las poblaciones mostraron valores negativos (Tabla 3). A pesar de que los árboles de C. alliodora —objeto de esta investigación— hacen parte de una población seleccionada para mejoramiento, el valor del índice de fijación (F) para C. alliodora estuvo por debajo de cero, lo cual muestra una baja consanguinidad entre los árboles estudiados. Entre tanto, el coeficiente de endogamia (Fis) arrojó un valor promedio de -0.337; ambos resultados (F y Fis) demuestran que el programa de selección asistida con marcadores moleculares AFLP y RAPD (Marulanda et al. 2002, Márquez 2003) que ha apoyado el programa de mejoramiento de Cenicafé en el pasado ha sido muy útil en la selección de los árboles para el establecimiento de los huertos semilleros, pues ha permitido seleccionar árboles no emparentados entre sí (Tabla 3).
Los promedios para todas las poblaciones y todos los loci fueron: promedio de alelos por locus (Na) de 2.650;heterocigosidad esperada (He) de 0.457; heterocigosidad observada (Ho) de 0.630; el índice de fijación (F) fue -0.391 y los alelos informativos (Ne) de 2167. En el caso de las veinte secuencias SSR de C. alliodora evaluadas, siete fueron secuencias de tri-nucleótidos: CaC7, CaC8, CaD9, CaC108, CaD103a, CaD1 y CaC103, y una pertenece a un microsatélite imperfecto (CaD103). Las doce secuencias restantes fueron microsatélites dinucleótidos que fueron todos informativos y polimórficos (Tabla 2).
MEDIDAS DE DIVERSIDAD GENÉTICA
Los parámetros de diversidad genética encontrados al analizar 28 loci permitieron identificar poblaciones con baja consanguinidad, marcadores multialélicos y heterocigosidades esperadas iguales a las observadas.
El análisis de varianza molecular (AMOVA) (Tabla 4) reveló que el mayor porcentaje de variación lo aportaron las diferencias en las poblaciones (95%), seguida de la variación entre poblaciones (5%). Estos datos ratifican que una parte importante de la variación va a estar en las poblaciones y que esta característica unida a datos de alelos exclusivos, distancias genéticas importantes y la asociación a características morfológicas de interés determinarán la permanencia de los materiales en el programa de mejoramiento genético.

MEDIDAS DE DISTANCIA GENÉTICA
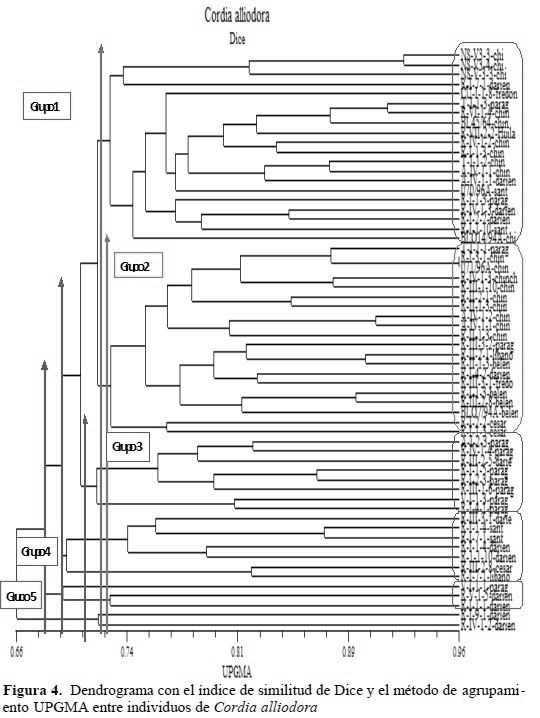
La distancia genética entre individuos se puede evidenciar en la figura 4, en la que se forman cinco grupos; todos los grupos presentan individuos risaraldenses. La distancia genética entre las poblaciones (Tabla 5) indica que las poblaciones con mayores distancias genéticas las presentan las poblaciones de Tolima y Centro América (0.447) y de Tolima con Norte de Santander (0.436); entre tanto, las menores distancias genéticas están entre Risaralda y Centro América (0.130) y Antioquia y Risaralda (0.197).

En el análisis de coordenadas principales, basado en las distancias genéticas entre los individuos (Figura 2), se observa una mayor cercanía entre los materiales risaraldenses y los centroamericanos. Algunos materiales antioqueños, norte santandereanos y tolimenses se encuentran separados del grupo de individuos risaraldenses y centroamericanos. En el análisis de coordenadas principales, la mayor cantidad de individuos se agrupan en el primer y cuarto cuadrante, mientras tanto, los individuos más separados genéticamente se encuentran en el segundo y tercer cuadrante.

PROBABILIDAD DE IDENTIDAD
En C. alliodora, la combinación de doce microsatélites permite la obtención de una probabilidad única para un individuo. La figura 3 muestra cómo a partir de la combinación de cinco loci, el número de individuos con un genotipo único comienza a aumentar mientras desciende el número de individuos con genotipos iguales. La mayor variabilidad de los microsatélites, en comparación con otros marcadores, aumenta la probabilidad de que cada individuo en una población tenga un genotipo único, haciendo que los microsatélites sean muy útiles en el desarrollo de huellas digitales, estudios sobre el flujo de polen o la dispersión de semillas. Además, estos marcadores son sensibles a cambios en el tamaño de las poblaciones de mejoramiento, a cambios en la estructura de las poblaciones, así como cambios en las tasas de dispersión (Slatkin 1995).

EVALUACIÓN MORFOLÓGICA
Las distancias euclideanas agrupadas por UPGMA se observan en un dendrograma (Figura 5), en el que se evidencia la formación de tres grupos de similitud. En este dendrograma se observa la formación de un grupo con mayores diferencias con respecto al resto de las progenies y está compuesto por: R-III-2-8C; A-IV-1-1C; R-III-2-8-R; R-II-1-3C y 070/96AS. Estas progenies se caracterizan por los mayores valores de DAP. El grupo 1 se identifica por los genotipos con mayores valores de DAP y altura al mismo tiempo, es decir, los individuos con mejores características forestales (RI- 1-10S, R III-1-10C, RI 1-3R, RI-VI-3C, RI-1-4S, NSV3-3C, BL7/94AR).

DISCUSIÓN
En los análisis de la variabilidad genética, los veintiocho loci obtenidos en C. alliodora son altos, si se comparan con los obtenidos en otras especies de árboles como: Quercus myrsinifolia en los que se detectaron nueve loci; Q. petraea en el que se obtuvierondiecisiete loci; en Shorea curtissii se obtuvieronnueve loci; en Symphonia globulifera se detectaron tres loci; en Swietenia humilis se obtuvieron tres loci (Isagi & Suhandono 1997, Steinkellner et al. 1997, Ujino et al. 1998, Aldrich et al. 1998, White & Powell 1997a, 1997b).
En otras especies forestales, en las cuales se han situado microsatélites en mapas genéticos y se ha descrito el número de loci, estos últimos están muy en concordancia con los obtenidos en el presente estudio de C. alliodora; por ejemplo, en Eucalyptus grandis x E. urophylla en el que se obtuvieron diecinueve loci, en Pinus radiata se detectaron dieciséis loci; en Pinus taeda se obtuvieron once loci, en Quercus robur se obtuvieron dieciocho loci y en Acacia mangium se obtuvieron treinta loci (Echt & Nelson 1997, Brondini et al. 1998, Barrenche et al. 1998, Butcher et al. 1999, Devey et al. 1999). Los datos obtenidos con microsatélites indican que, además de ser generalmente más informativos que otros tipos de marcadores, los loci microsatélites están dispersos por igual en todo el genoma (Butcher et al. 1999).
En relación con las medidas de diversidad genética, Miller & Schaal (2006) analizaron la estructura de las poblaciones naturales y cultivadas de una especie forestal de fruto comestible (Spondias purpurea) y encontraron queel porcentaje de variación entre poblaciones naturales fue del 35.65% y al interior de las poblaciones del 64.35%, mientras que en materiales cultivados son de 30.19% y 69.81% respectivamente. Estos resultados explican, en parte, que la mayor variación para C. alliodora sea mayor en las poblaciones (95%). Hamrick et al. (1979) observaron una notable heterogeneidad en las especies para niveles de variación dentro de las poblaciones. Una proporción significativa de esta variación ha sido asociada con la historia de vida y las características ecológicas de las especies. Estas especies están ampliamente dispersas, perennes, de polinización cruzada, anemófilas y un alto número de cromosomas están relacionadas con los mayores niveles de variación intrapoblacional.
Las heterocigosidades promedio obtenidas en todos los loci en esta investigación en C. alliodora fueron elevadas si se comparan con los trabajos en Eucalyptus spp. de Grattapaglia & Kirst (2008) quienes desarrollaron microsatélites con tetra y pentanucleótidos, que les permitieron obtener heterocigosidades por encima de 0.45. Estos autores consideran que los perfiles de ADN de los árboles analizados fueron significativamente más robustos, que cuando utilizaron secuencias microsatélites dinucleótidas.
Las medidas de distancia genética muestran algúna agrupación de las progenies con respecto a su origen geográfico o procedencia y están en mayor concordancia con los reportados por Boshier et al. (1995a) en C. alliodora en bosques tropicales en Costa Rica, quienes demostraron qué árboles cercanos están más relacionados genéticamente en comparación con árboles distantes. En contraste, los resultados obtenidos en esta investigación difieren a los reportados por Marulanda et al. (2000) y Márquez (2003) en investigaciones anteriores con genotipos de C. alliodora de las mismas procedencias, utilizando marcadores RAPD y AFLP, en los cuales no se encontró agrupación de los genotipos de acuerdo con su origen geográfico. Es importante resaltar que los marcadores microsatélites tienen un mayor poder de resolución y que la población de árboles analizados es un grupo muy seleccionado, en el cual se ha ido reduciendo la base genética y se han realizado procesos de movilización de semillas de los mejores árboles, por parte de los agricultores, como lo reportó Márquez (2003). También es importante tener en cuenta que los estudios de Boshier (1995) y Boshier et al. (1995a, 1995b) se realizaron en bosques naturales relativamente bien conservados, mientras los trabajos realizados en Colombia han sido con árboles de relictos de bosques, bosques secundarios y en sistemas agroforestales asociados con café.
Por último, la probabilidad de identidad encontrada en el presente estudio arroja una herramienta invaluable para estudios de identificación de clones. Otros investigadores han demostrado el poder discriminatorio de los microsatélites como el caso de Chase et al. (1996) con el árbol tropical Pithecellobium elegans quienes pudieron distinguir entre el 80% de los individuos en una población con solo tres loci microsatélites, comparado con el 37% de los individuos, cuando utilizaron seis loci de isoenzimas (Butcher et al. 1999). Se han reportado importantes trabajos con microsatélites en otras especies forestales como en Q. robur en las cuales losmicrosatélites se utilizaron para determinar las relaciones genéticas entre árboles seleccionados (Lefort et al. 1998), esto confirma la relevancia de abordar este tipo de investigaciones tal y como se han realizado en el caso de C. alliodora. Utilizando nueve loci, se estableció que cinco árboles seleccionados no estaban estrechamente relacionados entre ellos, por lo tanto, constituirían una fuente de semillas adecuadas para un programa de mejoramiento avanzado. En un segundo estudio, nueve loci microsatélites se utilizaron para detectar contaminantes de semillas y confirmar las relaciones de familias de semillas de medios hermanos procedentes de un solo árbol de Quercus robur (Lexer et al. 1999). Los anteriores son ejemplos de la aplicación práctica de los marcadores microsatélites en programas de selección y mejoramiento en forestales. En el caso del eucalipto, la probabilidad de identidad (PI) con seis loci fue menor de 1 en 2000 millones (5 x 10-10). Los coeficientes de similitud calculados a partir de datos de microsatélites fueron mucho más pequeños, por lo tanto, más selectivos, que aquellos obtenidos en estudios similares con RAPD y AFLP (Grattapaglia 2003).
El alto poder de discriminación de los SSR y la aceptación este tipo de tecnología por parte de los mejoradores dio lugar a la inclusión de los marcadores moleculares como descriptores adicionales en los protocolos requeridos para la protección varietal (Brondani & Grattapaglia, 2002; Grattapaglia 2003). La inclusión de marcadores de ADN representó un avance notable en el panorama internacional de protección de variedades de árboles forestales; actualmente, todas las solicitudes para la protección clonal van acompañados de un perfil de ADN (huella de ADN) multilocus de quince a veinte microsatélites, los cuales se recomiendan basados en varios aspectos, como robustez, polimorfismo y disponibilidad de los SSR de dominio público. Las perspectivas para los siguientes años es que habrá un número creciente de solicitudes de protección de clones por parte de las empresas forestales en relación con el valor de clones élite de eucalipto manteniendo la competitividad de la industria silvicultora (Brondani & Grattapaglia 2002, Grattapaglia 2003).
CONCLUSIONES
Los árboles de C. alliodora objeto de esta investigación y que hacen parte de una población seleccionada presentan índices de fijación de Wright (F) inferiores a cero, lo que muestra una baja consanguinidad entre los árboles estudiados. Lo anterior también indica que el programa de mejoramiento de Cenicafé ha establecido huertos semilleros no emparentados entre sí. En C. alliodora, a partir de la combinación de doce microsatélites, se obtiene un 100% de los individuos con un genotipo único. En consecuencia, con los veinte microsatélites descritos en este artículo se entrega una herramienta de identificación clonal y protección varietal para C. alliodora.
De las veinte secuencias SSR de C. alliodora evaluadas, siete fueron secuencias de tri-nucleótidos, una pertenece a un microsatélite imperfecto (CaD103), las doce secuencias restantes fueron microsatélites dinucleótidos, todos mostraron ser informativos y polimórficos.
El análisis de varianza molecular permitió distinguir el gran aporte de los individuos a la variabilidad, superando los aportes que hacen las poblaciones sobre esta. Entonces, es preciso que, aunque el programa de mejoramiento demande la selección de materiales, se procure la conservación de materiales en campo y semillas de aquellos que no se seleccionen, esto a fin de evitar la pérdida de genes y alelos que podrían ser de importancia adaptativa de la especie.
Las caracterizaciones genéticas servirán en un futuro para direccionar polinizaciones controladas entre individuos con características morfológicas adecuadas y de similitudes genéticas bajas.
AGRADECIMIENTOS
Los autores expresan sus sinceros agradecimientos al Ministerio de Agricultura y Desarrollo Rural de la República de Colombia, a la Federación Nacional de Cafeteros de Colombia, a la Gobernación del Risaralda y a la Universidad Tecnológica de Pereira por la financiación de esta investigación. A la Administradora Ambiental y candidata a M. Sc. Juliana Arias Villegas por la administración de este proyecto. A todo el personal del Laboratorio de Biotecnología Vegetal de la Universidad Tecnológica de Pereira por sus valiosos aportes en todo el desarrollo de la presente investigación.
REFERENCIAS BIBLIOGRÁFICAS
Aldrich, P. R., J. L. Hamrick, P. Chavarriaga & G. Kochert. 1998. Microsatellite analysis of demographic genetic structure in fragmented populations of the tropical tree Symphonia globulifera. Molecular Ecology 7: 933-944. [ Links ]
Barreneche, T., C. Bodenes, C. Lexer, J. -F. Trontin, S. Fluch, R. Streiff, C. Plomion, G. Roussel, H. Steinkellner, K. Burg, J. -M. Favre, J. Glossl & A. Kremer. 1998. A genetic linkage map of Quercus robur L. (pedunculate oak) based on RAPD, SCAR, microsatellite, minisatellite, isozyme and 5S rDNA markers. Theoretical and Applied Genetics 97: 1090-1103. [ Links ]
Benbouza, H., J. M. Jacquemin, J. P. Baudoin & G. Mergeai. 2006. Optimization of a reliable, fast, cheap and sensitive silver staining method to detect SSR markers in polyacrylamide gels. Biotechnologie, Agronomie, Société et Environnement 10 (2): 77-81. [ Links ]
Boshier, D. H. 1995. Incompatibility in Cordia alliodora (Boraginaceae), a neotropical tree. Canadian Journal of Botany 73: 445-456. [ Links ]
Boshier, D. H., M. R. Chase & M. R. Bawa. 1995a. Population genetics of Cordia alliodora (Boraginaceae), a neotropical tree. 2. Mating system. American Journal of Botany 82: 476-483. [ Links ]
Boshier, D. H., M. R. Chase & M. R. Bawa. 1995b. Population genetics of Cordia alliodora (Boraginaceae), a neotropical tree. Gene flow, neighborhood, and population substructure. American Journal of Botany 82 (4):484-490. [ Links ]
Boshier, D. H. & M. Henson. 1997. Genetic variation in Cordia alliodora: genetics and tree improvement, pp. 39-65. En: Boshier, D. H. & A. T. Lamb (eds). Tropical Forestry Papers No. 36. Oxford Forestry Institute, Oxford. [ Links ]
Brondani, R. P. V., C. Brondani, R. Tarchini & D. Grattapaglia. 1998. Development and mapping of microsatellite based markers in Eucalyptus. Theoretical and Applied Genetics 97: 816-829. [ Links ]
Brondani, R. P. V. & D. Grattapaglia. 2002. Towards the construction of a genus wide reference linkage map for Eucalyptus based on microsatellite markers. Molecular and General Genetics 267: 338-347. [ Links ]
Buso, G. S. C., Z. P. S. Amaral, R. P. V. Brondani & M. E. Ferreira. 2006. Microsatellite markers for the common bean Phaseolus vulgaris. Molecular Ecology Notes 6 (1): 252-254. [ Links ]
Butcher, P. A., Glaubitz, J. C. & Moran, G. F. 1999. Applications for microsatellite markers in the domestication and conservation of forest trees. Forest Genetic Resources 27:34-42. [ Links ]
Carson, M., C. Walter & S. Carson. 2004. The Future of Forest Biotechnology, pp. 13-40. En: A Challenge Document for Presentation and Discussion at the Workshop Biotecnología Forestal. Global biotechnology forum-march 2-5, Concepción. [ Links ]
Cenicafé -Federación Nacional de Cafeteros de Colombia-. 2000. Ensayo de procedencias y progenies para dos especies forestales tropicales de alto valor comercial (marzo de 1996-febrero del 2000). Federacafé - Min. Ambiente. 73p. [ Links ]
Chase, M., R. Kesseli & K. Bawa. 1996. Microsatellite markers for population and conservation genetics of tropical trees. American Journal of Botany 83: 51-57. [ Links ]
Collevatti, R. G., R. P. Brondani & D. Grattapaglia. 1999. Development and characterization of microsatellite markers for genetic analysis of a Brazilian endangered tree species of Caryocar brasiliense. Heredity 83:748-756. [ Links ]
Daynandan, S., K. S. Bawa & R. Kesseli. 1997. Conservation of microsatellites among tropical tree (Leguminosae). American Journal of Botany 84:1658-1663. [ Links ]
Devey, M. E. , M. M. Sewell, T. L. Uren & D. B. Neale. 1999. Comparative mapping in loblolly and radiata pine using RFLP and microsatellite markers. Theoretical Applied Genetics 99:656-662. [ Links ]
Doyle, J. J. & J. L. Doyle. 1990. Isolation of plant DNA from fresh tissue. Focus 12: 13-15. [ Links ]
Echt, C. S. & C. D. Nelson. 1997. Linkage mapping and genome length in eastern white pine (Pinus strobus L. ). Theoretical and Applied Genetics 94: 1031-1037. [ Links ]
Gartland, K. M. A., R. C. Kellison & T. M. Fenning. 2002. Biotechnology and Europe's Forests of the future. A challenge document for presentation and discussion at Forest Biotechnology Forum in Europe: Impending Barriers, Policy, and Implications. Edinburgh, UK. 22 p. [ Links ]
Grattapaglia, D. 2003. Genolyptus pp. : 51-72 En: Borém, A., M. Giudice & T. Sediyama (eds. ). Melhoramento Genômico. Editora da UFV. Viçosa [ Links ]
Grattapaglia, D. & M. Kirst. 2008. Eucalyptus applied genomics: from gene sequences to breeding tools. New Phytologist 179: 911-929. [ Links ]
Hamrick, J. L., Y. B. Linhart & Mitton, J. B. 1979. Relationships between life history characteristics and electrophoretically detectable genetic variation in plants. Annual Review of Ecology, Evolution, and Systematics. 10: 173-200. [ Links ]
Isagi, Y. & S. Suhandono. 1997. PCR primers amplifying microsatellite loci of Quercus myrsinifolia. Blume and their conservation between oak species. Molecular Ecology 6: 897-899. [ Links ]
Jones, E. S., N. L. Mahoney, M. D. Hayword, I. P. Armstead, J. G. Jones & M. O. Humphreys, 2002. An enhanced molecular marker based genetic map of perennial ryegrass (Loliumperenne) reveals comparative relationships with other Poaceae genomes. Genome 45: 282-295. [ Links ]
Lefort, F., M. Lally, D. Thompson & G. C. Douglas. 1998. Morphological traits, microsatellite fingerprinting and genetic relatedness of a stand of elite oaks (Q. robur L. ) at Tullynally, Ireland. Silvae Genetica 47: 257-262. [ Links ]
Lexer, C., B. Heinze, H. Steinkellner, S. Kampfer, B. Ziegenhagen & J. Glosssl. 1999. Microsatellite analysis of maternal half-sib families of Quercus robur, pedunculate oak: detection of seed contaminations and inference of the seed parents form the offspring. Theoretical and Applied Genetics 99: 185-191. [ Links ]
Márquez, M. P. 2003. Caracterización molecular y morfológica de progenies de árboles plus seleccionadas dentro del "Ensayo de procedencias y progenies de Cordia alliodora" de CENICAFE - Colombia. Tesis Magíster Scientiae. CATIE. Turrialba. 80 p. [ Links ]
Marulanda, M. L., M. P. Márquez, R. J. Hernández. 2002. Selección y Caracterización de germoplasma de Cordia alliodora mediante marcadores moleculares RAPD, en Colombia, Sur América. Memorias VII Congreso Interamericano sobre el Medio Ambiente y VI Seminario Internacional del Medio Ambiente y desarrollo sostenible. Cartagena. 623 p. [ Links ]
Miller, A. J. & B. A. Schaal. 2006. Domestication and the distribution of genetic variation in wild and cultivated populations of the Mesoamerican fruit tree, Spondias purpurea L. (Anacardiaceae). Molecular Ecology 15: 1467-1480. [ Links ]
Nei, M. 1972. Genetic distance between populations. American Naturalist 106: 283-392. [ Links ]
O'Malley, D. M., D. Grattapaglia, J. X. Chaparro, P. L. Wilcox , H. V. Amerson, B. H. Liu, R. Whetten, S. McKeand, E. G. Kulhman, S. McCord, B. Crane & R. Sederoff. 1996. Molecular markers, forest genetics and tree breeding. Genomes of plants and animals: 21st Stadler Genetics Symposium. New York. 325 p. [ Links ]
Ospina, C., R. Hernández, S. Yandar, F. Aristizabal, E. Rincón, Z. Gil, J. García & N. Paternina 2008. El nogal cafetero (Cordia alliodora). Guías silviculturales para el manejo de especies forestales con miras a la producción de madera en la zona andina colombiana. Edited by CENICAFÉ. Manizales. 43 p. [ Links ]
Peakall, R. & P. Smouse. 2006. GENALEX 6: Genetic analysis in Excel. Population genetics software for teaching and research. Molecular Ecology notes 6: 288-295. [ Links ]
Phillips-Mora, W., H. Rodríguez & P. J. Fritz, 1995. Marcadores de ADN: Teoría, Aplicaciones y Protocolos de trabajo. Unidad de Biotecnología Centro Agronómico Tropical de Investigación y Enseñanza CATIE. Turrialba. 183 p. [ Links ]
Rohlf, J. 1989. NTSYS-Pc: Numerical Taxonomy and Multivariate Analysis System. Exeter publisher. New York. 250 p. [ Links ]
Sebbenn, A. M., P. Y. Kageyama & A. C. Zanatto. 2002. Estimativas de ganhos genéticos na seleção em populações de Cariniana legalis (Mart. ) O. Ktze, incorporando informações do sistema misto de reprodução. Revista do Instituto Florestal 14: 65-77. [ Links ]
Sebbenn, A. M., A. C. Zanatto, M. L. Freitas, A. S. Sato & L. C. Ettori. 2005. Genetic variation in Araucaria cunninghamii provenances in Luiz Antonio- SP, Brazil. Crop Breeding and Applied Biotechnology 4: 1-8. [ Links ]
Sebbenn, A. M., D. H. Boshier, M. L. M. Freitas, A. C. S. Zanatto, A. S. Sato & L. C. Ettori, E. Moraes. 2007. Results of an International Provenance Trial of Cordia alliodora in São Paulo, Brazil at Five and 23 Years of Age. Silvae Genetica 56: 3-4. [ Links ]
Slatkin, M. 1995. A measure of population subdivision based on microsatellite allele frequencies. Genetics 139: 457-462. [ Links ]
Smith, O. S. & J. S. C. Smith. 1992. Measurement of genetic diversity a comparison of isozymic, RFLP, pedigree, and heterosis data. Maydica 37: 53-60. [ Links ]
Steinkellner, H., S. Fluch, E. Turetschek, C. Lexer, R. Streiff, A. Kremer, K. Burg & J. Glossl. 1997. Identification and characterization of (GA/CT)n -microsatellite loci from Quercus petraea. Plant Molecular Biology 33: 1093-1096. [ Links ]
Tessier, C., J. David, P. This, J. M. Boursiquot & A. Charrier. 1999. Optimization of the choice of molecular markers for varietal identification of Vitis vinifera L. Theoretical and Applied Genetics 98 (1): 171-177. [ Links ]
Ujino, T., T. Kawahara, Y. Tsumura, T. Nagamitsu, H. Yoshimaru & W. Ratnam. 1998. Development and polymorphism of simple sequence repeat DNA markers for Shorea curtisii and other Dipterocarpaceae species. Heredity 81: 422-428. [ Links ]
White, G. & W. Powell. 1997a. Isolation and characterization of microsatellite loci in Swietenia humilis (Meliaceae): an endangered tropical hardwood species. Molecular Ecology 6: 851-860. [ Links ]
White, G. & W. Powell. 1997b. Cross-species amplification of SSR loci in the Meliaceae family. Molecular Ecology 6: 1195-1197. [ Links ]














