Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Agronómica
Print version ISSN 0120-2812
Acta Agron. vol.60 no.2 Palmira Apr./June 2011
1Facultad de Ciencias Agropecuarias Universidad Nacional de Colombia sede Palmira, A.A. 237, Palmira, Valle del Cauca, Colombia. 2Departamento de Biología Universidad del Valle, A.A. 25360, Santiago de Cali, Valle del Cauca, Colombia. 3Fundación Centro para la Investigación en Sistemas Sostenibles de Producción Agropecuaria (CIPAV), Carrera 25 No 6-62 Cali, Valle del Cauca, Colombia. *Autor para correspondencia: ampossot@unal.edu.co, ampossot@gmail.com
Con el objeto de estudiar la diversidad genética del árbol forrajero nacedero Trichanthera gigantea (Humb. Bonpl.) Nees, existente en la colección nacional de la Fundación Centro para la Investigación en Sistemas Sostenibles de Producción Agropecuaria (CIPAV) se utilizaron cinco cebadores RAMs en 38 accesiones procedentes de once departamentos de Colombia y dos estados de Venezuela. Se obtuvieron 71 bandas usadas para estimar los parámetros de diversidad genética. Se adaptó un protocolo de extracción de ADN para la especie y se estableció un banco de ADN de las accesiones en la Universidad Nacional de Colombia sede Palmira, para la realización de trabajos futuros. Los resultados indicaron que la colección nacional de nacedero presenta heterogeneidad en sus accesiones, alta diversidad genética y no se encontró relación entre los grupos genéticos formados mediante el análisis molecular y las zonas geográficas de origen.
Palabra clave: ADN, bancos de germoplasma, extracción de ADN, marcadores genéticos, nacedero, Trichanthera gigantea.
To study the genetic diversity of the national collection of Trichanthera gigantea (Humb. & Bonpl.) Nees from the Foundation Center for Research in Sustainable Systems for Agricultural Production (CIPAV), five primers RAMs were used on 38 accessions from 11 departments of Colombia and two states of Venezuela. A pattern of 71 bands was obtained and used to estimate genetic diversity parameters. A specific protocol for DNA isolation was adapted and a DNA bank was established at the National University of Colombia, Palmira campus for further investigations. The results showed heterogeneity and high genetic diversity in these accessions. No relationship was found among molecular group analysis and the geographical origin zones.
Key words: DNA, DNA isolation, genetic markers, germplasm banks, nacedero, Trichanthera gigantea.
Durante los últimos años el desarrollo e implementación de alternativas económicas de alimentación animal tropical ha avanzado en el estudio y valoración de los recursos forrajeros locales, adaptados al medio y aceptados por los productores. Los árboles y arbustos multipropósito tienen un elevado potencial natural pero, paradójicamente, no han sido suficientemente investigados, pese a la necesidad de encontrar nuevas fuentes proteicas y de minerales para la alimentación animal. La utilización de plantas arbóreas y arbustivas forrajeras se presenta como una alternativa para el sector agropecuario en Colombia, sin embargo, en el país se desconocen características de importancia económica de recursos como el nacedero (Trichanthera gigantea), lo que limita su empleo como especie forrajera arbórea con potencial de uso dentro del enfoque de la agricultura sostenible en bancos de proteína o sistemas silvopastoriles. El nacedero ofrece algunas ventajas para su utilización como fuente de forraje por su adaptación a diversos agroecosistemas, un alto contenido de proteína, palatabilidad, digestibilidad y buena aceptación por los diferentes animales domésticos como bovinos, ovinos, porcinos y aves de producción familiar campesina (Leterme y Buldgen, 2007; Murgueitio, 1990).
Científicos y algunas organizaciones internacionales han realizado esfuerzos por investigar y difundir los beneficios de esta especie en América y Asia tropical (Ríos, 2002; Nguyen y Phan, 1995). Este auge ha contribuido a ampliar el conocimiento sobre la oferta nutricional en diferentes especies animales, principalmente en monogástricos, donde se empezó a observar una alta variabilidad en las evaluaciones con animales (Ospina, 2000). Con el fin de establecer si la variación fenotípica observada es fundamentalmente de origen genético, ambiental o producto de la interacción genotipo x ambiente, la Fundación Centro para la Investigación en Sistemas Sostenibles de Producción Agropecuaria (CIPAV) estableció en Colombia un banco de germoplasma con accesiones de Colombia y Venezuela (Ríos, 2002). Esta colección es, hasta la fecha, la más completa en términos de número de procedencias incluidas (Jiménez, 2006) y está registrada oficialmente ante el Instituto Humboldt, responsable del registro de colecciones relacionadas con la biodiversidad en Colombia.
En los trabajos de investigación realizados por el CIPAV se ha identificado el nacedero como una de las especies más promisorias como fuente de alimento para animales, tanto en monogástricos (conejos, curíes, gallinas y cerdos) como en rumiantes (ovinos y bovinos) (Ospina, 2002; Gómez et al., 1995). Esta planta es un árbol de la familia Acanthacea originario de los Andes del Norte, con distribución original en Ecuador, Colombia y Venezuela (Gentry, 1993) a la cual se le han registrado numerosos usos en las culturas popular indígena y campesina como atributos medicinales, ambientales (atracción del agua en los manantiales), mágico-religiosos o forrajeros (Ríos, 1994).
Estudios de caracterización molecular con utilización de patrones isoenzimáticos confirmaron la existencia de una alta variabilidad genética dentro de la colección en Cipav (Ríos, 1994). Esta variabilidad puede manifestarse en diferencias en la composición química de las accesiones (Ospina, 2000) y existen evidencias que señalan la presencia de diferencias en la calidad nutritiva de tipo intraespecífico, en los tenores de proteína, fibras, minerales y metabolitos secundarios en T. gigantea que invocan la necesidad de profundizar en la investigación genética de diferentes accesiones del árbol (Rosales, 1997; Leterme y Muñoz, 2007). Sin embargo, se requieren estudios más precisos para la evaluación de la diversidad de la colección utilizando marcadores de ADN. Por otra parte, el diagnóstico reciente sobre el estado de biodiversidad en Colombia indica que varios de los ecosistemas de montaña se encuentran seriamente amenazados con alto riesgo de pérdida de los recursos genéticos, razón por la cual se hace necesario caracterizar y evaluar los materiales recolectados para su posterior utilización en programas de conservación y mejoramiento (Romero et al., 2008).
En este trabajo se estudió la diversidad genética de la colección nacional de nacedero establecida por el CIPAV, usando la técnica molecular RAMs (Random Amplified Microsatellites), con el propósito de utilizar estos resultados en el futuro desarrollo de estrategias de conservación de germoplasma, selección de materiales superiores y programas de mejoramiento genético.
Se recolectaron hojas y brotes jóvenes de 41 accesiones procedentes de ocho departamentos de Colombia y tres estados de Venezuela, de la colección nacional de nacedero establecida por el CIPAV localizada al suroccidente de Cali, Valle del Cauca, Colombia a 1.042 m.s.n.m, con una precipitación promedio de 1.887 mm y 25 °C de temperatura. Las muestras se conservaron en nitrógeno líquido para su transporte y hasta el momento de su procesamiento en el Laboratorio de Biología Molecular de la Universidad Nacional de Colombia sede Palmira, donde se realizaron todos los análisis de laboratorio y estadísticos de resultados. Para la extracción de ADN se evaluaron las metodologías: (1) la descrita por Dellaporta et al. (1983) con algunas modificaciones y el kit comercial Plant DNEasy Mini Kit de Qiagen ® utilizando las recomendaciones del fabricante. (2) La evaluación de la calidad y cantidad del ADN obtenido se realizó mediante geles de agarosa al 0.8% corridos en tampón TBE 0.5X (Tris-borato 0045M; EDTA 0.001M) a 100 voltios durante una hora y teñidos con bromuro de etidio a una concentración final de 0.5 ug/ml. Las concentraciones de ADN se determinaron mediante comparación con concentraciones conocidas de ADN del bacteriófago Lambda.
Para el establecimiento de un banco de ADN, las muestras extraídas con sus identificaciones respectivas fueron almacenadas en cajas criogénicas a -80 °C en la Universidad Nacional de Colombia sede Palmira. Se conformó una base de datos automatizada donde se asignó un código único y se registró toda la información disponible para cada una de las accesiones. Para visualizar la distribución de las accesiones de la colección con la información geográfica disponible, las accesiones se ubicaron espacialmente en un mapa utilizando el programa DIVA-GIS versión 7.7.7.2.
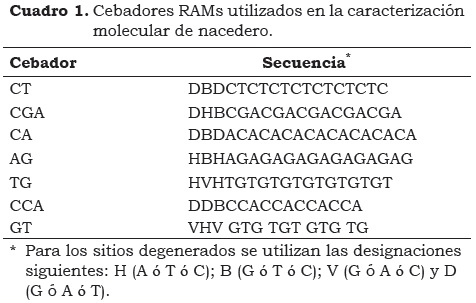
Para evaluar la diversidad genética se emplearon los siete cebadores RAMs (Cuadro 1) con las condiciones de amplificación por PCR descritas por Bonilla et al. (2008) para uchuva (Physallis peruviana). Los productos amplificados se visualizaron en geles de poliacrilamida al 6% (37:1 -acrilamida:bisacrilamida) corridos a 160 voltios por 1 hora y teñidos con sales de plata como se describe en protocolos estándar (Sambrook et al., 1989). La información de los patrones de bandas obtenidos se registró en una matriz binaria de presencia (1) o, ausencia (0). Para la selección de bandas polimórficas se consideró como locus polimórfico aquel en el cual la frecuencia del alelo más común fue < 95%. Los análisis estadísticos fueron realizados a partir de esta matriz con el uso de los programas Simqual del paquete NTSYS- PC (Numerical Taxonomy System for Personal Computer) y el programa TFPGA (Tools for Population Genetic Analisys).
La similitud genética se calculó mediante el coeficiente de Dice (Nei, 1978): S= 2a/(2a + b + c), donde a son las bandas compartidas por ambos individuos, b son las bandas presentes en el individuo (1), pero no en (2), y c son las bandas presentes en el individuo (2), pero no en (1). El análisis de agrupamiento se realizó con el programa SAHN de NTSYS -PC (versión 2.02g, 1998) utilizando el método UPGM A, un método gráfico de agrupamiento por parejas que usa el promedio aritmético no ponderado. El dendrograma se construyó con el programa TREE de NTSYS -PC (versión 2.02g, 1998). Se hizo un análisis de correspondencia múltiple (ACM) para asociar columnas y filas de la matriz binaria determinando el nivel de asociación o determinar proximidad (Joseph et al., 1992). Para estimar la diversidad genética se utilizaron los parámetros de heterocigocidad promedio esperada (He) y el porcentaje de loci polimórficos (P), los cuales se estimaron sobre todos los loci y el promedio de los mismos, de acuerdo con la fórmula no sesgada de Nei (1973).
Extracción y establecimiento del banco de ADN
Mediante la metodología de Dellaporta et al. (1983) se obtuvieron concentraciones de ADN entre 30 y 300 ng/ul, de buena calidad para la amplificación por PCR de los RAMs (Foto 1). Se hicieron algunas modificaciones adicionales a la metodología consistentes en la creación de una precipitación adicional con fenol: cloroformo: alcohol isoamílico en proporción 24:24:1 y la dilución final del ADN en 100 ul de tampón TE 1X, lo que aseguró una buena concentración y la eliminación de otras sustancias que pudieran interferir en las amplificaciones. La extracción mediante el kit comercial Plant DNEasy Mini Kit de Qiagen ® permitió la extracción de ADN en concentraciones entre 30 y 50 ng/ul en un volumen de dilución final de 50 ul, una modificación efectuada a las condiciones sugeridas por el fabricante con el fin de obtener una mayor concentración de ADN. De igual manera, el ADN obtenido presentó la cantidad y calidad suficientes para la realización de las amplificaciones por PCR de los RAMs.
Las metodologías empleadas en esta investigación para la extracción de ADN han permitido obtener ADN de buena calidad y de concentración adecuada en especies vegetales como Physalis peruviana (Bonilla et al., 2008), Lycopersicum sculentum (Restrepo, 2003), Phaseolus vulgaris (Henríquez, 2000) y Manihot esculenta (Mejía, 2002). No obstante, con base en los resultados de esta investigación se sugiere la utilización de la metodología modificada de Dellaporta et al. (1983) en nacedero, en donde el 70.7% de las muestras presentó una concentración > 50 ng/ul, ya que en tiempos similares es posible obtener una mayor cantidad y concentración de ADN de buena calidad a un menor costo (datos no publicados) y utilizando insumos básicos de laboratorio que no son empleados por la otra metodología.
Los resultados permitieron establecer un banco de ADN con 41 muestras extraídas, correspondientes a las accesiones de la colección del CIPAV, el cual se encuentra disponible en el Laboratorio de Biología Molecular de la Universidad Nacional de Colombia sede Palmira, a una temperatura de -20 °C, acompañado de una base de datos automatizada en la cual se encuentran registrados los datos de pasaporte disponibles por accesión y las respectivas concentraciones de ADN por muestra. Mediante la utilización de programas de sistemas de información geográfica se realizó un mapa para visualizar la distribución de las accesiones y garantizar el acceso rápido a la información de la base de datos.
Características de los cebadores
Para la evaluación de la diversidad genética de la colección se seleccionaron cinco de los siete cebadores RAMs utilizados por presentar algún grado de polimorfismo. El cebador AG no amplificó para la mayoría de las muestras, posiblemente por las condiciones no apropiadas de PCR para este cebador específico, mientras que el cebador GT no fue incluido en el análisis por presentar un patrón uniforme de pocas bandas para todas las accesiones, sin posibilidad de discriminación entre ellas. Los cinco cebadores utilizados generaron un patrón de 71 bandas con pesos moleculares entre 1500 y 120 pb y el número de bandas por cebador estuvo entre 11 para CGA y 18 para CT. En trabajos sobre diversidad genética de plantas (Marulanda y Márquez, 2001) seleccionaron 12 de 71 cebadores RAPDs y obtuvieron 45 bandas totales, de las cuales 38 fueron polimórficas con pesos moleculares entre 805 y 1501 pb. En otros estudios con especies silvestres de fresa (Rubus idaeus) mediante la misma técnica se obtuvieron 62 bandas, de las cuales 47 fueron polimórficas (Graham y McNicol, 1995). En uchuva, con la técnica RAMs se obtuvo un patrón de 50 bandas con siete cebadores para la evaluación de la diversidad genética de 43 accesiones del Banco de Germoplasma de la Universidad Nacional de Colombia sede Palmira (Bonilla et al., 2008). En mora (Rubus sp.) se obtuvo un patrón de 58 bandas al utilizar seis cebadores RAMs en la evaluación de la diversidad genética de 38 accesiones (Morillo et al., 2005). En guayaba (Psidium sp.) se obtuvieron 72 bandas con cinco cebadores RAMs para la evaluación de la diversidad genética de árboles nativos (Sanabria et al., 2006). Al comparar el número de loci RAMs generados por los cinco cebadores de este estudio (71 en total) con los propuestos de manera general para estudios moleculares en plantas (Nybom y Bartish, 2000), se concluye que el número de loci para estimar parámetros genéticos con el fin de evaluar la diversidad de la colección nacional de nacedero fue adecuado y confiable para el análisis estadístico.
Los valores de heterocigocidad esperada (He) calculados mediante el programa TFPGA considerando todas las accesiones del banco como un solo grupo y para cada uno de los cebadores, mostraron que el valor más alto fue para el cebador CT (0.31) y el más bajo para el cebador CCA (0.13). El valor de porcentaje de loci polimórficos más alto se encontró con el cebador TG (81.25%) y el más bajo con el cebador CCA (30.77%). Posiblemente el alto polimorfismo hallado con el cebador TG (81.25%) y heterocigocidad esperada (0.25) se debe a una mayor frecuencia entre los sitios de hibridación y las cadenas de ADN complementarias de esa secuencia, lo que evidencia la presencia de las regiones microsatélites TG y CT. El valor alto de polimorfismo (77.78% y 81.25%) y de He (0.31 y 0.25) encontrados con los cebadores CT y TG respectivamente, permiten concluir que estos cebadores son los que más aportan a la diversidad y resultan apropiados para su utilización en futuras investigaciones sobre la evaluación de la diversidad genética y estructura poblacional de accesiones de nacedero. Sin embargo, los cebadores CGA y CA mostraron valores importantes de polimorfismo (54.54% y 53.85% y He 0.23 y 0.20, respectivamente). El cebador CCA mostró la He y polimorfismo más bajos (0.13 y 30.77%) por lo cual se considera que tiene el menor aporte a la diversidad. De manera general, se observó correlación entre He y polimorfismo en los cebadores evaluados.
Análisis descriptivo
El análisis del dendrograma realizado con el coeficiente de Dice (Nei, 1978) y mediante el método de clasificación UPGMA (Figura 1) mostró que no existe una relación significativa entre los agrupamientos formados y los departamentos de procedencia de las accesiones. A un nivel aproximado de similitud de 0.8 se formaron dos grupos (A y B). El grupo A con las accesiones procedentes principalmente de Venezuela y los departamentos colombianos de Antioquia, Quindío, Boyacá y Valle del Cauca. El grupo B, conformado principalmente por las accesiones provenientes de Nariño, Meta, Tolima y Guaviare. Las accesiones 12 y 46 provenientes de Antioquia no formaron un grupo definido y, por el contrario, se ubicaron de manera más cercana a accesiones provenientes de Venezuela. Las accesiones 33, 49 y 50, originarias del Quindío, se ubicaron de manera dispersa en el grupo A y están relacionadas con accesiones de Venezuela, Valle del Cauca y Santander, respectivamente. Las accesiones 39 - 47, provenientes de Cundinamarca, y 42 - 16, naturales de Santander, se ubicaron en grupos diferentes (grupo B y A, respectivamente) mucho más relacionadas con accesiones de Venezuela. El ACM (Figura 2) separó las introducciones en dos grupos: A y B, de acuerdo con el dendrograma, a una distancia de 0.8. Esto confirma que no existe una relación entre los agrupamientos y los departamentos o estados de procedencia de las accesiones, además, muestra la heterogeneidad de las accesiones dentro de la colección de nacedero.
Los resultados de esta investigación están de acuerdo con lo encontrado en caracterizaciones previas de la colección de nacedero al utilizar marcadores isoenzimáticos, donde se formaron ocho grupos electroforéticos diferentes. El grupo principal incluyó accesiones procedentes tanto de Colombia como de Venezuela, mientras que los siete grupos restantes incluyeron sólo accesiones venezolanas (Ríos, 1994). En un análisis cualitativo con empleo de 25 descriptores morfológicos para la caracterización de algunas accesiones de la colección de nacedero, se formaron cuatro grupos sin ninguna relación entre sitios geográficos de procedencia y grupos formados. Del mismo modo, mediante un análisis de variables cuantitativas relacionadas con la producción y calidad del forraje, se formaron cinco grupos sin relación con los departamentos de procedencia (Ospina, 2000), no obstante, uno de estos grupos correspondiente a las accesiones procedentes del Valle del Cauca se agrupó en este estudio a un nivel de similitud 0.8 (Figura 1).
La ausencia de agrupamientos relacionados con el departamento de procedencia a partir del cálculo de semejanza mediante el uso de marcadores moleculares RAMs realizado en este estudio y los resultados de la caracterización morfo-agronómica previa (Ospina, 2000), indican heterogeneidad en las accesiones de la colección nacional de nacedero establecida por el CIPAV. La mayoría de los grupos formados mediante los análisis de trabajos anteriores no tuvieron relación con los grupos formados a nivel molecular, posiblemente debido a que los caracteres morfológicos y agronómicos tenidos en cuenta para la evaluación inicial del banco: (1) son susceptibles de modificar por factores ambientales, (2) no reflejan la variabilidad presente en la colección, (3) no fueron suficientes para una evaluación en detalle (Moreno et al., 2004).
La técnica molecular empleada en este estudio mostró un alto grado de polimorfismo y sensibilidad para la discriminación de las accesiones de la colección, lo que sugiere que los marcadores moleculares RAMs son útiles para la evaluación de la diversidad genética de colecciones, bancos de germoplasma y poblaciones naturales; además la técnica ha sido reportada para la caracterización molecular de especies vegetales como soya (Glycine max) (Akkaya et al., 1992) y frutales como uchuva (P. peruviana) (Bonilla et al., 2008), mora (Rubus sp.) (Morillo et al., 2006) y guayaba (Psidium sp.) (Sanabria et al., 2006). Entre las ventajas se encuentran bajo costo, reproducibilidad, utilización de un solo cebador, no requiere información previa, alto polimorfismo y fácil implementación (Muñoz et al., 2008).
Diversidad genética
Teniendo en cuenta la distribución geográfica de las accesiones se realizó una división de la colección en cuatro grupos, de acuerdo con la zona geográfica de procedencia (Cuadro 2). La heterocigocidad esperada para la colección total fue de 0.23, lo que revela un alto grado de diversidad en la colección. Se escogió la heterocigocidad de Nei (1978), ya que ésta presenta una corrección por el número de muestras el cual en este estudio es bajo (3 a 14 individuos por grupo) (Morillo et al., 2005). El valor encontrado para la colección de nacedero es alto (0 < He < 0.3) y parece que no existen organismos bisexuales con valores He > 0.31 (Nei, 1978; Selander 1979, citado por Caujapé-Castells, 2006). Aunque no hay estudios de diversidad genética usando marcadores moleculares en nacedero para realizar comparaciones, en investigaciones de la colección nacional de guayaba (Psidium sp.), utilizando marcadores RAPD y donde se incluyeron accesiones procedentes de diferentes continentes, se encontró un valor de He = 0.20, el cual es considerado alto (Rueda et al., 2006). No obstante, en un estudio con árboles nativos del Valle del Cauca de la misma especie se halló una He de 0.44 usando marcadores moleculares RAMs (Sanabria et al., 2006). En accesiones de mora (Rubus sp.) (Morillo et al., 2005) y uchuva (Physallis peruviana) (Bonilla et al., 2008) del Banco de Germoplasma de la Universidad Nacional de Colombia sede Palmira, utilizando RAMs se encontraron valores de He de 0.31 y 0.25, respectivamente, que se consideran altos comparados con resultados obtenidos en cultivares comerciales de tomate (Lycopersicon sculentum) que presentaron una He de 0.18 (Restrepo, 2003).
El porcentaje de loci polimórficos hallado para la colección fue de 61.97%, lo que indica que la colección incluye una buena diversidad y heterogeneidad genéticas. Ambos grupos 2 y 3 presentaron los valores más altos de He (0.22), seguidos por los grupos 1 y 4 (0.15). El porcentaje de loci polimórfico más alto se observó en el grupo 2 (57.74%) seguido por los grupos 3 (54.93%), 4 (40.84%) y 1 (30.98%). Los valores cercanos de heterocigocidad esperada para los grupos formados hace presumir que las accesiones en cada uno de estos proviene de una población histórica inicial y corrobora la propuesta de que la colección es una muestra representativa de una única población panmíctica.
Los valores bajos de He y polimorfismo en el grupo 1 pueden ser explicados por el bajo número de accesiones procedentes de esa zona particular. Por lo anterior, existe la necesidad de incluir en la colección más accesiones procedentes de los departamentos de Nariño, Putumayo y Cauca que representen de manera más amplia la diversidad de nacedero existente de esa región. El grupo 4, con accesiones procedentes de Venezuela, presentó uno de los valores más bajos de He (0.15). Sin embargo, el porcentaje de loci polimórfico fue 40.84%, lo que sugiere una menor diversidad de las accesiones venezolanas, en comparación con las colombianas de los grupos 2 y 3. El grupo 2, con accesiones de los departamentos de Valle del Cauca, Quindío, Antioquia, Tolima y Caldas mostró los valores más altos de heterocigocidad esperada y polimorfismo (0.22 y 57.74%) lo que indica que es el grupo con la mayor diversidad genética de la colección. Lo anterior se explica debido a que estas accesiones son procedentes de zonas con alto uso agrícola y frecuente utilización del nacedero en sistemas de producción agroforestales (Murgueitio, 1990), lo que hace presumir una alta variabilidad de las accesiones de esta zona. No obstante, el grupo 3 con accesiones provenientes de zonas de ladera con alto uso agrícola mostró valores muy cercanos al grupo 2, lo que hace presumir que la mayor diversidad genética de la colección nacional de nacedero establecida por el CIPAV está representada por las accesiones colombianas de la zona central del país.
Diferenciación de las accesiones
Utilizando la subdivisión de los grupos usada para la estimación de la heterocigocidad esperada se realizó la estimación del índice de fijación (Fst) para verificar la existencia de diferenciación genética de las accesiones de acuerdo con cada uno de los grupos formados. El valor de Fst hallado en este estudio fue de 0.15 (D.S. = 0.02, 1000 replicaciones) lo que indica que existe una baja diferenciación genética de los grupos (Wright, 1978). En nacedero, no obstante la escasa producción de semilla sexual causada por problemas en la polinización (McDade, 1983), es posible que el flujo genético se haya dado principalmente por transporte de aquellas, ya que se han reportado murciélagos (Glossophaga saricina), diferentes especies de aves, abejas (Eulaema sp.), hormigas y lepidópteros de la familia Sphingidae que se alimentan de flores y semillas de este árbol, especialmente en horas de la tarde (Ríos, 1993). En estudios de diversidad genética en colecciones y bancos de germoplasma de guayaba (Rueda et al., 2006), cacao (Theobroma sp.) (Moreno et al., 2004) y uchuva (Bonilla et al., 2008) no se incluyen estadísticos de estructura poblacional por considerar que las accesiones de los bancos no corresponden a una muestra representativa de poblaciones naturales y, en muchas ocasiones, una región geográfica se encuentra representada por solo una o dos accesiones, además, porque el agrupamiento de las accesiones recolectadas no se relaciona con la distribución geográfica y en consecuencia no se considera pertinente calcular los estadísticos de diferenciación poblacional.
En la colección de nacedero del CIPAV, varios de los departamentos de procedencia de las accesiones se encuentran representados únicamente por un par de accesiones. Aunque no existen valores de Fst reportados para esta planta que permitan hacer comparaciones, el valor encontrado confirma que las accesiones proceden históricamente de una única población panmíctica y que la colección es una muestra representativa de la misma. Los resultados sugieren la necesidad de emprender trabajos orientados a recolectar nuevas accesiones que representen de una manera más amplia la diversidad que puede existir en esta especie, especialmente en los departamentos colombianos de Nariño, Cauca y Meta, donde se recolectaron pocas accesiones; y Putumayo, Caquetá, Huila y Chocó donde no se han realizado recolecciones. Los trabajos futuros deben ampliar las características biogeográficas de cada accesión ya que la denominación actual por departamentos (Colombia) o estados (Venezuela) es de tipo político y administrativo y no representa diferencias o similitudes por ecosistemas o agroecosistemas.
Distancia genética
A pesar que los valores de distancia genética calculados con el índice de Nei (1978) no fueron muy altos (entre 0.03 y 0.18) comparados con otros estudios de diversidad genética en bancos de germoplasma (Bonilla et al., 2008; Sanabria et al., 2006; Morillo et al., 2005; Rueda et al., 2006), la mayor distancia genética se encontró en los grupos 1 y 4 (0.18), resultado que puede ser explicado por la relativa alta distancia geográfica entre las accesiones de estos grupos (Nariño-Cauca y Venezuela, respectivamente) y la más baja para los grupos 2 y 3 (0,03), lo que puede ser explicado por la cercanía geográfica entre las accesiones de ambos grupos (región Andina colombiana) que facilita el intercambio o flujo genético entre departamentos. El dendrograma para los cuatro grupos construido mediante el método UPGM A (Figura 3) y con un 'bootstrapp' de 1000 repeticiones, mostró un alto porcentaje de loci que soportan la formación de los tres nodos (61.97, 56.34 y 100, respectivamente) lo que indica una alta confiabilidad de los loci utilizados en el análisis de diversidad genética de la colección y la efectividad de la técnica molecular RAMs para el estudio realizado.
La ausencia de agrupamientos relacionados con el departamento de procedencia a partir del análisis molecular realizado en este estudio y los resultados de caracterizaciones morfológicas previas, indican heterogeneidad de las accesiones de la colección nacional de nacedero establecida por el CIPAV. No obstante los valores bajos de heterocigosidad esperada y polimorfismo obtenidos en el grupo 1 sugieren que existe la necesidad de incluir en la colección más accesiones procedentes de los departamentos de Nariño, Putumayo y Cauca, que representen de manera más amplia la diversidad existente en esa región.
Este trabajo estableció las condiciones de laboratorio necesarias para la realización de estudios de diversidad genética en nacedero; además, el análisis mediante marcadores moleculares RAMs permitió detectar la variabilidad genética de las accesiones de la colección y mostró un alto grado de polimorfismo y sensibilidad para su discriminación. Por lo anterior esta técnica puede ser considerada como una herramienta útil para evaluar la diversidad genética de colecciones, bancos de germoplasma y poblaciones naturales.
Al Grupo de Investigación en Diversidad Biológica de la Universidad Nacional de Colombia sede Palmira, por la financiación de este trabajo.
Akkaya, M. S.; Bhagwat, A. A.; y Gregan, P. B. 1992. Lenght polymorphisms of simple sequence repeat DNA in soybean. Genetics 132:1131 - 1139. [ Links ]
Bonilla, M. L.; Espinosa, K.; Posso, A. M.; Vasquez, H.; y Muñoz, J. E. 2008. Caracterización molecular de 43 accesiones de uchuva de seis departamentos de Colombia. Acta Agron. 57(2):109 - 115. [ Links ]
Caujapé-Castells, J. 2006. Brújula para botánicos desorientados en la genética de poblaciones. Exegen Ediciones. Las Palmas de Gran Canaria. España. 132 p. [ Links ]
Dellaporta, S. L.; Wood, J.; y Hicks, J. B. 1983. A plant DNA minipreparation: Versión II. Plan. Molec. Biol. Rep. 14:19 - 21. [ Links ]
Gentry, A. 1993. A Field Guide to the families and genera of woody plants of Northwestern South America (Colombia, Ecuador, Perú) -with supplementary notes on herbaceous taxa. Conservation International, Washington, D.C. [ Links ]
Gómez, M. E.; Rodríguez, L.; Murgueitio, E.; Ríos, C. I.; Molina, C. H.; Molina, E.; y Molina. J. P. 1995. árboles y arbustos forrajeros utilizados en la alimentación animal como fuente proteica: matarratón (Gliricidia sepium), nacedero (Trichanthera gigantea), pízamo (Erythrina fusca) y botón de oro (Tithonia diversifolia). Centro para la Investigación en Sistemas Sostenibles de Producción Agropecuaria (CIPAV), Cali, Colombia. 129 p. [ Links ]
Graham, J. y McNicol, R. J. 1995. An examination of the ability of RAPD markers to determine the relationships within and between Rubus species. Theor. Appl. Genet. 90(7 - 8):1128 - 1132. [ Links ]
Jiménez, M. A. 2006. Producción de biomasa de Nacedero (Trichanthera gigantea) en diferentes escenarios de sombra y frecuencias de cortes, en el Rancho Ebenezer. Niquinohomo, Masaya. Tesis de grado Ingeniero Agrónomo. Managua Nicaragua. Universidad Nacional Agraria, Facultad de Ciencia Animal. [ Links ]
Joseph, H.; Anderson, R.; Tatham, R.; y Black, W. 1992. Multivariate data analysis with readings. 3rd Ed. Riverside. 554 p. [ Links ]
Leterme, P. y Buldgen, A. 2007. Nutritional value of tree leaves in pigs. En: Leterme, P.; Buldgen, A.; Murgueitio, E.; y Cuartas, C. (eds.). Fooder banks for sustainable pig production systems. Centro para la Investigación en Sistemas Sostenibles de Producción Agropecuaria (CIPAV), Cali. p. 35 - 66. [ Links ]
Leterme, P. y Muñoz, C. 2007. Inter- and intraspecific genetic variability among forage trees use in agroforestry systems. En: Leterme, P.; Buldgen, A.; Murgueitio, E.; y Cuartas, C. (eds.). Fooder banks for sustainable pig production systems. Centro para la Investigación en Sistemas Sostenibles de Producción Agropecuaria (CIPAV), Cali. p. 123 - 138. [ Links ]
Marulanda, M. L. y Márquez, M. 2001. Caracterización de la diversidad Genética de Rubus glaucus Benth con marcadores moleculares (RAPD). Actual. Biol. 23(74):57 - 63. [ Links ]
McDade, L. 1983. Pollination intensity and seed set in Trichanthera gigantea (Acanthaceae). Biotropica 15(2):122 - 124. [ Links ]
Mejia, J. F. 2002. Caracterización molecular y patogénica de aislamientos de Sphaceloma manihoticola provenientes de la región Centro-Sur de Brasil. Trabajo de grado Agronomía.. Universidad Nacional de Colombia, sede Palmira. 126 p. [ Links ]
Moreno, Y.; Melgarejo, L. M.; Hernández, M. S.; Quintero, L.; y Vargas, G. 2004. Caracterización molecular de un banco de germoplasma del género Theobroma mediante la técnica RAPD. Rev. Col. Biot. 4 (2):15 - 24. [ Links ]
Morillo, Y.; Morillo, A. C.; Muñoz, J. E.; Vásquez, H. D.; y Zamorano, A. 2005. Caracterización molecular con microsatélites aleatorios RAMs de la colección de mora, Rubus spp., de la Universidad Nacional de Colombia sede Palmira. Acta Agron. 54 (2):51 - 60. [ Links ]
Muñoz, J. E.; Morillo, A. C.; y Morillo, Y. 2008. Microsatélites amplificados al azar (RAMs) en estudios de diversidad genética vegetal. Acta Agron. 57(4):219 - 26. [ Links ]
Murgueitio, E. 1990. Los árboles forrajeros como fuente de proteína. Convenio Interinstitucional para la Producción Agropecuaria del Valle de río Cauca. Centro para la Investigación en Sistemas Sostenibles de Producción Agropecuaria (CIPAV). Cali, Colombia. [ Links ]
Nei, M. 1973. Analysis of genetic diversity in subdivided populations. Proc. Nat. Acad. Sci. 70:3321 - 3326. [ Links ]
Nei, M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics 89:583 - 590. [ Links ]
Nguyen, N. H. y Phan, T., P. 1995. Vegetative propagation capacities and effect of fertilization on biomass production of Trichanthera gigantea. Livest. Res. Rural Develop. 7(1):1 - 4. [ Links ]
Nybom, M. y Bartish, I. 2000. Effects of life history traits and sampling strategies on genetic diversity estimates obtained with RAPD markers in plants. In: perspectives in plant ecology, evolution and systematic. Washington, D.C. 3:93 - 114. [ Links ]
Ospina, S. D. 2000. Caracterización de la variación genotípica en la composición química y digestibilidad de Trichanthera gigantea. Tesis de grado Zootecnia. Universidad Nacional de Colombia sede Palmira, Facultad de Ciencias Agropecuarias. 129 p. [ Links ]
Ospina, S. D. 2002. Establecimiento de la colección nacional de procedencias de Trichanthera gigantea (H & B) Nees en siete predios institucionales y/o campesinos del suroccidente colombiano. En: Tres especies vegetales promisorias: nacedero, botón de oro y bore. Centro para la Investigación en Sistemas Sostenibles de Producción Agropecuaria (CIPAV), Cali. p. 115 - 127. [ Links ]
Restrepo, E. 2003. Estudio de la diversidad del tomate cultivado tipo chonto Lycopersicum esculentum en las zonas productoras de Colombia. Trabajo de Maestría en Recursos Fitogenéticos Neotropicales. Universidad Nacional de Colombia, Sede Palmira. [ Links ]
Ríos, C. I. 1994. Apuntes etnobotánicos y aportes al conocimiento del nacedero Trichanthera gigantea (H & B) Nees. Tesis, Maestría en Desarrollo Sostenible de Sistemas Agrarios. Universidad Javeriana - CIPAV - IMCA. 71 p. [ Links ]
Ríos, C. I. 2002. Usos, manejo y producción de nacedero Trichanthera gigantea (H & B) Nees. En: Tres especies vegetales promisorias: nacedero, botón de oro y bore. Centro para la Investigación en Sistemas Sostenibles de Producción Agropecuaria (CIPAV). Cali. p. 97 - 114. [ Links ]
Romero, M.; Cabrera, E.; y Ortiz, N. 2008. Informe sobre el estado de la biodiversidad en Colombia 2006-2007. Instituto de Investigación Alexander von Humboldt. Bogotá, D.C. Colombia. 186 p. [ Links ]
Rosales, M. 1997. Trichanthera gigantea (Humboldt & Bonpland) Nees: A review. Livestock Research for Rural Development 9 (4) Art 7. www.cipav.org.co/lrrd/lrrd9/4/mauro942.htm [ Links ]
Rueda, E.; Muñoz, J. E.; Saavedra, R.; Palacio, J. D.; y Bravo, E. 2006. Caracterización molecular del banco de germoplasma de guayaba Psidium spp. del centro de investigación Corpoica-Palmira. Fitotécnia Colombiana (6):2 - 9. [ Links ]
Sambrook, J.; Fritsch, E. F; y Maniatis, T. 1989. Molecular cloning. A laboratory manual. Second edition. Cold Spring Harbor Laboratory Press. [ Links ]
Sanabria, H.; García, M.; Díaz, H.; y Muñoz, J. E. 2006. Caracterización molecular con marcadores RAMs de árboles nativos de Psidium guajava (guayaba) en el Valle del Cauca. Acta Agron. 55 (1):23 - 30. [ Links ]
Wright, S. 1978. Evolution and the genetics of populations, variability within and among natural populations, Vol 4. University of Chicago Press. Chicago. [ Links ]














