Introducción
La uchuva (Physalis peruviana L.) es uno de los cultivos de mayor crecimiento en la zona andina de Colombia, ya que ha pasado de 1170 ha en 2015 a 1713 ha en 2019. Se estima que este cultivo representa anualmente cerca de 35.6 millones de dólares en divisas para el país (Agronet, 2020; Procolombia, 2020). Los principales productores de esta fruta son Boyacá (40 %), Cundinamarca (31 %) y Antioquia (10 %), y es este último el que presenta los mayores rendimientos con 24.1 t/ha, muy superiores a los alcanzados en las otras regiones (11.1-14.5 t/ha) (Agronet, 2020). Los principales problemas fitosanitarios de la uchuva incluyen el marchitamiento vascular (Fusarium oxysporum), la muerte descendente (Phoma sp.) y la marchitez bacterial (Ralstonia solanacearum) (Fischer et al., 2014). Sin embargo, en los últimos años se han registrado de manera creciente diferentes enfermedades de origen viral causadas por un complejo de virus que puede incluir al tobacco mosaic virus (TMV) (Gómez et al., 1997), potato virus X (PVX) (Zapata et al., 2005; Gutiérrez et al., 2015a), potato virus Y (PVY) (Aguirre et al., 2014; Álvarez et al., 2018; Cuttler et al., 2018), potato virus V (PVV) (Álvarez et al., 2018), cape gooseberry ilarvirus 1 (CGIV-1) (Gallo et al., 2018), potato mop-top virus (PMTV), potato yellow vein virus (PYVV), potato yellowing virus (PYV), southern tomato virus (STV) y potato virus S (PVS) (Gallo et al., 2020). Además, recientemente, Corrales-Cabra et al. (2021) identificaron tres nuevas especies de virus en el oriente de Antioquia: el nepovirus Physalis vein necrosis virus (PhyVNV), el torradovirus Physalis torrado virus (PhyTV) y el potexvirus Physalis virus X (PhyVX). Estos nuevos hallazgos se suman a las cerca de 27 especies de virus reportados en uchuva en el mundo (Gallo et al., 2020; Kitajima, 2020; Savi et al., 2021).
La mayor parte de los registros de virus en Colombia se han realizado a partir de cultivos ubicados en el oriente de Antioquia (Gutiérrez et al., 2015a; Álvarez et al., 2018; Gallo et al., 2018; 2020; Corrales-Cabra et al., 2021), sin embargo, en este departamento hay otras zonas en el norte y suroeste donde se cultiva uchuva. Con el fin de generar una línea-base de riesgo fitosanitario viral para apoyar futuros planes de expansión de este cultivo en la subregión del suroeste Antioqueño, en esta investigación se evaluó, mediante RT-qPCR, la infección de ocho virus de RNA (CGIV-1, PhyVNV, PMTV, PVS, PVV, PVX, PVY y PYVV) en muestras foliares sintomáticas y asintomáticas de uchuva, y en semillas extraídas de frutos comercializados en la zona. Adicionalmente, en una muestra compuesta de los RNA obtenidos en las semillas sexuales, se evaluó la posible presencia de otros virus mediante NGS.
Materiales y métodos
Material vegetal
Entre marzo y agosto de 2021 se realizaron recorridos de reconocimiento de enfermedades virales en cultivos de uchuva de los municipios de Jardín (4 lotes) (5°35’54’’ N-75°49’11’’ O) y Urrao (4 lotes) (6°19’1 ’’ N-76°7’59’’ O) ubicados en el suroeste de Antioquia. En ambos municipios se detectaron plantas con síntomas asociados a enfermedades virales como: moteados, deformación foliar, clorosis intervenal, amarillamiento de nervaduras y clorosis general de la lámina foliar (Figura 1). En cada uno de los ocho lotes visitados, se colectaron dos muestras foliares compuestas (10x): una con hojas asintomáticas y otra con hojas sintomáticas. En esta misma subregión se obtuvieron ocho grupos de frutos de uchuva comercializados en mercados locales, de ellos se extrajeron aproximadamente 500 mg de semillas para inducir su germinación en cajas de Petri con papel absorbente humedecido.

Figura 1 Síntomas observados en tejido foliar de plantas de uchuva del suroeste de Antioquia. (A) Hoja asintomática. (B) Hoja con deformación (C) Clorosis intervenal. (D) Amarillamiento de nervaduras. (E) Moteado foliar y clorosis intervenal. (F) Clorosis generalizada de lámina foliar.
Extracción de ácidos nucleicos
En cada una de las muestras foliares se procedió a la extracción de RNA total con el método del Trizol (Chomczynski y Sacchi, 1987) (Thermo-Fisher Scientific, EE. UU.), a partir de la maceración de 100 mg de tejido con nitrógeno líquido. Para el caso de las semillas germinadas, se realizó la extracción del RNA utilizando el kit GeneJET Plant RNA Purification (Thermo-Fisher Scientific), según recomendaciones del fabricante. La concentración del RNA y su pureza fue evaluada mediante lecturas a 260 nm y 280 nm en un equipo Nanodrop 2000C (Thermo-Fisher Scientific).
Detección de virus por RT-qPCR
La síntesis de cDNA se realizó utilizando 200 U de la enzima RevertAid Reverse Transcriptase (Thermo-Fisher Scientific), 1X del buffer RT, 0.5 mM dNTPs, 20 U de inhibidor de RNAsas RiboLock, 100 ng de RNA, además de 100 pmoles de los primers: PYVV_R_CP, Ilar3CP_R, y 123-end, utilizados para la detección de los virus PYVV, CGIV-1 y PMTV, respectivamente; mientras que el primer Oligo-(dT)18 se usó para los otros virus (tabla 1). Las reacciones sin la enzima se ubicaron a 65° C por 5 min y posteriormente se incubaron a 42° C por 1 h; la enzima se desactivó a 70° C por 10 min. Posteriormente, la PCR en tiempo real se realizó en un volumen final de 14 μL conteniendo 6.25 μL del kit Maxima SYBR Green/ROX (Thermo-Fisher Scientific), 50-100 ng del cDNA y 0.3 μM de los primers específicos para cada virus (tabla 1), en un equipo Rotor-Gene Q-5plex (Qiagen, Alemania). El programa de amplificación consistió en 10 min a 95° C, seguido de 35 ciclos de 15 s a 95° C y 60 s a 52° C. Una vez finalizados los corridos se registraron los valores de Ct (threshold cycle) y de Tm (meltingtemperature) en el rango 50-99° C mediante la aplicación HRM del equipo (High Resolution Melting). En todos los corridos de qPCR se incluyeron muestras positivas y negativas para cada virus evaluado.
Secuenciación de nueva generación (NGS).
Ya que el cultivo de uchuva en Colombia se propaga mediante semilla sexual, en este estudio se complementaron las pruebas de RT-qPCR con la evaluación de virus presentes en dicho material mediante NGS del transcriptoma derivado de una muestra compuesta (bulk) del RNA procedente de los lotes de semilla. La cantidad y calidad del RNA fueron evaluadas con un equipo Bioanalyzer 2100 (Agilent Technologies, EE. UU.), y la librería de cDNA fue sintetizada con el kit TruSeq Stranded Total RNA LT Sample (Illumina, EE. UU.), previa remoción del RNA ribosomal (rRNA) con el kit Ribo-Zero Plant (Illumina). La secuenciación se realizó en la Plataforma NovaSeq de la compañía Macrogen (Corea del Sur). La identificación de reads asociados a genomas virales se realizó con el software Plant Virus Detection Pipeline (Gutiérrez et al., 2021). Los genomas de aquellos virus de RNA detectados en el transcriptoma, fueron posteriormente mapeados con Magic-BLAST (ncbi.github.io/magicblast/) y visualizados mediante el programa Integrative Genomics Viewer (https://software.broadinstitute.org/software/). Los ORF de los genomas virales fueron identificados con ayuda de ORFfinder (https://www.ncbi.nlm.nih.gov/orffinder) y BLASTX (Gish y States, 1993). Finalmente, se realizaron análisis filogenéticos de los scaffolds virales, a partir de alineamientos obtenidos con MUSCLE (Edgar, 2004). Los árboles filogenéticos fueron generados con el método de Neighbor-Joining (Saitou y Nei, 1987) con 1000 réplicas de bootstrap usando el programa MEGAX (Kumar et al., 2018).
Resultados y discusión
Detección de virus en cultivos de uchuva
Mediante pruebas de RT-qPCR se detectó la infección de cuatro (PMTV, PVS, PVX y PVY) y cinco (CGIV-1, PMTV, PVS, PVX y PVY) de los ocho virus de RNA evaluados en muestras de tejidos foliares sintomáticos y asintomáticos de uchuva, respectivamente. Los virus PMTV (75 %) y PVY (50 %) fueron los que presentaron mayor prevalencia en las muestras sintomáticas; mientras que PVX (62.5 %), PMTV (50 %) y PVY (50 %) lo fueron para las muestras asintomáticas. Los virus PhyVNV, PVV y PYVV no se detectaron en el estudio (Figura 2A). Estos resultados son muy similares a los encontrados en trabajos recientes realizados por Gallo (2020) y Corrales-Cabra et al. (2021) en cultivos de uchuva del oriente de Antioquia; en el primer caso se evaluaron cuatro virus de RNA (PVX, PVY, CGIV- 1 y PVV) en muestras sintomáticas, encontrándose los tres primeros en niveles de 56 %, 56 % y 17 %, respectivamente; mientras que el PVV no se detectó. Para el segundo caso, de siete virus evaluados (PVY, PVX, PVS, PVV, PYVV, CGIV-1 y PhyVNV), el PVX fue el más prevalente tanto en muestras asintomáticas (73.3 %) como sintomáticas (66.7 %), mientras que PVY, PVS y CGIV-1 y PhyVNV se detectaron con niveles del 13.3 % al 46.6 % (Corrales-Cabra et al., 2021) En dicho trabajo tampoco se encontraron diferencias en la detección de virus entre las muestras sintomáticas y asintomáticas, e incluso para algunos virus, la prevalencia de virus como PVX, CGIV-1 y PhyVNV fue mayor en estas últimas. Esta situación representa un riesgo para los programas de manejo integrado de enfermedades en este cultivo, por cuanto en ausencia de pruebas moleculares como la RT-qPCR, se dificulta la detección temprana de virus en los cultivos y se facilita su dispersión con el movimiento de material vegetal asintomático entre las regiones cultivadoras de este frutal.
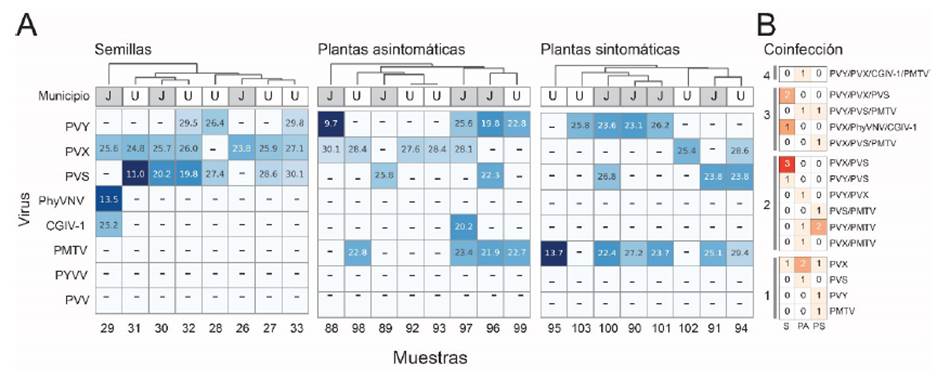
Figura 2 Prevalencia de virus de RNA en tejido foliar de plantas y en semillas de uchuva del suroeste de Antioquia. (A) Valores de Ct para cada virus evaluado en las ocho muestras de semillas, plantas asintomáticas y sintomáticas. La intensidad del color azul señala aquellas muestras con menor valor de Ct. (B) Análisis de coinfecciones en cada tipo de muestra evaluada. El símbolo - indica muestras negativas; J = Jardín; U: Urrao; S: semilla; PA: Plantas asintomáticas; PS: Plantas sintomáticas.
Por otra parte, es de especial interés la detección del pomovirus (Virgaviridae) PMTV, el cual presentó altos niveles de prevalencia en las muestras foliares de uchuva. Este virus es transmitido en papa por el plasmodiofórido del suelo Spongospora subterranea f.sp. subterranea y aunque en Colombia el PMTV se ha detectado en dicho hospedante en la región Andina (Gil et al., 2011, 2016), fue solo hasta 2020 cuando se registró su presencia en uchuva (Gallo et al., 2020). El hallazgo del PMTV en el suroeste de Antioquia, donde no se cultiva papa, plantea varios interrogantes sobre su forma de transmisión y los efectos sobre los rendimientos y longevidad de los cultivos de uchuva, siendo necesaria la realización de un estudio que evalúe la presencia de S. subterránea f.sp. subterranea en los suelos de esta zona y la posible presencia de agallas con quistosoros del patógeno en las raíces de las plantas.
Así mismo, la detección del ilarvirus (Bromoviridae) CGIV-1 en solo una de las muestras foliares evaluadas y la ausencia de detección en campo del nepovirus (.Secoviridae) PhyVNV plantean la necesidad de reforzar las medidas fitosanitarias que impidan la dispersión de estos virus en Colombia. Ambos virus fueron recientemente registrados por primera vez en el oriente de Antioquia, CGIV-1 en 2018 en Marinilla (Gallo et al., 2018) y PhyVNV en 2021 en La Unión y Marinilla (Corrales-Cabra et al., 2021); en este último trabajo, los niveles de prevalencia ya alcanzaban el 46.6 % (CGIV-1) y 20 % (PhyVNV) en muestras asintomáticas de campo y valores alarmantes del 53.3 % (CGIV-1) y 86.7 % (PhyVNV) en plántulas de uchuva. Para el establecimiento de diferentes cultivos en el país, es cada vez más frecuente la utilización de plántulas producidas y distribuidas por viveros. Por ello, la expansión de cultivos de uchuva en el suroeste de Antioquia y en otras regiones debería considerar la evaluación viral previa de este material de propagación.
Al evaluar los valores de Ct obtenidos para cada virus detectado, se presentó un rango de variación entre 9.7 y 30.1, que denota diferentes niveles de carga viral en las muestras, aunque fue evidente que PVY y PMTV fueron los que presentaron mayores títulos virales, con valores promedio de Ct para PVY de 19.5 y 24.7; y de 22.7 y 20.2 para PMTV, en tejidos asintomáticos y sintomáticos, respectivamente (Figura 2A). La manifestación de síntomas en tejidos vegetales infectados por virus no solo depende de la carga viral, sino también de diferentes factores que incluyen el nivel de resistencia o susceptibilidad del hospedante, las condiciones ambientales que rigen dicha interacción y el estado fenológico del cultivo, entre otros aspectos (Hull, 2013; Marín y Gutiérrez, 2016). El hecho de que un tejido presente un alto título viral favorece los procesos de dispersión de los virus, especialmente en aquellos casos en que existen poblaciones altas de vectores (Hull, 2013; Marín y Gutiérrez, 2016). En este sentido, las prácticas dirigidas a la disminución de dichas cargas virales (p. ej. podas fitosanitarias, eliminación de plantas sintomáticas, manejo de arvenses) y de las poblaciones de vectores, deben ser parte fundamental de los programas de manejo integrado de estas enfermedades. Con respecto a los valores de Tm obtenidos, estos se presentaron en los rangos de temperatura antes reportados por Gallo (2020) y Corrales-Cabra et al. (2021) para estos virus, siendo los valores promedio para el PVY de 79.1 (DE=0.5), PVX de 83.7 (DE=0.6), PVS de 81.7 (DE=0.5) y PMTV de 79.8 (DE=1.0).
Al evaluarse los niveles de coinfección en el material foliar, se encontró un promedio de 2 virus por muestra sintomática y 1.9 virus en las asintomáticas; la infección mixta por 4 virus (PVY/ PVX/CGIV-1/PMTV) fue la más alta encontrada, mientras que un solo virus se detectó en tres de las muestras sintomáticas y asintomáticas (Figura 2B).
Detección de virus en semillas de uchuva
Con respecto a la evaluación de virus en brotes derivados de semilla sexual, en este estudio se detectó, en al menos un lote, cinco de los ocho virus evaluados (PVY, PVX, PVS, PhyVNV y CGIV-1), siendo los más prevalentes PVX, PVS y PVY con valores de 87.5 %, 75 % y 37.5 %, respectivamente; mientras que PhyVNV y CGIV-1 solo se encontraron en una de las muestras (12.5 %). Los valores de Ct determinados en las pruebas de RT-qPCR para estos virus, se presentaron en rangos similares a los encontrados en las muestras foliares, con valores promedio de 28.6, 25.6 y 22.8 para PVY, PVX y PVS, respectivamente (Figura 2A).
La detección de PVX con 87.5 % de prevalencia en semilla sexual de uchuva coincide con el reporte reciente de Corrales-Cabra et al. (2021) sobre el hallazgo de este mismo virus en el 80 % de las muestras de semillas evaluadas en el oriente de Antioquia; estos autores también reportaron a PVS, PVY, PhyVNV y CGIV-1 en el material de siembra. El promedio de virus detectado por muestra de semilla sexual fue de 2.25 con un máximo de tres virus detectados en tres lotes y un mínimo de un virus registrado en un lote de semilla (Figura 2B). Estos valores son muy similares a los observados en las muestras foliares y soportan la hipótesis planteada inicialmente por Gallo (2020) y Corrales-Cabra et al. (2021), que indican que este material de siembra juega un papel importante en la transmisión de virus en uchuva. Sin embargo, dado que el número de muestras evaluadas fue relativamente pequeño y restringido a una subregión geográfica de Antioquia, será necesario realizar estudios con un volumen mayor de muestras y de cobertura geográfica, de manera que se determinen con mayor precisión los niveles de prevalencia de los virus encontrados en este estudio en los cultivos de uchuva del país.
En el presente estudio, los resultados obtenidos se complementaron con el análisis bioinformático del transcriptoma de un bulk de los RNA derivados de los lotes de semillas. En total se obtuvieron 20 836 536 reads de 101 nt, de los cuales 91 100 equivalentes al 0.44 %, correspondieron a secuencias asociadas con genomas virales (Figura 3A), siendo las secuencias de virus de DNA de los géneros Solendovirus (RPM=79.2). y Badnavirus (RPM=27.7) (Caulimoviridae), así como del mycovirus Pittosporum cryptic virus (RPM=130.1) las más numerosas (no mostrados). Ya que es posible que los caulimoviridos se encuentren integrados en el genoma del hospedante, es necesario validar en el futuro estos resultados mediante evaluaciones con metodologías como la amplificación por círculo rodante (RCA), la utilización de plataformas de NGS de tercera generación o la detección directa con pruebas serológicas (Geering et al., 2010; Sepúlveda et al., 2021).

Figura 3 Detección mediante NGS de virus en semilla sexual de uchuva. (A) Proporción de secuencias asociadas a genomas virales. (B) Virus de RNA identificados en el transcriptoma analizado mediante el programa PVDP. Los puntos indican los reads y su posición con respecto a cada genoma viral. La intensidad del color azul de los puntos es un indicativo de sus niveles de identidad con los genomas de referencia. (C) Árboles filogenéticos basados en regiones alineables de los scaffolds obtenidos con respecto a genomas de referencia de estas especies de virus. Los números sobre las ramas indican los valores de bootstrap
Mediante NGS se detectaron secuencias en niveles inferiores a 2 RPM de dos componentes genómicos (RNA 3 y RNA1) del cucumber mosaic virus (CMV), PVS, PhyVNV (RNA 1 y RNA2) y del tamarillo leaf malformation virus (TaLMV) (Figura 3B). Este último es un potyvirus asociado con la virosis que afecta el tomate de árbol en Antioquia, enfermedad considerada como uno de los principales problemas fitopatológicos de este cultivo y cuyo genoma completo fue secuenciado en muestras procedentes del oriente y norte de Antioquia (Gutiérrez et al., 2015b; Duque et al., 2017). Ya que existen reportes de primers útiles para la detección de TaLMV mediante RT-qPCR, se recomienda confirmar la presencia de este virus en cultivos de uchuva de otras regiones del país. La ausencia de reads asociados al genoma de PVX en la secuenciación NGS, puede deberse al bajo título viral que presentó dicho virus en el bulk de semillas sexuales preparado, por lo que se sugiere en el futuro enriquecer las muestras utilizadas para este tipo de análisis con el uso de metodologías como VANA y extracción de dsRNA.
Para tres de los virus de RNA identificados (CMV, PVS y TaLMV) fue posible la obtención de un scaffold del tamaño suficiente (PVS_RL5_BPP17: 800 nt, PVS_Antioquia_BPP17: 666 nt, TaLMV: 1354 nt, CMV: 1141 nt) para generar alineamientos con genomas de referencia disponibles en GenBank y obtener árboles filogenéticos que confirmaron la identidad taxonómica de dichos virus; el aislamiento de CMV se ubicó en un clado con secuencias de diferentes hospedantes (p. ej. Spinacia oleraceae y Cucumis melo) y asociado al subgrupo IA; mientras que el PVS se presentó asociado a dos clados: el del linaje PVSPhu reportado por Gutiérrez et al. (2013) y el correspondiente a la raza PVSA. Finalmente, el TaLMV se ubicó en un clado con aislamientos de este virus obtenidos en S. betaceum en Colombia y claramente diferenciado de otros potyvirus (Figura 3C).
Los resultados obtenidos demuestran que la semilla sexual de uchuva constituye una fuente de transmisión de virus entre regiones cultivadoras de este frutal y plantean la necesidad de establecer un programa de certificación de semilla en el país. Además, se sugiere utilizar pruebas moleculares altamente sensibles como la RT-qPCR y NGS, al menos durante las fases iniciales para obtención de plantas madre derivadas de cultivo in vitro.
Conclusiones
En evaluaciones de tejidos foliares de plantas sintomáticas y asintomáticas de uchuva obtenidas en ocho cultivos del suroeste de Antioquia (Colombia) se detectaron los virus de RNA: PVY, PVX, PVS, CGIV-1 y PMTV en al menos una de las muestras, siendo el PMTV (75 %) y el PVX (62.5 %) los virus que presentaron mayores niveles de prevalencia en muestras sintomáticas y asintomáticas, respectivamente.
Las evaluaciones de RT-qPCR realizadas en ocho lotes de semilla sexual de uchuva señalaron al PVX (87.5 %) y PVS (75 %) como los virus con mayores niveles de prevalencia en este material, aunque también se detectaron el PVY (37.5 %), PhyVNV (12.5 %) y CGIV-1 (12.5 %).
Mediante NGS se identificaron reads asociados a los genomas de CMV, PVS, PhyVNV y TaLMV, lo que plantea la necesidad de realizar pruebas biológicas que evalúen los mecanismos de transmisión, la sintomatología que inducen y las pérdidas económicas que causan dichos virus en los cultivos de uchuva de Colombia.















