Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO  Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.25 no.4 Bogotá Dec. 2005
Polimorfismos genéticos de aislamientos del género Malassezia obtenidos en Colombia de pacientes con lesión dermatológica y sin ella
Adriana M. Celis, Maria Caridad Cepero de García
CIMIC, Centro de Investigaciones Microbiológicas, Universidad de los Andes, Bogotá, D.C., Colombia.
Introducción. Las especies del género Malassezia se consideran levaduras oportunistas emergentes de gran importancia. Han sido asociadas a diferentes patologías dermatológicas y sistémicas de las cuales se aislan una o más especies de este género. El papel de estas levaduras en las enfermedades dermatológicas no se ha aclarado completamente, ya que la Malassezia spp. pertenece a la flora normal de la piel.
Objetivo. Buscar marcadores genéticos en los aislamientos de Malassezia spp. que permitan correlacionar las lesiones dermatológicas con las especies aisladas.
Materiales y métodos. Se obtuvieron 103 aislamientos de Malassezia spp. a partir de muestras de pacientes con pitiriasis versicolor, dermatitis seborreica, dermatitis seborreica en pacientes positivos para VIH, dermatitis atópica, y de individuos sanos. Para los controles se usaron ocho cepas del Centraalbureau voor Schimmelcultures (CBS), Holanda. El perfil genético se realizó utilizando la técnica de ADN polimórfico amplificado aleatorio (RAPD) con tres iniciadores (OPA2, OPA4, OPA13). Los datos obtenidos se analizaron con los programas Diversity Database y SYN-TAX-PC.
Resultados. Se observó heterogeneidad genética intraespecífica en Malassezia furfur, Malassezia globosa, Malassezia restricta, Malassezia slooffiae y Malassezia obtusa, mientras que Malassezia sympodialis mostró mayor homogeneidad.
Conclusión. No se determinó ningún patrón genético específico mediante la técnica de RAPD para las especies de Malassezia que se pudiera relacionar con la entidad dermatológica implicada.
Palabras clave: Malassezia, RAPD, dermatitis seborreica, VIH, pitiriasis versicolor, dermatitis atópica.
Genetic polymorphism of Malassezia spp. yeast isolates from individuals with and without dermatological lesions
Introduction. Malassezia spp. yeasts are opportunistic and newly emergent diseases. One or more species have been isolated in association with dermatological pathology and systemic disease. Their pathological role has not been fully elucidated since Malassezia spp. are common in normal skin flora.
Objective. In the current study, a search was undertaken for genetic markers in the Malassezia spp. isolates that correlate with each type of dermatologic lesion.
Materials and methods. A total of 103 strains of Malassezia spp were isolated from patients with pytiriasis versicolor, seborrheic dermatitis, seborrheic dermatitis HIV (+), and atopic dermatitis, as well as from healthy individuals. Eight isolates from CBS Centraalbureau voor Schimmelcultures (Netherlands) were used as controls. Fingerprinting was done using random amplification of polymorphic DNA technique (RAPD) with three primers (OPA2, OPA4, OPA13).The data were analyzed with Diversity Database and SYN-TAX-PC programs.
Results. Intraspecies genetic heterogeneity in M. furfur, M. globosa, M. restricta, M. slooffiae, M. obtusa was observed, whereas M. sympodialis showed the greatest homogeneity.
Conclusion. The dermatological disease caused by these different species was not associated with distinctive RAPD fingerprints.
Key words: Malassezia, RAPD, pytiriasis versicolor, seborrheic dermatitis, HIV, atopic dermatitis.
Las especies del género Malassezia se consideran levaduras oportunistas emergentes de gran importancia (1,2). Se caracterizan por ser lipofílicas y se encuentran como flora normal en la piel de humanos y mamíferos de sangre caliente; pueden actuar como patógenos cuando se exponen a ciertos cambios en el microclima de la piel y, por tal razón, se les asocia con diferentes patologías dermatológicas, sistémicas y presentes en pacientes inmunosuprimidos (3-8).
La taxonomía y nomenclatura del género Malassezia fue materia de controversia durante muchas décadas. Hasta 1990 sólo se reconocían tres especies: M. furfur (Robin) Baillon, M. sympodialis (Simmons y Guého), lípido dependientes, y M. pachydermatis (Weidman) C.W. Dodge, especie no lipofílica (9-11). Nuevos estudios realizados en 1995 con base en la morfología, la ultraestructura y la fisiología por medio de técnicas moleculares sirvieron para describir cuatro nuevas especies: M. globosa, M. restricta, M. obtusa y M. slooffiae; recientemente se describieron otras dos especies, M. dermatis y M. japonica (12-15).
La identificación de las diferentes especies se basa en su morfología celular y en sus aspectos fisiológicos, como la asimilación de detergentes no iónicos como única fuente lipídica (Tween 20, 40, 60 y 80), la presencia de catalasa, el crecimiento en cremophor EL y la actividad de
b- glucosidasa, entre otros (13,16-18). Varios métodos de tipificación molecular se han utilizado como herramientas epidemiológicas, confirmando la robustez de esta nueva estructura taxonómica (12). Así mismo, se han desarrollado estudios para la identificación rápida de especies del género Malassezia utilizando técnicas como PFGE, RAPD, PCR anidada, AFLPTm, PCR-RFLP y PCR-REA (19-26).En Colombia, nuestro grupo de investigación ha venido desarrollando estudios con el fin de determinar si la distribución de las especies de Malassezia está relacionada con la presencia de distintos tipos de lesiones dermatológicas; hemos encontrado que M. globosa es la especie predominante en pitiriasis versicolor y dermatitis seborreica en pacientes positivos para VIH, mientras que M. furfur y M. restricta aparecieron como las especies predominantes en dermatitis atópica y dermatitis seborreica, respectivamente (18). Sin embargo, debido a la dificultad que involucra el aislamiento en cultivo e identificación del género Malassezia por métodos morfológicos y fisiológicos, se hace necesaria la utilización de técnicas moleculares que permitan ampliar el conocimiento de la ecología y epidemiología de este género en nuestra población. Por tal razón, el objetivo de este trabajo fue la búsqueda de marcadores genéticos que permitieran correlacionar las lesiones dermatológicas de dermatitis atópica, pitiriasis versicolor, dermatitis seborreica y dermatitis seborreica en pacientes positivos para VIH con las especies de Malassezia relacionadas.
Materiales y métodos
En este estudio se incluyeron los aislamientos de Malassezia spp. obtenidos en un estudio anterior (18) a partir de 8 pacientes con dermatitis atópica, 12 pacientes con pitiriasis versicolor, 12 pacientes con dermatitis seborreica, 18 pacientes positivos para VIH con dermatitis seborreica y 14 individuos sin lesiones. Se incluyeron 103 aislamientos, 23 de M. furfur, 22 de M. globosa, 23 de M. sympodialis, 14 de M. restricta, 11 de Malassezia sp, 4 de M. slooffiae, 4 de M. obtusa y 2 de M. pachydermatis (18) ; el número de aislamientos es superior al de pacientes, ya que de cada paciente se tomaron, al menos, dos morfotipos de colonia (18,27,28). Como controles se emplearon 8 cepas de referencia M. furfur CBS 1878 NT, M. furfur CBS 7019 NT, M. sympodialis CBS 7222 T, M. globosa CBS 7966 T, M. restricta CBS 7877 T, M. slooffiae CBS 7956 T, M. obtusa CBS 7876 T, M. pachydermatis CBS 1879 NT, obtenidas de la CBS (Holanda) y dos cepas de referencia como grupo externo, Candida krusei ATCC 6255 y Candida parapsilosis ATCC 22019. Los aislamientos se identificaron con los métodos convencionales (13,16-18). La extracción de AND se realizó siguiendo el protocolo modificado de Saghai Marrof et al. (27,29,30). Posteriormente, se hizo la purificación del ADN con el juego de reactivos wizard TM DNA clean up SYSTEM # A7280 de Promega, de acuerdo con las especificaciones de la casa comercial, y se cuantificó por espectrofotometría (Beckman DN- 65), midiendo la absorbancia a 260 y 280 nm con el fin de conocer la concentración y la pureza del ADN.
Para el RAPD se utilizaron tres cebadores: OPA2 (TGCCGAGCTG), OPA 4 (AATCGGGCTG) y OPA13 (CAGCACCCAC) (20). El volumen de reacción fue de 25
ml y contenía 2,5 ml de tampón 10 X, 2,5 ml de MgCl2 25 mM, 2 ml de dNTPs 10 mM, 0,226 ml de cebador (0,1 mM) , 0,1 ml de taq polimerasa (TucanTaq, Corporgen) y 5 ml de AND (25 ng/ml). La amplificación se llevó a cabo en un termociclador PTC-100 MJ Research, Inc, con un programa que incluyó una desnaturalización inicial a 94°C de 4 minutos, seguida por 94°C (60 s), 34°C (60 s) y 72°C (2 min) durante 40 ciclos y una extensión final de 15 minutos a 72°C. La visualización de los productos de amplificación se hizo mediante una electroforesis convencional en geles de agarosa al 1,5 % en tampón TBE 0,5X con bromuro de etidio en una concentración de 0,5 mg/ml. Además, se corrió una muestra que no contenía ADN como control negativo, y se usó como patrón de peso molecular la escalera 1 kb plus DNA Invitrogen.El análisis de los datos se realizó independientemente para cada uno de los cebadores y para todos en conjunto mediante los programas Diversity Database de Bio-Rad y SYN-TAX-PC, con los cuales se trabajó el coeficiente de similitud de Jaccard y los agrupamientos por métodos jerárquicos utilizando UPGMA.
Resultados
Todos los aislamientos amplificaron de forma reproducible con los cebadores probados. Mediante la técnica de RAPD, se determinó que los aislamientos de M. furfur, M. globosa (
figuras 1A y 1B, respectivamente), M. restricta, M. slooffiae y M. obtusa (datos no mostrados) muestran una alta heterogeneidad genética intraespecífica.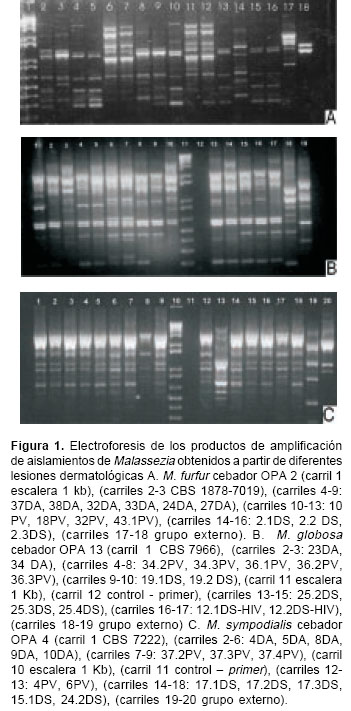
En los fenogramas obtenidos mediante SYN-TAXPC, en el caso de M. furfur, el aislamiento 9,1 DS-HIV fue el más alejado con 95% de disimilitud con respecto a los aislamientos restantes (
figura 2A). Los aislamientos provenientes de un mismo paciente, como 37 DA y 38 DA, 32 DA y 33 DA, 9,1 DS y 9,2 DS, mostraron bajos porcentajes de disimilitud entre 0,5 y 21%. Además, se observó una alta variabilidad en los aislamientos 2,2 DS y 2,3 DS, identificados mediante las pruebas convencionales como M. furfur, los cuales se agruparon con 0,4 % de disimilitud, mientras que el 2,1 DS se ubicó en un cluster diferente con alto porcentaje de disimilitud con respecto a los aislamientos antes mencionados, pese a provenir del mismo paciente (figura 2A). De igual forma ocurrió con M. globosa como se observa en la figura 2B en los aislamientos 36,1 PV, 36,2 PV y 36,3 PV. Los aislamientos de M. sympodialis mostraron los patrones más homogéneos con los cebadores probados (figura 1C); los aislamientos con mayor porcentaje de disimilitud en esta especie fueron 48 DS-HIV con un 61% y 6 PV con 88% (figura 2C). El análisis mediante el programa Diversity Database (datos no presentados) mostró datos similares, lo que permitió mayor confiabilidad de los resultados obtenidos.
No se determinó ningún patrón genético específico entre las especies de Malassezia relacionado con la entidad dermatológica implicada (pitiriasis versicolor, dermatitis seborreica, dermatitis seborreica en pacientes positivos para VIH, dermatitis atópica); por el contrario, como puede verse en la
figura 2A, para M. furfur los conglomerados claramente agruparon aislamientos de dermatitis atópica, dermatitis seborreica y pitiriasis versicolor; lo mismo puede observarse para M. sympodialis en la figura 2C; en este caso también se encuentran agrupadas estas entidades dermatológicas con aislamientos obtenidos a partir de individuos sin lesión dermatológica.Discusión
En años recientes los estudios del género Malassezia se han encaminado a la utilización de nuevas técnicas que permitan una mejor identificación de las especies de este género, con el fin de ampliar el conocimiento en cuanto a su papel patológico, ecológico y epidemiológico (13,16-26).
Varios autores han determinado la variabilidad genética intraespecífica dentro del género mediante la utilización de diversas técnicas como PFGE, PCR-RFLP, AFLP y análisis de secuencias (19-26), lo que concuerda con nuestros hallazgos, en los cuales los aislamientos de M. furfur, M. globosa, M. slooffiae, M. obtusa y M. pachydermatis mostraron una alta heterogeneidad genética mediante la técnica de RAPD.
Las especies del género Malassezia presentan cariotipos distintos que permiten diferenciarlas entre si; sin embargo, M. furfur presenta dos cariotipos diferentes que poseen una composición de G+C del 66,5% y el mismo valor de reasociación ADN/ADN >85% (20). Aunque esta variación permanece sin explicación, se ve que existe una relación con la alta variabilidad intraespecífica encontrada en esta especie. Estos cambios de patrones en el cariotipo pueden proveer variaciones a nivel genético y, a su vez, esto puede llevar a cambios en su fenotipo (31), como se evidenció en los aislamientos de esta especie obtenidos a partir de un mismo paciente, los cuales presentaron varios morfotipos de colonia (17,26,27). Los resultados de esta investigación indican la presencia de subpoblaciones entre las especies de Malassezia, lo que, como se reportó en un estudio anterior (20), impide la obtención de un patrón específico para cada entidad dermatológica. La posible presencia de subpoblaciones puede estar influenciada o no por ciertos factores exógenos, tales como las altas temperaturas, la alta humedad relativa y factores endógenos como piel grasa, hiperhidrosis, factores hereditarios, tratamiento con corticoesteroides e inmunodeficiencia (3-8). Se requiere adelantar otros estudios que permitan determinar si la variación genética dentro de estas especies está asociada con el cambio de saprobio a patógeno, lo que implicaría su participación en la aparición de lesiones dermatológicas tales como dermatitis seborreica y dermatitis atópica.
En el caso de la pitiriasis versicolor, cuyo agente etiológico es M. globosa según algunos autores (32), la ausencia de un patrón genético específico detectable mediante la técnica de RAPD y, por el contrario, la presencia de un alto polimorfismo podría verse asociadas con un aumento de su capacidad patogénica, lo que explicaría las variaciones en los grados de susceptibilidad in vitro a algunos medicamentos, según lo reportan Guého et al., quienes demostraron resistencia al fluconazol en M. globosa (33), y Rincón, que en un estudio reciente reportó concentraciones mínimas inhibitorias altas (CIM) frente a voriconazol, itraconazol y ketoconazol con respecto a las obtenidas para las restantes especies de Malassezia (34).
Por otra parte, en este trabajo los aislamientos estudiados de M. sympodialis mostraron homogeneidad en su patrón de amplificación, lo cual contrasta con lo observado por otros investigadores que reportan un alto polimorfismo, excepto para OPA 4, con el cual hay patrones de bandeo relativamente uniformes (20). De igual forma, mediante la PCR basada en consenso de secuencias intergénicas repetitivas enterobacterianas (ERIC-PCR), se determinaron diferencias genéticas entre los aislamientos analizados (27). Esta variación genética pudo deberse a diversos factores, como diferencias en la toma de la muestra, el sitio de la lesión, el medio de cultivo utilizado en su aislamiento, la distribución geográfica (diferentes grupos étnicos) y, además, a variaciones en la técnica de RAPD como tal (4-8).
Son diversos los factores que podrían estar involucrados en el fenómeno, entre ellos, sus características ecológicas, fisiológicas, inmunológicas y genéticas. Es necesario realizar nuevos estudios con nuevas técnicas moleculares que permitan dilucidar más el papel de estas especies en las diferentes entidades dermatológicas con las que se encuentran implicadas, ya que, como lo señalan Ashbee y Glyn, "hasta el momento son desconocidas las razones por las cuales algunos individuos desarrollan una enfermedad causada por levaduras del género Malassezia y, sin embargo, en otras personas éstas permanecen como flora normal" (35).
Agradecimientos
Los autores expresan su agradecimiento a Martha Emiliana Cárdenas por su invaluable colaboración en el análisis de los resultados y en la corrección del manuscrito.
Conflicto de intereses
Los autores expresan que no tienen ningún conflicto de intereses.
Financiación
Este estudio fue financiado por Colciencias, código: 1204-04-10175.
Correspondencia:
María Caridad Cepero de García. Centro de Investigaciones Microbiológicas, CIMIC. Departamento de Ciencias Biológicas. Universidad de los Andes. Cra 1a 18 A-10, Bogotá, D.C., Colombia.
Teléfono 3394949 Extensión 3762
Recibido: 24/09/04; aceptado: 16/08/05
Referencias
1. Guého E, Faergemann J, Lyman C, Anaissie E. Malassezia and Trichosporum: two emerging pathogenic basidiomycetous yeast-like fungi. J Med Vet Mycol 1994;32:367-78. [ Links ]
2. Perfect JR, Schell WA. The new fungal opportunists are coming. Clin Infect Dis 1996;22:112-8. [ Links ]
3. Marcon MJ, Powell DA. Human infections due to Malassezia spp. Clin Microbiol Rev 1992;5:101-19. [ Links ]
4. Aspiroz C, Ara M, Varea M, Rezusta A, Rubio C. Isolation of M. sympodialis from patients with pityriasis versicolor in Spain. Mycopathologia 2002;154:111-7. [ Links ]
5. Crespo V, Delgado V. Malassezia species in skin diseases. Curr Opin Infect Dis 2002;15:133-42. [ Links ]
6. Chryssanthou E, Broberger U, Petrini B. Malassezia pachydermatis fungaemia in a neonatal intensive care unit. Acta Paediatr 2001;90:323-7. [ Links ]
7. Nakabayashi A, Sei Y, Guillot J. Identification of Malassezia species isolated from patients with seborrhoeic dermatitis, atopic dermatitis, pityriasis versicolor and normal subjects. Med Mycol 2000;38: 337-41. [ Links ]
8. Gupta AK, Kohli Y, Faergemann J, Summerbell RC. Epidemiology of Malassezia yeast associated with pityriasis versicolor in Ontario, Canada. Med Mycol 2001;39:199-206. [ Links ]
9. Guého E, Meyer SA. A reevaluation of the genus Malassezia by means of genome comparison. Antonie van Leeuwenhoek 1989;55:245-51. [ Links ]
10. Simmons RB, Guého E. A new species of Malassezia. Mycol Res 1990;94:1146-9. [ Links ]
11. Guillot J, Guého E, Chermette R. Confirmation of the nomenclatural status of Malassezia pachydermatis. Antonie van Leeuwenhoek 1995;67:173-6. [ Links ]
12. Guillot J, Guého E. The diversity of Malassezia yeast confirmed by rRNA sequence and nuclear DNA comparisons. Antonie van Leeuwenhoek 1995;67:297-314. [ Links ]
13. Guého E, Midgley G, Guillot J. The genus Malassezia with description of four new species. Antonie van Leeuwenhoek 1996;69:337-55. [ Links ]
14. Sugita T, Takashima M, Shinoda T, Suto H, Unno T, Tsuboi R et al. New yeast species, Malassezia dermatis, isolated from patients with atopic dermatitis. J Clin Microbiol 2002;40:1363-7. [ Links ]
15. Sugita T Takashima M, Komada. Description of a new yeast species, Malassezia japonica, its detection in patients with atopic dermatitis and healthy subjets. J Clin Microbiol 2003;41:4695-9. [ Links ]
16. Guillot J, Guého E, Lesourd M, Midgley G, Chevrier G, Dupont B. Identification of Malassezia species. J Mycol Med 1996;6:103-10. [ Links ]
17. Mayser P, Haze P, Papavassilis C, Pickel M, Gruender K, Guého E. Differentiation of Malassezia species: selectivity of cremophor EL, castor oil and ricinoleic acid for M. furfur. Brit J Dermatol 1997;137:208- 13. [ Links ]
18. Rincón S, Celis A, Sopo L, Motta A, Cepero de García MC. Malassezia species isolated from patients with dermatologic lesions and normal subjects in Bogotá, Colombia. Biomédica 2005;25:189-95. [ Links ]
19. Boekhout T, Bosboom RW. Kariotyping of Malassezia yeast: Taxonomic and epidemiological implications. System Appl Microbiol 1994;17:146-53. [ Links ]
20. Boekhout T, Kamp M, Guého E. Molecular typing of Malassezia species with PFGE and RAPD. Med Mycol 1998;36:365-72. [ Links ]
21. Guillot J, Deville M, Berthelemy M, Provost F, Guého E. A single PCR-restriction endonuclease analysis for rapid identification of Malassezia species. Lett Appl Microbiol 2000;31:400-3. [ Links ]
22. Theelen B, Silvestri M, Guého E, Belkum AV, Boekhout T. Identification and typing of Malassezia yeast using amplified fragment length polymorphism (AFLPTm), random amplified polimorphic DNA (RAPD) and denaturing gradient gel electrophoresis (DGGE). FEMS Yeast Res 2001;1:79-86. [ Links ]
23. Gemmer CHM, DE Angelis YM, Telen B, Boekhout T, Dawson TL Jr. Fast, noninvasive method for molecular detection and differentiation of Malassezia yeast species on human skin and application of the method to Dandruff microbiology. J Clin Microbiol 2002; 9:3350-7. [ Links ]
24. Sugita T, Suto H, Unno T, Tsuboi R, Ogawa H, Shinoda T, et al. Molecular analysis of Malassezia microflora on the skin of atopic dermatitis patients and healthy subjects. J Clin Microbiol 2001;39:3486-90. [ Links ]
25. Gaitanis G, Velegraki A, Frangoulis E, Mitroussia A, Legakis NJ. Differentiation of Malassezia spp. PCR- RFLP analysis and subtyping of clinical isolates in Greece. Rev Iberoam Micol 2000;17:135. [ Links ]
26. Gupta AK, Kohli Y, Summerbell RC. Molecular differentiation of seven Malassezia species. J Clin Microbiol 2000;38:1869-75. [ Links ]
27. Cepero de García MC. Caracterización morfológica, fisiológica y genética de aislamientos humanos nativos de Malassezia spp. (tesis). Bogotá. Universidad de los Andes; 1999. [ Links ]
28. Celis A. Polimorfismos genéticos de aislamientos del género Malassezia obtenidos a partir de pacientes con lesión dermatológica e individuos sin lesión (tesis). Bogotá: Universidad de los Andes; 2003. [ Links ]
29. Weising K, Nybom H, Wolff K, Meyer W. DNA fingerprinting in plants and fungi. Boca Ratón; CRC Press Inc; 1995. p.61-75. [ Links ]
30. Velegraki A, Kambouris M, Kostourou, Chalevelaskis G, Legakis NJ. Rapid extraction of fungal DNA from clinical samples for PCR amplification. Med Mycol 1999;37:69-73. [ Links ]
31. Senczek D, Siesenop U, Bohm KH. Characterization of Malassezia species by means of phenotypic characteristics and detection of electrophoretic kariotypes by pulsed-field gel electrophoresis (PFGE). Mycoses 1999;42:409-14. [ Links ]
32. Crespo V, Martos A, Vera A, Crespo A, Sánchez F. Malassezia globosa as the causative agent of pityriasis versicolor. Brit J Dermatol 2000;143:799-803. [ Links ]
33. Guého E, Oster C, Improvisi L. Antifungal in vitro susceptibility of Malassezia yeasts. Proceedings of the Journées de Mycologie Médicale; 28-29 November 1997; Paris. Paris: Société Française de Mycologie Médiclae; 1997. p 6. [ Links ]
34. Rincón S. Determinación de la susceptibilidad de especies de Malassezia aisladas de individuos con y sin patología dermatológica frente a tres compuestos azoles (tesis). Bogotá: Universidad de los Andes; 2003. [ Links ]
35. Ashbee HR, Glyn E. Immunology of diseases associated with Malassezia species. Clin Microbiol Rev 2002; 15:21-57. [ Links ]














