Introducción
Para el año 2019 la Organización Mundial de la Salud (OMS) en el marco del seguimiento de los Objetivos de Desarrollo Sostenible (ODS), incluyó un nuevo indicador que permitiera el seguimiento de dos patógenos concretos como es Staphylococcus aureus resistente a la meticilina (SARM), y E. coli resistente a cefalosporinas de tercera generación, para esta bacteria la resistencia reportada ese mismo año fue de 36 %; así mismo en el año 2021, la OMS reporta el incremento de la resistencia bacteriana frente a antibióticos habitualmente empleados en tratamiento de infecciones causadas por estos patógenos, destacando la resistencia a la ciprofloxacina entre el 8.4 % y 92.9 % en Escherichia coli (E. coli) y entre 4.1 % y el 79.4 % para Klebsiella pneumoniae, esta última es un patógeno importante por su capacidad de mortalidad e Infecciones Asociadas a la Atención en Salud (lAAS)1.
Es por ello que la farmacorresistencia microbiana ha llevado a proponer diferentes líneas de contención de los patógenos Multi Drug Resistant (MDR), siendo una de ellas la realización de estudios que permitan la identificación microbiológica fenotípica y molecular de los diferentes mecanismos de resistencia expresados por los microorganismos de interés, vigilancia y control en el ámbito hospitalario2, estos estudios han permitido describir que la resistencia a los antibióticos y biocidas comparten algunas estrategias comunes al incorporar la participación de mecanismos adicionales a los genes de resistencia clásicos, esto involucra una serie de genes que codifican para bombas de expulsión de fármacos, anteriormente relacionadas con resistencia a biocidas, presentes en muchas bacterias MDR3.
El aumento de la aparición de bacterias resistentes a múltiples fármacos ha acelerado la necesidad de mejorar las terapias con antimicrobianos, el estudio de nuevos antibióticos y una comprensión más profunda del mecanismo de resistencia a los medicamentos; las bombas expulsión o también denominadas de "eflujo" han demostrado un papel relevante en la resistencia bacteriana intrínseca y extrínseca, esto gracias a la capacidad que le confiere a la bacteria para reducir la concentración del fármaco antimicrobiano dentro de la misma, así como la expulsión de estas sustancias al exterior; este mecanismo cuenta con una serie de proteínas de transporte presentes en la membrana y el periplasma bacteriano que permiten la eliminación de diferentes compuestos dentro de los cuáles se describe antimicrobianos, metales pesados tóxicos, detergentes, colorantes entre otros4, lo cual ha llevado inclusive a realizar estudios sobre los genes y proteínas involucradas en este mecanismo; en bacterias gram negativas se ha descrito la presencia de este mecanismo en E. coli, Pseudomonas aeuroginosa, Klebsiella pneumoniae, Neisseria gonorrhoeae entre otras, y cuyo ejercicio de la función va a depender de la energía, por lo que se han clasificado en dos grandes grupos dependiendo de su fuente de energía: bombas de eflujo primarias que dependen de la hidrólisis del ATP, y secundarias cuya extracción energética se deriva de gradientes químicos compuestos por protones o iones como el sodio5.
Recientemente se ha publicado que en bacterias gram negativas, la transformación por transferencia de plásmidos que codifican para la bomba AcrAB-TolC, están implicadas en mecanismos de resistencia a tetraciclinas6, por lo que se hace indispensable el desarrollo de técnicas y herramientas que permitan diferenciar actividades inhibidoras de eflujo multidrogas y desestabilizadoras de la membrana externa de inhibidores de las bombas de expulsión en las bacterias, facilitando la identificación de inhibidores de la bomba de eflujo (EPI) de molécula pequeña, capaces de restaurar la efectividad de los antibióticos disponibles7,8 la participación activa de los EPI conlleva a un impacto importante para la prevención de la resistencia a antbióticos a nivel mundial, pues su principal función se basa en la restricción de la actividad propia del sistema de bombas eflujo, con la disminución de la afinidad de unión del antibótico a los transportadores a través de modificaciones estructurales del fármaco, aumentando la permeabilidad celular que permite una elevada concentración del antibiótico en el interior de la célula, o impidiendo de la codificación de genes implicados con la expresión de este mecanismo4, es por esto que se han considerado como agentes terapéuticos potenciales que mejoran la actividad de los antibióticos a los cuáles ya se ha desarrollado resistencia por parte de la célula bacteriana5.
Aunque los EPI se encuentran el fase de experimentación desde inicios de este siglo5, diversos estudios han demostrado cómo inhibidores de las bombas de expulsión como el Phenylalanine-arginine ß-naphthylamide (PAßN)7 (primero en descubrirse en el año 2001), tienen capacidad de unión al sitio hidrofóbico ubicado dentro del AcrB4 inhibiendo así la expulsión del antibiótico; carbonyl cyanide 3-chlorophenylhydrazone (CCCP)3,9, EPI más conocido en los laboratorios, siendo un ionóforo que altera la fuerza motriz del protón en sus dos componentes Δψ y ΔpH así como al metabolismo bacteriano5, pequeños ARN Reguladores (sRNA)10, compuestos naturales como diadzein, lanatoside C, ácido protocatechuic, escopolamina, ácido gentísico11, ácido gálico12, y derivados alcaloides13, muestran efectos sinérgicos con diferentes antibióticos como la eritromicina, tetraciclina, rifampicina, fluoroquinolona, fosfomicina y levofloxacina3 al disminuir las concentraciones de antibióticos requeridas para inhibir el crecimiento bacteriano.
Este grupo de moléculas conocidas como EPI se han clasificado de acuerdo a su mecanismo de acción como disipadores de energía como el CCCP, e inhibidores por unión directa como el PAßN; otro grupo en donde se ha demostrado su potencial inhibidor pero de los cuáles se desconoce el mecanismo de acción, se ha denominado como EPI de otros orígenes o según su fuente incluyendo 3 subgrupos a mencionar: de origen sintético, originados de bibliotecas químicas diversificadas sintéticas o semisintéticas como los derivados de las quinolonas, compuestos peptidomiméticos, derivados de arilpiperazina y arilpiperazina y los derivados de piridopirimidina y piranopiridina; en grupo de los EPI de origen vegetal se contempla una gran diversidad de adyuvantes químicos que mejoran sinérgicamente la eficacia del fármaco hasta 30 veces, dentro de este subgrupo se incluyen alcaloides, flavonoides, polifenoles y diterpenos fenólicos; el último subgrupo se deriva de los microorganismos como el EA-3710 y EA-371d, cuya primera extracción se realizó a partir del extracto de fermentación de Streptomyces spp; y la P. aeruginosa que se ha reconocido como inhibidor específico de la bomba MexAB-OprM5.
Aunque los métodos microbiológicos convencionales son útiles para identificar especies y evaluar perfiles de resistencia, no son eficaces para trazar la diseminación de una población clonal dentro de un centro hospitalario o región, ni para determinar los mecanismos de resistencia presentes, así mismo, el uso clínico de antibiótico e inhibidor de bomba de expulsión (EPI) como estrategia de terapia combinada, va a depender de la permeabilidad de la membrana externa de la célula bacteriana4. de manera tal que se imposibilite el transporte activo y/o expulsión del fármaco ya que permite que el antibiótico permanezca en mayor concentración dentro de la célula5, y es aquí en dónde radica la importancia de estudiar los EPI en cepas circulantes en la región, pues al ser empleados como complemento en combinación con el tratamiento se podría mejorar la actividad del antibiótico contra bacterias que expresen este mecanismo de resistencia
Teniendo presente lo expuesto, y con el fin de ampliar la información sobre el comportamiento de la resistencia antimicrobiana en la región, el objetivo del presente estudio fue evaluar el efecto del inhibidor de bombas de eflujo Phenylalanine-arginine ß-naphthylamide sobre la resistencia bacteriana a los antibióticos ceftazidima, clortetraciclina, ciprofloxacina, ceftriaxona, ampicilina y gentamicina en cepas de bacilos gram negativos con presencia de múltiples genes BLEE aislados.
Materiales y métodos
Tipo de estudio:
Se realizó un estudio de tipo transversal, con diseño experimental para determinar el efecto el efecto del bloqueo de las bombas de eflujo con el EPI Phenylalanine-arginine ß-naphthylamide sobre la resistencia antimicrobiana en cepas de E. coli, y K. pneumoniae que presentaban genes de resistencia tipo BLEE.
Muestras:
Para el estudio se emplearon un total de 70 cepas de bacterias gram negativas a partir de 81 aislamientos bacterianos en una institución de tercer nivel de atención del departamento de Boyacá durante el año 2018, y los cuales presentaron genes de resistencia a los antibióticos, por lo cual, la caracterización fenotípica de las cepas permitió identificar fenotipos de resistencia con 35 cepas con betalactamasas de espectro extendido (BLEE), 25 cepas con BLEE tipo AmpC y 8 cepas con fenotipo de carbapenemasas; la caracterización ampliada fue reportada por nuestro grupo anteriormente14.
El primer paso fue la reactivación de las cepas en medio BHI, con crecimiento durante 24 horas, seguido a la reactivación se realizó la extracción de ADN empleando el kit de Promega "Wizard" Genomic DNA Purification siguiendo las instrucciones del fabricante, y una vez finalizada la extracción, se cuantificó el ADN con un micro-espectrofotómetro (NANO Maestrogen) con una relación de absorbancia 260/280 mayor o igual a 1,8; el ADN fue almacenado a -80 °C hasta su análisis.
Identificación molecular de los mecanismos de resistencia BLEE:
Todas las amplificaciones se realizaron mediante PCR convencional en un termociclador Labocon, utilizando el reactivo premezclado 2X PCR Taq MasterMix con dye de Applied Biological Materials (ABM) que contiene una concentración balanceada de ADN Taq polimerasa (50 unidades/ml), dNTPs (0.4 mM), MgCl2 (3 mM) y demás componentes necesarios para la PCR; las amplificaciones fueron realizadas en un volumen final de 25 uL (master master mix 1X; 0.2 uM de cada primer y 2 ng/uL de ADN) según los protocolos especificados para cada gen. Todos los iniciadores fueron sintetizados por Macrogen Korea.
Caracterización de genes blaTEM, blaCTMXi, blaSHVen cepas gram negativas: para la detección molecular de los genes de resistencia en Escherichia coli y Klebsiella pneumoniae se utilizó el protocolo 005 del Laboratorio de Epidemiología Molecular empleando iniciadores y condiciones establecidas por Paterson 200315; para la detección de los genes bla TEM (900 pb), se emplearon los iniciadores, F5'-AAACGCTGGTGAAAGTA-3' y R5'-AGCGATCTGTCTAT-3' y las siguientes condiciones: desnaturalización inicial de 94 °C 30 s seguido por 35 ciclos de 94 °C 30 s, alineamiento a 49 °C 1 min y extensión 72 °C 1 min con una extensión final de 72°C 10 min; para bla CTMX1 (500 pb), se utilizaron los iniciadores: F5'-GACGATGTCACTGGCTGAGC-3' y R5'-AGCCGCCGACGCTAATACA-3', amplificado bajo las condiciones: desnaturalización inicial de 94 °C 30s seguido por 35 ciclos de 94 °C 30 s, alineamiento a 58 °C 1 min y extensión 72 °C 1 min con una extensión final de 72 °C 10 min; para bla SHV (700pb), se emplearon los siguientes iniciadores, F5'ATGCGTTATATTCGCCTGTG-3' y R5'-TGCTTTGTTATTCGGGCCAA-3', amplificado bajo las condiciones: desnaturalización inicial de 94 °C 30s seguido por 35 ciclos de 94 °C 30s, alineamiento a 56 °C 1 min y extensión 72 °C 1 min con una extensión final de 72 °C 10 min.
Los productos de amplificación se evidenciaron mediante electroforesis en gel de agarosa al 1 % en TAE 1 %, empleando safeview classic (ABM) como agente intercalante, y opti DNA Marker 1Kb (ABM) como marcador de peso molecular, las bandas se evidenciaron en un transiluminador UltraSlim Led Illuminator Maestrogen.
Descripción Filogenética de los genes identificados:
Posterior a las pruebas moleculares, en todas las muestras en donde se evidenció amplificación de ADN, se realizó el protocolo de corte y purificación de banda a partir del gel de agarosa; para este procedimiento se utilizó el kit de promega para purificación de los productos de PCR; los productos de PCR purificados fueron enviados a la empresa Applied Biological Materials Inc en Canadá para la secuenciación; una vez recibido los análisis de secuenciación se procedió a la revisión de las cada una de las muestras enviadas empleando el software FinchTV 1.4.0 de Digital World Biology ® y posteriormente se realizó un análisis de homología empleando la herramienta BLAST nucleotide de NCBI. Finalmente, se construyeron árboles filogenéticos en el programa MEGA software 10.
Selección de muestras para la evaluación del efecto del bloqueo de las bombas de eflujo:
Considerando el interés de estudiar la participación de las bombas de expulsión en los mecanismos de multirresistencia, se seleccionaron aislados que presentaban los tres genes de resistencia identificados en el apartado anterior.
Evaluación del efecto del bloqueo de las bombas de eflujo:
Se empleó el ensayo de checkerboard16 para evaluar las interacciones entre dos agentes, en este caso la interacción de los antibióticos ceftazidima, clortetraciclina, ciprofloxacina, ceftriaxona, ampicilina y gentamicina vs el EPI: Phenylalanine-arginine ß-naphthylamide (PAßN) todos obtenidos desde Sigma aldrich.
En la ejecución del ensayo se prepararon diluciones de los antibióticos (0; 0,25; 1; 2; 4; 8; 13; 64; 128 mcg/ mL) y se dispensaron en 50 uL de MHB en cada pozo de la micro placa; cada antibiótico de la combinación a evaluar se diluyó en serie a lo largo del eje de las ordenadas, mientras que para el PAßN se prepararon 6 diluciones (0; 0,76; 3,12; 12,5; 25; 50 mcg/mL) y se adicionaron a lo largo del eje de las abscisas16.
De cada cepa seleccionada cepas se preparó un inóculo de turbidimetría de 0,5 McFarland, con posterior inoculación en cada pozo de 100 uL (5x105 CFU/mL), los platos fueron incubados a 37 °C por 48 h en condiciones aeróbicas; después de la incubación se realizó la lectura a 655 nm para evaluar el crecimiento microbiano16. Es importante mencionar que una sexta cepa de E coli y una de Pseudomona aeuroginosa positivas para los tres genes fue excluida ya que no fue posible reactivarla para los estudios.
Para establecer la interacción, se determinó el índice de concentración fraccionada de inhibición (ΣFIC) con la ecuación modificada de Berenbaum17 donde, ΣTIC = FICA + FICB. FICA es la MIC de la droga A en combinación/MIC de la droga A sola. FICB es la MIC de la droga B en combinación/MIC de la droga B sola16; los resultados se interpretaron de forma que si ΣTIC ≤ 0.5 se consideran sinérgicas, ΣFIC > 0.5 y ≤ 4 se consideran indiferentes o de efecto aditivo y ΣFIC > 4 se consideran antagonistas18.
Análisis de la información.
Los datos obtenidos fueron sistematizados en el Programa Microsoft Excel 2019, graficados y analizados en el programa GraphPad Prisma 5, y las diferencias entre las MIC se analizaron mediante al Prueba T y se consideró significativo el valor de p<0.05.
Consideraciones éticas
El estudio contó con aval del Comité de Bioética de la ESE Hospital Universitario San Rafael de Tunja, Acta de evaluación No 4 del 18 de abril de 2018.
Resultados
La extracción de ADN se realizó en las 70 cepas de bacterias gram negativas obteniendo rangos de concentración entre 80 y 200 ng/uL y una relación A260/A280 superior a 1.6 para todas las extracciones, los genes bla TEM y bla CTM-X1 se encontraron en el 41,4 % de las cepas (29/70), lo cual evidencia que estos mecanismos se expresan en menos de la mitad de las cepas aisladas y evaluadas; el gen bla SHV fue identificado en el 51,4 % de las cepas analizadas (36/70) evidenciando que este mecanismo es más común en el ambiente desde el cual se obtuvieron los asilados bacterianos, detectándose en al menos la mitad de las bacterias Gram negativas aisladas.
Al realizar un análisis por cepas bacterianas, todas las cepas de Pseudomona aeuroginosa (5/5), Enterobacter aerogenes (4/4), Citrobacter fameri (1/1) y Citrobacter freundii (1/1) presentaron al menos un gen de resistencia a betalactámicos, en las cepas de Pseudomona aeuroginosa predominó el gen bla crMXi al igual que en Enterobacter aerogenes mientras que en las cepas de Citrobacter fameri y Citrobacter freundii predomina el gen bla SHV ; las cepas de Escherichia coli presentaron al menos un gen de resistencia en el 91,7 % de las cepas analizadas (22/24) con predominancia del gen bla TEM , seguido por Klebsiella pneumoniae donde el 90.9 % (10/11) de las cepas portan uno o más de los genes evaluados con mayor número de cepas portando el gen bla SHV .
Las cepas aisladas de Enterobacter cloacae presentaron mecanismos de resistencia en el 66,7 % de las cepas (8/12) predominando el gen bla SHV . Finalmente, las cepas de Klebsiella oxytoca y Serratia marcescens presentaron genes de resistencia en el 50 % de las cepas analizadas (1/2) y (5/10) respectivamente, con mayor presencia del gen bla SHV (ver Tabla 1).
Tabla 1 Análisis molecular de genes de resistencia en cepas gram negativas, presencia de genes de resistencia tipo betalactamasas
| Presencia de genes por PCR | ||||||||
|---|---|---|---|---|---|---|---|---|
| Cepa Bacteriana | Número de Cepas | blaTEM | blaSHV | blaCTM-X1 | blaTEM / blaSHV | blaTEM / blaCTM-X1 | blaSHV / blaCTM-X1 | blaTEM / blaSHV / blaCTM-X1 |
| Klebsiella pneumoniae | 11 | 5 | 7 | 6 | 3 | 4 | 3 | 2 |
| Escherichia coli | 24 | 18 | 12 | 11 | 10 | 7 | 8 | 6 |
| Pseudomona aeuroginosa | 5 | 3 | 2 | 4 | 1 | 1 | 3 | 1 |
| Klebsiella oxytoca | 2 | 0 | 1 | 1 | 0 | 1 | 0 | 0 |
| Serratia marcescens | 10 | 1 | 4 | 1 | 0 | 1 | 0 | 0 |
| Enterobacter cloacae | 12 | 1 | 6 | 3 | 0 | 1 | 1 | 0 |
| Enterobacter aerogenes | 4 | 1 | 2 | 3 | 1 | 1 | 0 | 0 |
| Citrobacter fameri | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| Citrobacter freundii | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| Total: | 70 | 29 | 36 | 29 | 15 | 16 | 15 | 9 |
Fuente: autores.
Los resultados indican que la presencia de dos genes de resistencia se identificó en el 64,9 % de las cepas resistentes (37/57), con una distribución equitativa en todas las combinaciones de genes posibles; al analizar las cepas bacterianas se evidencia que la mayor prevalencia de este patrón de resistencia lo expresan Pseudomona aeuroginosa (80 %), Escherichia coli (79,2 %) y Klebsiella pneumoniae (72,7 %), de igual forma, solo estas tres cepas presentaron aislados con presencia de los tres genes evaluados con un porcentaje de 15,8 % de las cepas resistentes (9/57), con una distribución por cepas de 6 de 24 aislados de Escherichia coli (25 %), 1 de 5 aislados de Pseudomona aeuroginosa (20 %), y 2 de 11 aislados de Klebsiella pneumoniae (18,2 %) (ver Figura 1).
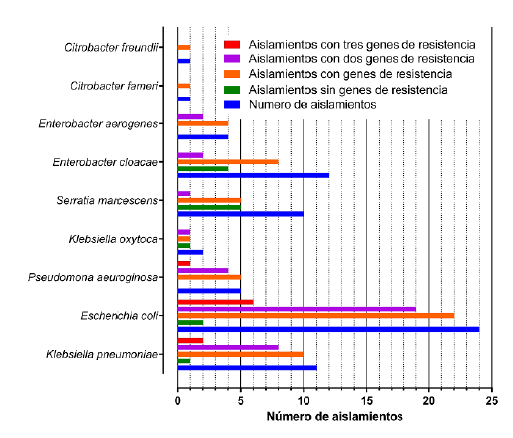
Fuente: autores
Figura 1 Presencia de genes de resistencia blaTEM, blaSHV y blaCTM-X1 evaluado por PCR en los 70 aislamientos de bacterias Gram negativas.
Para el análisis de homologías del gen bla TEM se enviaron a secuenciar 26 amplicones, de los cuales al analizar la secuencia arrojada solo 9 de esos presentaron una longitud de pares de bases adecuada para el análisis BLAST, los datos erróneos encontrados en 17 muestras restantes pueden deberse principalmente a daños por degradación del ADN sufrida durante el proceso de purificación de la banda.
Los resultados mostraron 6 muestras con homología superior al 90 % versus la Escherichia coli isolate GD24, con la variante bla TEM1 (MT387486.1) siendo esta la variante más frecuente entre las cepas aisladas, dos cepas mostraron una homología superior al 98 % con Escherichia coli isolate GD8 con la variante bla TEM-116 allele (MT387477.1) y finalmente una cepa mostró una homología de 100 % con Escherichia coli isolate GD7 class A con la variante bla TEMiSi (MT387476.1).
Para los análisis de bla SHV , se enviaron a secuenciar 21 amplicones positivos para bla SHV obteniendo la secuencia de 18 de ellos, de estos datos la variante de bla SHV más frecuentes fueron la 1 y 2 respectivamente; 12 de los aislados presentaron homologías superiores al 98 % con Klebsiella pneumoniae subsp. pneumoniae strain KPB420/14 beta-lactamase SHV-1 (MF662994.1), y 9 aislados presentaron homologías superiores al 94 % con Klebsiella pneumoniae strain ZUKP128, bla SHV-2 (MF402903.1), se encontraron además dos aislados con homología superior al 96 % con Escherichia coli 713 bla SHV-4 (NG_066766.1); adicionalmente se identificaron otras variantes de este gen con un solo aislamiento cada uno según, siendo homólogos a Klebsiella pneumoniae strain 2207 bla SHV-5 (AF117747.1), Escherichia coli bla SHVS (NG_050115.1), Klebsiella pneumoniae strain NC2, bla SHV-11 (MN786386.1), Klebsiella pneumoniae SHV-49 (NG_050086.1), Escherichia coli D2616 SHV-197 (NG_052582.1) y Klebsiella pneumoniae SHV 222 (NG_062294.1). Al comparar con un estudio previo realizado por Velandia y colaboradores19 en instituciones de salud de Boyacá no encontramos ninguna similitud con las cepas reportadas en este estudio.
La historia evolutiva se infiere utilizando el método UPGMA, el árbol de consenso bootstrap inferido de 500 repeticiones se toma para representar la historia evolutiva de los taxones analizados; las ramas correspondientes a particiones reproducidas en menos del 50 % de réplicas de bootstrap se colapsan, el porcentaje de árboles replicados en los que los taxones asociados se agruparon en la prueba de arranque (500 repeticiones) se muestran junto a las ramas y las distancias evolutivas se calcularon utilizando el método de máxima verosimilitud compuesta y están en las unidades del número de sustituciones de bases por sitios (ver Figuras 2 y 3).
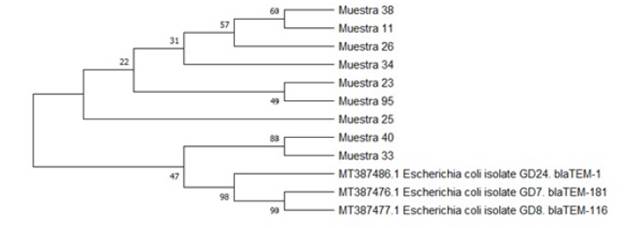
Fuente: autores.
Figura 2 Árbol filogenético de presencia de genes de resistencia blaTEM en bacterias gram negativas.
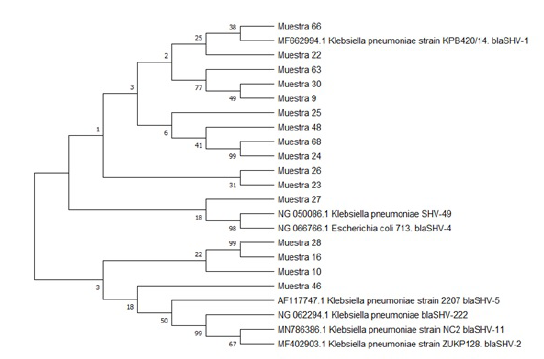
Fuente: autores.
Figura 3 Árbol filogenético de presencia de genes de resistencia blaSHV en bacterias Gram negativas.
Este análisis para bla TEM involucró 12 secuencias de nucleótidos, se eliminaron todas las posiciones ambiguas para cada par de secuencias (opción de eliminación por pares); hubo un total de 861 posiciones en el conjunto de datos final. Los análisis evolutivos se realizaron en MEGA X20, en este se evidencia que a excepción de las muestras 33 y 40, existe poco emparentamiento con las cepas homólogas lo cual indica que pueden haber surgido evolutivamente de forma paralela a las cepas reportadas en Genbank.
Este análisis involucró 23 secuencias de nucleótidos, se eliminaron todas las posiciones ambiguas para cada par de secuencias (opción de eliminación por pares), hubo un total de 1061 posiciones en el conjunto de datos final; al realizar el análisis del árbol filogenético se observa que 9 muestras de las evaluadas se encuentran emparentadas con el gen blaSHV formando parte de dos ramas comunes a la misma, y que esta variante tiene mucha similitud entre todos los aislados, lo cual puede indicar un origen común entre los alelos de las cepas aisladas; el resto de las variantes encontradas están emparentadas entre sí mostrando un origen común a todas estas variantes.
Evaluación del efecto de bloqueo de las bombas de eflujo:
Se seleccionaron 5 cepas de E coli, y 2 cepas de K. pneumoniae que presentaban tres genes de resistencia evaluados (bla TEM ; blaSHV; bla CTXM1 ), correspondientes a aislados provenientes de muestras de secreción de ojo (cepa 22, K. pneumoniae) y de urocultivos (cepas 21, E. coli; 23, E. coli; 24, K. pneumoniae; 31, E. coli; 34, E. coli y 38, E. coli).
Los resultados del análisis de interacción de los antibióticos y el EPI, evidencian que las diferentes cepas pueden expresar diferentes niveles de resistencia y se evidencia la participación de las bombas de expulsión en mayor o menor medida en las cepas evaluadas donde ΣFIC ≤ 0.5 se consideran sinérgicas, ΣFIC > 0.5 y < 4 se consideran indiferentes o de efecto aditivo y ΣFIC > 4 se consideran antagonistas18 (ver Tabla 2).
Tabla 2 Interacción del bloqueo de las bombas de eflujo en las cepas analizadas
| Cepa 21 | Cepa 22 | Cepa 23 | Cepa 24 | Cepa 31 | Cepa 34 | Cepa 38 | |
|---|---|---|---|---|---|---|---|
| Antibiotico vs PAβN | ∑FIC | ∑FIC | ∑FIC | ∑FIC | ∑FIC | ∑FIC | ∑FIC |
| Ceftazidime | 2 | R | 1,01 | 1,01 | 0,14 | 0,08 | 0,05 |
| Antagonista | Resistente | Indiferentes | Indiferentes | Sinérgicos | Sinérgicos | Sinérgicos | |
| Clortetraciclina | R | 0,06 | 0,13 | 0,28 | 0,02 | 0,02 | 0,14 |
| Resistente | Sinérgicos | Sinérgicos | Sinérgicos | Sinérgicos | Sinérgicos | Sinérgicos | |
| Ciprofloxacina | 0,75 | 0,53 | 0,75 | 0,51 | 0,56 | 0,25 | 1,00 |
| Indiferentes | Indiferentes | Indiferentes | Sinérgicos | Indiferentes | Sinérgicos | Indiferentes | |
| Ceftriaxona | 0,63 | R | R | R | 0,02 | 0,02 | 0,05 |
| Indiferentes | Resistente | Resistente | Resistente | Sinérgicos | Sinérgicos | Sinérgicos | |
| Ampicilina | R | R | R | R | 0,02 | 0,02 | 0,02 |
| Resistente | Resistente | Resistente | Resistente | Sinérgicos | Sinérgicos | Sinérgicos | |
| Gentamicina | 0,51 | 1,01 | R | R | 0,25 | 0,50 | 1,00 |
| Sinérgicos | Indiferentes | Resistente | Resistente | Sinérgicos | Sinérgicos | Indiferentes |
R: La cepa mantiene su fenotipo perfil de resistencia.
Fuente: autores.
Los resultados del análisis de interacción de los antibióticos y el EPI, muestra que la inhibición de las bombas de eflujo presenta sinergia con la clortetraciclina en seis de las cepas testeadas, en contraposición de la ciprofloxacina, donde la mayoría de las cepas se comportan de forma aditiva, revelando la necesidad de estudios moleculares más profundos para identificar las bombas involucradas en cada caso.
Finalmente, para dar respuesta al planteamiento inicial de cuál es el efecto del inhibidor sobre los mecanismos de resistencia en las cepas que presentaron interacciones sinérgicas entre ambos tratamientos, se analizaron las concentraciones MIC mcg/mL de cada antibiótico en comparación con la MIC del antibiótico requerida en presencia de PAßN, encontrándose una reducción significativa en la MIC de clortetraciclina, ceftriaxona y ampicilina (ver Tabla 3).
Tabla 3 Efecto del tratamiento con en la MIC de los antibióticos testeados
| Antimicrobiano | Número de cepas con sinergia | MIC mcg/mL | MIC mcg/mL | Prueba t |
|---|---|---|---|---|
| Antibiótico | Antibiótico + PAβN | p (dos colas) | ||
| Ceftazidime | 3 | 29,333 | 1,333 | 0,2443 |
| Clortetraciclina | 6 | 86,667 | 6,792 | 0,0111 |
| Ciprofloxacina | 2 | 128,000 | 32,125 | 0,2043 |
| Ceftriaxona | 3 | 126,667 | 1,500 | 0,0004 |
| Ampicilina | 3 | 128,000 | 0,333 | 4,26E-07 |
| Gentamicina | 3 | 86,667 | 0,833 | 0,1772 |
Fuente: autores.
Discusión
Diversos estudios sobre resistencia bacteriana han demostrado la presencia de diversidad de bombas de expulsión de fármacos dejando sin efecto a la mayoría de los antimicrobianos disponibles4, por lo cual se requiere investigar nuevas estrategias para hacer frente a la problemática mundial.
En un estudio realizado en Teherán, Irán, por Peymani et al, identificaron la presencia de los genes bla CTXM , bla SHV1 y bla SHV12 , en un 17,3 %, 6,7 % y 4 % respectivamente, en cepas de Pseudomonas aeruginosa aisladas a partir de muestras de pacientes que ingresaron al hospital de Qazvin y Teherán21; esto demuestra que la resistencia a antibióticos betalactámicos se encuentra ampliamente distribuida en el mundo, en el departamento de Boyacá, López et al encontraron la presencia del gen bla CTXM en un 61,1 %, bla SHV en 50 % y bla TEM en 38,88 %, los cuáles fueron identificados de cepas de gram negativos aislados a partir de muestras de urocultivo22, genes que fueron hallados en el presente estudio en cepas del mismo tipo pero de diferentes muestras incluida la orina; de otra parte, Zanguña et al en el 2020 reportaron la presencia de los genes para bombas de expulsión MexA, MexC y AcrB en aislamientos de Enterobacter cloacae y Enterobacter aerogenes obtenidos de una institución de salud de Boyacá, y que a su vez presentaron un perfil de resistencia BLEE y AmpC23. Con esto queda en evidencia que en los hospitales del departamento de Boyacá circulan cepas bacterianas que pueden estar empleando bombas de eflujo para bloquear el efecto de los antimicrobianos, lo que lleva a replantearse las alternativas farmacológicas para el tratamiento de los diferentes procesos infecciosos.
Recientemente se ha publicado que en bacterias gram negativas, la transformación por transferencia de plásmidos que codifican para la bomba AcrAB-TolC, están implicadas en mecanismos de resistencia a tetraciclinas6, por lo que se hace indispensable el desarrollo de técnicas y herramientas que permitan diferenciar actividades inhibidoras de eflujo multidrogas y desestabilizadoras de la membrana externa de inhibidores de las bombas de expulsión en las bacterias, facilitando la identificación de EPI, capaces de restaurar la efectividad de los antibióticos disponibles7,8.
Al analizar los resultados obtenidos del ensayo de interacción Checkerboard, podemos evidenciar que las dos cepas de K. pneumoniae (cepas 22 y 24) incluidas en el estudio resultaron ser resistentes al menos a tres de los antibióticos testeados, incluso en presencia de un bloqueo de las bombas de expulsión.
En este caso Baron y Rolain en el 20183 realizaron un estudio en cepas de bacterias gram negativas, evaluando el efecto de los inhibidores de las bombas sobre la resistencia a colistina empleando CCCP, 2,4-dinitro-phenol (DNP), PAßN, reserpine, omeprazole y verapamil, encontraron que solo el CCCP presentó efectos sinérgicos, rescatando la susceptibilidad de las bacterias a la colistina. Esta diferencia puede estar relacionada a los mecanismos de acción reportados para estas moléculas inhibidoras, por ejemplo, el CCCP presenta como mecanismos de acción la inhibición del potencial energético, afectando la disponibilidad de protones requeridas para el funcionamiento de las bombas de expulsión descritas entre las familias 1 a 4, que requieren un gradiente de protones para el mecanismo de transporte antiporte protón/ sustrato9,10.
Por otra parte, el mecanismo reportado para el PAßN, corresponde a un mecanismo de inhibición por competencia con el antibiótico, o por el sitio de interacción de este con la bomba de expulsión24,25. Adicionalmente, Schuster et al en 201926, describieron dos mutantes generados de forma aleatoria desde la cepa E. coli 3-AG100 que presentaban una alteración que disminuye considerablemente el efecto sinérgico de PAßN frente a compuestos lipofílicos de gran tamaño, como es el caso de la ciprofloxacina y la clortetraciclina.
Estos datos pueden explicar, al menos en parte, porque las cepas de K. pneumoniae evaluadas presentan una respuesta baja a la inhibición con el PAßN, de esta forma se recomienda someter estas cepas resistentes a la prueba, empleando un EPI como el CCCP u otro inhibidor de potencial eléctrico mediado por protones; otra de las estrategias publicadas para estas cepas son las combinaciones de antibióticos betalactámicos27,28 o carbapenémicos29, donde se evidencia un potente efecto sinérgico al combinar los fármacos antibióticos, la inclusión de la colistina en conjunto con otros antibióticos30,31, con péptido sintético de la lactoferrina humana32, o en combinación de aceites esenciales extraídos de variedades vegetales33, por lo cual se pueden evaluar estas combinaciones en presencia del inhibidor de las bombas de expulsión.
De igual forma, dos de las cepas de E. coli (cepa 21 y 23) presentaron mecanismos de resistencia que permitieron mantener su fenotipo aún frente al inhibidor de las bombas empleado, por lo que se recomienda aplicar los mismos diseños propuestos para las cepas de K. pneumoniae anteriormente descritas.
Las cepas de E coli restantes (cepas 31, 34 y 38), las cuales presentaban un fenotipo de resistencia a tres antibióticos en común (ampicilina, ceftriaxona y ceftazidime) mostraron un resultado positivo al presentar sensibilidad a los antibióticos testeados en presencia del inhibidor PAßN, demostrando que, en estas cepas, el mecanismo competitivo de PAßN fue efectivo en el bloqueo de las bombas de expulsión involucradas, al igual que se reportó en cepas de E. coli aisladas de mastitis bovina34.
Los resultados del análisis con ceftazidime mostraron que tres cepas de las evaluadas (cepa 21, 23 y 24) no presentan ningún efecto, pues no se generan cambios en las MIC en presencia del EPI, al igual que lo reportado para Vibrio sp.35; por otra parte, tres cepas de E. coli, presentaron efecto sinérgico en presencia del EPI, de esta forma no hay datos concluyentes en nuestro estudio sobre la interacción del PAßN con el antibiótico evaluado.
Los análisis de clortetraciclina, muestran que solo la cepa 21 se comporta como una bacteria resistente a todas las concentraciones probadas; sin embargo, en las otras cepas testeadas, la presencia del EPI muestra un efecto sinérgico con el antibiótico, mostrando que el mecanismo de competencia del PAßN se muestra suficiente para inhibir la expulsión de la clortetraciclina.
Al analizar los resultados de la resistencia a ciprofloxacina, solo dos cepas de las evaluadas (cepas 24 K. pneumoniae y cepa 34 E. coli) presentaron un efecto sinérgico en presencia del EPI aplicado, con una disminución de 2 y 128 veces respectivamente. Al considerar los resultados reportados por Abdel-Halim et al en 201936, en donde se evalúa el efecto de inhibidores de las bombas de expulsión en cepas de E. coli MDR, se evidenció que diferentes inhibidores pueden tener un comportamiento muy diferente en las mismas cepas bacterianas expuestas a ciprofloxacina, donde la domperidona disminuyó 32 veces las concentraciones de ciprofloxacina requerida, mientras que la reserpina solo disminuye 8 veces la concentración requerida.
Wei et al en el 2020 reportan un estudio realizado en cepas de E. coli donde el tratamiento con un inhibidor extraído de la hierba china Artemisia annua, identificado como artesunato, produce una disminución de la MIC de ciprofloxacina en las cinco cepas MDR evaluadas37. Nuestros resultados indican además que de todas las cepas evaluadas solo dos de ellas presentan efectos positivos a la inhibición de las bombas de expulsión con PAßN. En un estudio realizado en cepas de Salmonella por Kang et al en 201438, se demostró que estas cepas de bacterias gram negativas, incrementan la expresión de las bombas de expulsión por presión selectiva mediante la exposición a ciprofloxacina. En referencia a las cinco cepas en las que no se evidencia una interacción positiva en los resultados, Pun et al en el 2023 reportan que en estudios con la bacteria Pectobacterium brasiliense 1692, la cual presenta una bomba de eflujo homóloga a la AcrB de Escherichia coli, la aplicación del EPI no genera una respuesta sinérgica con este antibiótico39, indicando que el mecanismo de resistencia frente a la ciprofloxacina puede ser independiente de las bombas de expulsión.
En referencia a la ceftriaxona, tres cepas (22, 23 y 24) resultaron resistentes a este antibiótico aun en presencia de EPI, en la cepa 21 no existe interacción entre las drogas evaluadas y, al igual que con otros antibióticos, las cepas 31, 34 y 38 en la presencia del PAßN presentan un efecto sinérgico.
En cuanto a la resistencia a ampicilina en este estudio, las cepas de K. pneumoniae y las cepas 21 y 24 de E. coli presentaron resistencia en presencia del EPI empleado, esto puede deberse al mecanismo de acción del EPI seleccionado o resistencia bacteriana al mismo. En un estudio realizado con artesunato, la presencia de este inhibidor de las bombas AcrAB-TolC demostró un efecto sinérgico en combinación con la ampicilina (ΣFIC de 0.3)40. Adicionalmente, en otras cepas bacterianas como la Pseudomonas putida KT2440 la adición de PAßN presenta un efecto sinérgico con la ampicilina incluso en presencia de indol41.
Al analizar la resistencia a gentamicina, en las cepas 21, 31 y 34 (tres de cinco cepas de E. coli analizadas) la adición de PAßN genera una disminución de 2 veces las concentraciones de MIC, similar a lo que reporta Bag et al42 en cepas de E. coli aisladas en muestras de urocultivos. En una cepa bacteriana de Pseudomonas sp. MR 02 super resistente, la presencia del PAßN muestra un efecto sinérgico disminuyendo 2 veces la concentración de gentamicina requerida43, lo que muestra que el mecanismo de expulsión para este antibiótico, independientemente de la cepa y la bomba involucrada es susceptible a la competencia generada con el EPI.
Al evaluar el impacto de la inhibición de las bombas de eflujo con el EPI PAßN sobre las MIC en la literatura se reporta la disminución de Resistencia Antimicrobiana (AMR) a tetraciclinas en cepas MDR de Salmonella enterica a niveles por debajo de punto de corte para establecer que una cepa es resistente a este antibiótico44. Otros estudios de cepas de K. pneumoniae resistente a carbapenémicos demostraron que la aplicación de PAßN generó una disminución de la MIC a tigeciclina en cuatro diluciones y adicionalmente, en este estudio se identifica que la cepa presentaba una sobreexpresión del sistema AcrAB45.
En nuestro estudio encontramos que el tratamiento con el inhibidor provoca una disminución de la MIC en mcg/mL para los antibióticos clortetraciclina, ceftriaxona y ampicilina, encontrándose además con diferencias significativas entre las concentraciones (ver Tabla 3). En los otros antibióticos testados se evidencia una tendencia biológica a la disminución de la MIC pero sin diferencias significativas, posiblemente debido a la necesidad de evaluar un número mayor de cepas, ya que por ejemplo en el caso de la ciprofloxacina en un estudio sobre A. baumannii se reportó que el tratamiento con PAßN disminuyó entre 2 y 4 veces la MIC frente a este antibiótico46, similar a lo evidenciado en nuestros resultados.
Finalmente, se evidenció dos comportamientos diferenciados en las cepas bacterianas 31, 34 y 38 que responden de forma sinérgica frente a la mayoría de los antibióticos, mientras que las cepas 21, 22, 23 y 24 presentan mecanismos de resistencia a los antibióticos que no son susceptibles a la inhibición por el PAßN. Además, frente a cada antibiótico no se evidenció un comportamiento homogéneo en las cepas estudiadas, lo que apunta a que la eficiencia del bloqueo de las bombas de eflujo depende de las bombas presentes y activas en las bacterias más que de los mecanismos de resistencia específicos, como es el caso en la resistencia antimicrobiana. En este punto se hace indispensable un estudio de identificación de la presencia de las bombas de eflujo y el análisis de la expresión de estas.
En los análisis filogenéticos no se encontró una fuerte relación de las cepas estudiadas y reportadas en la región, estos hallazgos nos permiten inferir que las cepas se encuentran en constante evolución respondiendo a las presiones selectivas a las que son sometidas por diversas razones, incluyendo la mala prescripción de antibióticos o la interrupción del tratamiento por parte de los pacientes. Se identifica la presencia de los genes BLEE bla TEM 1, 116 y 181 presentes en secuencias depositadas de Escherichia coli isolate GD24, GD8 y GD7 respectivamente. En relación al gen bla SHV se encontró gran diversidad de genes predominando los genes bla SHV 1 y 2.
En un estudio publicado por Pulido et al en el 2011, donde analizan muestras proveniente de 10 hospitales de la misma región geográfica, identifican que los genes bla SHV más prevalentes fueron bla SHV12 , bla SHV5 , bla SH27 y bla SHV2 47, estudio, años después solo se evidencia que continúa circulando el gen bla SH2 en conjunto con una variedad de genes emergentes en la región, demostrando la constante evolución de estos mecanismos antimicrobianos.
Durante la ejecución del estudio se presentaron como limitantes el bajo número de cepas multirresistentes disponibles en el cepario que se pudiesen incluir en la fase de evaluación del bloqueo de las bombas de eflujo y la disponibilidad de un único EPI para realizar las pruebas. Finalmente se recomienda evaluar la expresión de las bombas de eflujo por qPCR y/o Western Blot.
Conclusiones
En la cepas gram negativas aisladas se detectaron, a través de PCR convencional, genes que determinan la resistencia a antibióticos, como lo son bla TEM , bla SHV y bla CTX-M , así mismo, se determinó que todas las cepas bacterianas presentaron al menos un gen de resistencia, predominando el blaCTX-M. De las setenta bacterias evaluadas se identificaron nueve aislamientos que presentaban los tres genes en conjunto, generando una alerta temprana en la posible aparición de microorganismos multirresistentes.
Por otra parte, el análisis filogenético de los genes bla TEM y bla SHV mostró que las variantes alélicas de bla TEM y bla SHV de mayor circulación en la institución de salud fueron bla TEM-1 , bla SHV-1 y bla SHV-2 . Finalmente, con el análisis Checkerboard y el análisis en la reducción de las MIC de los antibióticos clortetraciclina, ceftriaxona y ampicilina, se estableció que el uso de inhibidores como terapia alternativa en cepas multirresistentes puede ser una opción terapéutica, sin embargo, se deben ampliar los estudios a otras familias de inhibidores para cubrir un mayor número de mecanismos de expulsión y garantizar un bloqueo efectivo en todas las cepas testadas.















