Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Vitae
Print version ISSN 0121-4004
Vitae vol.12 no.2 Medellín Jul./Dec. 2005
ESTUDIO DE CEPAS NATIVAS AMILOLÍTICAS
STUDY OF AMILOLYTIC NATIVE MICROBIAL STOCKS
SÁNCHEZ H. Claudia P. 1*, MEJÍA G. Carlos E.1, FIGUEROA M. Carlos1, ESQUIVIA M. Mabel1, AGUDELO E. Lina M.1, ZAPATA G. Norela2 y GÓMEZ M. Marcela3
1 Grupo de Biotecnología. Facultad de Ingeniería. Universidad de Antioquia. AA 1226. Medellín, Colombia
2 Escuela de Ciencias Agrarias. Politécnico Jaime Isaza Cadavid. AA 4932. Sede El Poblado, Medellín, Colombia.
3 Facultad de Ciencias Agrarias. Ingeniería Agropecuaria. Politécnico Jaime Isaza Cadavid. AA 4932. Sede El Poblado, Medellín, Colombia.
* Autor a quien se debe dirigir la correspondencia: csanchez@udea.edu.co
Recibido: junio 26 de 2005 Aceptado: septiembre 13 de 2005
RESUMEN
En el presente trabajo se realiza el estudio de cepas microbianas aisladas de diferentes nichos ecológicos, productoras de las enzimas amilolíticas, así: procesado de maíz, de papa y de yuca. Los criterios utilizados para el aislamiento de las cepas amilolíticas, son el crecimiento sobre los medios modificados con almidón y la positividad frente al test de lugol. Los medios empleados fueron CZAPEK, PCA y MRS modificados. Las cepas obtenidas son debidamente purificadas, crío conservadas e identificadas bioquímicamente, obteniendo un total de 103 cepas nativas. De éste total de cepas, se seleccionan debido a la similitud fisiológica entre microorganismos aislados, 9 cepas. Estas últimas son identificadas bioquímicamente y se obtienen los géneros Bacillus sp ,Clostridium sp y Kurthia sp. Las enzimas amilolíticas obtenidas a partir de las cepas nativas presentan pesos moleculares que oscilan entre 30 y 150 kDa aproximadamente.
Palabras clave: amiloglucosidasa, α-amilasa, almidón de yuca, enzimas amilolíticas, Bacillus sp,Clostridium sp, Kurthia sp.
ABSTRACT
The present work studies isolated microbial stocks of different ecological niches, amylolitic enzyme producers, such as: cassava, potato and corn starches. The criteria used for the isolation of the amylolitics stocks were the growth on modified starch medium and positive results in the test of lugol. The cultures employed were modified CZAPEK, PCA and MRS. The obtained stocks were properly purified, conserved, and biochemically identified leading to a total of 103 native stocks. From these, 9 were selected based upon their similarity between isolated microorganisms and were biochemically identified. The resulting genuses were Bacillus sp, Clostridium sp and Kurthia sp. The obtained amylolitics enzymes from the native stocks display molecular weights that oscillate between approximately 30 and 150 KDa.
Keywords: amiloglucosidase, α-amilase, cassava starch, enzymes amylolitics, Bacillus sp,Clostridium sp, Kurthia sp.
INTRODUCCIÓN
La producción de jarabes glucosados y/o fructosados se basa en la degradación del almidón, y ha sido empleada industrialmente desde la década del 70. Desde entonces, se han venido desarrollando procesos enzimáticos que permiten transformar el almidón en los llamados jarabes con alto contenido de glucosa y/o fructosa, dando origen a nuevos productos con mayor poder edulcorante y menor costo en relación con el azúcar de caña (1, 2). La producción de jarabes se da en dos etapas, en la primera se utiliza una enzima a-amilasa y en la segunda una amiloglucosidasa. En el mundo actualmente se buscan enzimas α amilasas y amiloglucosidasas con características diferentes a las ya comercializadas (3).
Los jarabes glucosados son soluciones de glucosa en alta concentración que se utilizan en diversas aplicaciones de la industria alimenticia y biotecnológica (1). Los jarabes de glucosa se obtienen por hidrólisis enzimática del almidón (3, 4). Este proceso requiere dos etapas, la primera etapa, denominada de licuefacción, que consiste en una hidrólisis de las largas cadenas de almidón para obtener maltodextrinas (cadenas mas cortas de almidón), esta es llevada a cabo por la enzima α-amilasa que corta los enlaces glicosídicos α-1,4 internos en forma aleatoria del almidón. En la segunda etapa, denominada sacarificación se hidrolizan los enlaces α-1,4 y/o α-1-6 del terminal no reductor de la cadena de dextrina, por la enzima amiloglucosidasa, liberando glucosa (5, 6).
Hasta el momento el proceso de hidrólisis enzimática del almidón hasta glucosa, se ha llevado a cabo en dos etapas, licuefacción y sacarificación, por lo que ha sido necesario cultivar y manipular dos microorganismos productores de enzimas amilolíticas separadamente, conllevando a un alto consumo de energía y de equipos para el proceso además de las diferencias existentes entre el pH y la temperatura que necesitan las dos enzimas, es difícil que ambos actúen simultáneamente con el almidón (3, 6).
Las perspectivas futuras con respecto a la hidrólisis del almidón es acortar las etapas del proceso, a una sola etapa ya sea con dos cepas microbianas juntas con sus respectivas enzimas, con una sola cepa con las dos enzimas amilolíticas, o con una enzima que tenga capacidad de romper todo tipo de enlace de las moléculas de almidón como lo hace la Glucoamilasa.
La amiloglucosidasa:AMG (Glucoamilasa o alfa-1,4 glucohidrolasa, EC 3.2.1.3) es una enzima GRAS (Generally Registred As Safe), extracelular producida por los hongos de los géneros Aspergillus, Rhizopus, principalmente, y de Mucor, Candida, Saccharomyces, Schwanniomyces, y de los géneros bacterianos Bacillus, Clostridium, Arthrobacter y una enzima tipo Glucoamilasa de Flavobacterium (7, 8. 9. 10, 11). En otros trabajos se presenta un alineamiento de 14 amiloglucosidasas por análisis de clusters hidrofóbicos (12, 13).
La enzima es una glucoproteína que contiene manosa, glucosa, galactosa y ácido urónico, con un peso molecular de 60-100.000 Da., con un pH óptimo de 4.3-4.5, que hidroliza fácilmente enlaces glicosídicos alfa-1,4 y alfa-1,6 con menor afinidad (14). La rata de hidrólisis enzimática es afectada por el tamaño del sustrato, por la estructura y los enlaces en la secuencia de la cadena. Al usar una preparación altamente purificada de A. niger se demostró que la amiloglucosidasa hidroliza maltosa 100 veces más rápido que isomaltosa, mientras que la panosa se hidrolizada en un 45 % de la rata de maltosa, lo que indica que los enlaces alfa-1,6 cerca a los enlaces alfa-1,4 fueron atacados más rápidamente que los mismos enlaces solos. Con enlaces alfa-1,6, un incremento en la longitud de la cadena causa un incremento en la rata de hidrólisis. La glucoamilasa tiene gran afinidad por cadenas largas, así la rata máxima incrementa con la longitud mientras que la constante de Michaelis disminuye (7).
La amiloglucosidasa se utiliza a gran escala en la industria del procesado del almidón en tanques discontinuos a 55-60°C y pH de 4.5 y con períodos de incubación de 48-92 h. para transformar las dextrinas formadas por la alfa-amilasa en glucosa. El calcio la estabiliza frente a la desnaturalización por el calor o el pH (9, 15, 16).
En las industrias de alimentos y bebidas, las enzimas han sido aplicadas satisfactoriamente desde hace muchos años y la necesidad de controlar los costos es dominante. Por ejemplo, en la producción de edulcorantes se han venido reemplazando la sacarosa de caña por otros provenientes de una materia prima más barata y accesible; como es el almidón de maíz que se puede transformar en glucosa y de ella producir jarabes ricos en fructosa con mayor poder endulzante y a menor costo, además, de las ventajas que tiene por ser producidos por vía enzimática y no por vía química. (1, 16).
Desde hace más de una década, en el mundo se están buscando enzimas α amilasas y amiloglucosidasas con características diferentes a las ya comercializadas. Esto se hace mediante estudios de bioprospección microbiana, que tratan de identificar las potencialidades de la flora microbiana presente en los diversos nichos ecológicos del mundo, que posibiliten la formación de industrias productoras de enzimas y las industrias derivadas de su aplicación (4, 7, 17, 18, 19, 20, 21, 22). En Colombia no se han realizado este tipo de estudios, pese a que posee una elevada diversidad biológica, esto se sustenta en la poca información publicada al respecto (23, 24). Por tal razón, el objetivo del presente trabajo fue el estudio de cepas microbianas aisladas de diferentes nichos ecológicos, productoras de enzimas amilolíticas. Los resultados obtenidos incluyen el aislamiento de 103 cepas amilolíticas. De estas, debido a la similitud entre microorganismos aislados, se seleccionaron 9 cepas. Estas últimas fueron identificadas bioquímicamente.
De las cepas estudiadas se seleccionó la que presentó mayor halo en la prueba de lugol (Bacillus sp) para establecer si presentaba mayor actividad amilolítica, para lo cual se estudió el efecto que presentan diferentes fuentes de carbono en la expresión de dicha enzima. Para la cual no se obtuvieron resultados satisfactorios de actividad amilolítica tanto para alfa amilasa como para amiloglucosidas, en cinéticas de 72 horas de duración.
MATERIALES Y MÉTODOS
Microorganismos
Se aislaron cepas microbianas de diferentes fuentes de almidón (maíz, papa y yuca) procedentes de sus respectivas procesadoras. Se identificaron por medio de coloraciones y crecimiento en cultivos selectivos (MRS, CZAPEK y PCA) (25), para que crecieran, según fueran mohos, levaduras o bacterias.
Recolección de las muestras de almidón
Recolección de las muestras de almidón de maíz
Las muestras fueron colectadas de los tanques de procesamiento final y de lavado, de la empresa Industrias del Maíz en 2 frascos estériles de 1000 mL, los cuales se llenaron completamente con la muestra liquida proveniente de los compartimientos descritos, se obtuvieron 1000 mL de muestra por cada tanque mencionado; luego de 2 horas de ser recolectadas las muestras se procedió a su siembra.
Recolección de las muestras de almidón de papa
Las muestras fueron colectadas del tanque de agua de lavado de la empresa Mekato, cuya materia prima es la papa, en un frasco estéril de 1000 mL, el cual se llenó en su totalidad, obteniéndose así 1000 mL de muestra; luego de 2 horas de ser recolectada la muestra se procedió a su siembra.
Recolección de la muestra de almidón de yuca
Las muestras fueron colectadas de la empresa Almidones La María, en tres frascos estériles de 1000 mL, los cuales fueron llenados completamente con almidón sólido perteneciente a tres puntos diferentes del tanque de fermentación, estas muestras fueron enviadas desde la empresa hacia la universidad, se procedió a su inmediato cultivo.
Procesamiento de las muestras
Procesamiento de las muestras de almidón de maíz: por cada muestra de almidón de maíz (una perteneciente al tanque de procesado final y otra proveniente del tanque del agua de lavado), se tomaron 50 de los 1000 mL recogidos inicialmente, luego se resuspendieron en 450 mL de agua peptonada estéril y se homogenizaron durante 10 min.; seguidamente se realizaron diluciones seriadas hasta 10-9 en tubos que contenían 9 mL de agua peptonada.
Se continuó con la siembra, por triplicado, de 100 µL de cada dilución en cajas de petri con los medios: MRS, CZAPECK y PCA, modificados con almidón. Posteriormente se dejaron incubando a 30 °C durante 72 horas, tiempo en el que se hizo el conteo de colonias, descartándose las cajas de petri que presentaban un contenido mucho mayor de 300 colonias.
Procesamiento de la muestra de almidón de papa: la muestra obtenida, fue procesada siguiendo los mismos parámetros utilizados para la muestra de almidón de maíz.
Procesamiento de la muestra de almidón de yuca: se tomaron 100 g. de almidón de yuca, a partir de la mezcla de las tres muestras colectadas y se resuspendieron en 900 mL de solución salina, luego se homogenizaron durante 20 min.; seguidamente se realizaron diluciones seriadas hasta 10-9 en tubos que contenían 9 mL de agua peptonada.
Se continuó con la siembra por triplicado; de 100µL de cada dilución en cajas de petri con los medios: MRS, CZAPECK y PCA, modificados. Posteriormente se dejaron incubando a 30 °C durante 72 horas, tiempo al que se hizo el conteo de colonias, se descartaron las cajas de petri que presentaron un contenido mucho mayor de 300 colonias.
Aislamiento e identificación de cepas microbianas con actividad amilolítica
Aislamiento de las cepas amilolíticas
Con base en el crecimiento sobre los medios modificados con almidón, la respuesta positiva frente al test de lugol, y los aspectos morfológicos de los microorganismos, se hizo el aislamiento de las cepas amilolíticas.
Estas cepas se sembraron por agotamiento en el medio sólido en el que anteriormente habían crecido y se dejaron incubando a 30 °C por 24 horas. Luego se describió su morfología gruesa, teniendo en cuenta tamaño, tipo de borde, coloración y textura; seguidamente se les practicó la prueba de lugol, notificando como positivas aquellas que presentaron la formación de un halo transparente alrededor de la colonia luego de añadirle lugol en la superficie del agar. Las cepas positivas fueron sembradas en 5 mL del respectivo medio líquido e incubadas a 30°C por 24 horas. Las cepas se crioconservaron en glicerol al 30% y a -20°C.
Purificación de las cepas amilolíticas aisladas
Las cepas aisladas fueron purificadas, sembrándolas en 5 mL del respectivo medio de cultivo e incubándolas a 30°C durante 72 horas; seguidamente se les aplicó coloración de Gram, notificándose los resultados obtenidos. Luego se sembraron en medio sólido por agotamiento y se incubaron a 30°C durante 48 horas, después se les aplicó la prueba de filancia (KOH), agregándoles directamente a las colonias este reactivo; se notificaron como positivas aquellas que formaban un filamento, luego de agitarlas con un asa de ojo. Posteriormente se sembraron estas cepas, (tomándose una sola colonia con asa de ojo) en 5 mL del medio en el que habían reportado crecimiento y se incubaron a 30°C durante 48 horas. Por último, se crioconservaron en glicerol al 30% y se dejaron en nevera a –20°C (26).
Identificación bioquímica de las cepas amilolíticas aisladas
Para la identificación de las cepas obtenidas, se tuvieron en cuenta los aspectos morfológicos macro y microscópicos y se realizaron las pruebas bioquímicas de oxidasa, catalasa, citrato, SIM (sulfuro- indol - movilidad) y TSI (triple- azúcar -hierro). Los parámetros utilizados para el análisis de resultados y la clasificación de los microorganismos manejados son los descritos por el Manual de Bergeys (27).
Condiciones de cultivo
Se utilizaron frascos tapa rosca de 500 mL conteniendo medio de cultivo PCA en volumen de 150 mL por duplicado, incluyendo el almidón como substrato de degradación para las enzimas amilolíticas y como fuente de carbono para los microorganismos (28). Estos cultivos se colocaron en agitadores orbitales (BOEKEL cerant ORS 200) a una velocidad de agitación de 130 r.p.m., temperatura de 30°C, durante 72 horas.
Determinación del peso molecular de las de enzimas producidas por las cepas amilolíticas aisladas
Las cepas positivas al lugol, fueron crecidas en medio líquido, PCA modificado con almidón, se centrifugaron a 8000 g y 4 °C. El sobrenadante se recuperó y sometió a electroforesis de proteínas en SDS-PAGE, en geles al 8% de poliacrylamida se uso un marcador de peso molecular conocido (Sigma B2787) para determinar así la presencia de enzimas extracelulares y sus respectivos pesos moleculares (26, 29).
Nota: Se estimaron como cepas amilolíticas aquellas que presentaron la prueba de lugol positiva. El principio de esta prueba se basa en la incapacidad que presenta el lugol para pigmentar las zonas en las que el almidón ha sido hidrolizado por acción enzimática. Por esta razón se utilizó la prueba de lugol para la determinación de microorganismos que producen enzimas amilolíticas exocelulares, ya que este test permite el reconocimiento de las mismas por la formación de un halo no pigmentado y de diámetro variable alrededor de aquellas colonias que sintetizan este tipo de enzimas. Este fenómeno se observa cuando se expone el cultivo a la acción del lugol durante un periodo aproximado de 1 a 3 min.
Determinación del efecto de diferentes fuentes de carbono sobre la expresión de proteínas extracelulares para la cepa 24
La fermentación se realizó en erlenmeyers de 250 mL, se sometió a un baño agitado (BOEKEL cerant ORS 200) a una velocidad de agitación de 130 r.p.m. y a 30 °C, 50 mL del medio PCA modificado con 5 g/L de cada una de las tres diferentes fuentes de carbono utilizadas (almidón, dextrinas y glucosa) fueron inoculados con la cepa seleccionada (Bacillus sp). Se realizó toma de muestra en cámara de flujo laminar para evitar la contaminación del cultivo. Posteriormente se sometieron a centrifugación, recuperándose los sobrenadantes a los cuales se les realizó electroforesis SDS-PAGE para determinar la presencia y pesos moleculares de las proteínas obtenidas (23,24, 30).
Fermentación
El inóculo para la fermentación, se preparó con la cepa incubada en cajas petri. Tomando una asada de células y adicionándola a 20 mL de medio líquido PCA, hasta obtener una concentración adecuada, verificada con la lectura de la densidad óptica de la muestra en un espectrofotómetro, a una longitud de onda de 600 nm. Posteriormente se adicionó al medio líquido para ser fermentada en un baño agitado a una velocidad de 130 r.p.m. y 30 °C durante un período de 24 a 96 horas.
Determinación de la Actividad enzimática
Se tomaron 10 mL de muestra de la fermentación a las 24, 48, 72 y 96 horas, en cámara de flujo laminar para evitar contaminación. Posteriormente la muestra fue centrifugada a 4000 r.p.m durante 5 min. a temperatura ambiente. Se tomó el líquido decantado y se practicó la determinación de actividad enzimática de la AMG, utilizando la técnica desarrollada por Industrias NOVO, en la que 1 AGU (unidad de amiloglucosidasa) es la cantidad de enzima que a condiciones estándar hidroliza una micromol de maltosa por minuto a 25° C y pH 4.3 (31).
Se realizó también la determinación de la actividad enzimática de la alfa amilasa siguiendo el protocolo descrito en el método analítico Novozyme. EB-SM- 0009 02/01. Método basado en la degradación de almidón por la enzima alfa amilasa, en el que 1 KNU (Unidad Kilo Novo) es la cantidad de enzima la cual degrada 5 mg de almidón soluble por hora bajo condiciones estándar.
Adicionalmente, se realizó la concentración de las muestras y se les practicó las pruebas de medición de la actividad tanto amiloglucosidasa como alfa amilasa, siguiendo los protocolos establecidos. Para la concentración se tomaron 5 mL de muestra se le adicionaron 15 mL de sulfato de amonio concentrado (solución saturada), se guardó en nevera a 4° C por 60 minutos, luego se centrifugó a 10000 r.p.m por 10 min a 4° C, se eliminó el decantado y se resuspendió el precipitado en 200 µL de solución buffer fosfato (23, 26) .
RESULTADOS Y DISCUSIÓN
Proceso de aislamiento y selección
Se aislaron 103 cepas, teniendo en cuenta los parámetros morfológicos, el crecimiento en los medios modificados con almidón y la positividad frente al test de lugol. Dentro de las cepas obtenidas figuran microorganismos de tipo bacilos y cocos Gram Positivos y Gram negativos. Debido a la marcada similitud fisiológica y bioquímica existente entre algunos de las cepas aisladas, entre ellas, y con base en las diferencias de tipo morfológico, se seleccionaron 13 para su respectiva identificación, las cuales se presentan en la Tabla 1.
Identificación bioquímica
Las cepas amilolíticas seleccionadas fueron identificadas bioquímicamente, encontrando que pertenecen a los géneros Bacillus sp, Clostridium sp y Kurthia sp., siendo el primero el más predominante dentro de las cepas seleccionadas. Los resultados obtenidos a partir del análisis bioquímico de las cepas seleccionadas y de la clasificación según género de las mismas se ve en la Tabla 2. Clasificación de las cepas amilolíticas seleccionadas según género. Los parámetros utilizados para la clasificación y análisis de resultados de los microorganismos manejados son los descritos por Bergeys (27).
Determinación del peso molecular de las de enzimas producidas por las cepas amilolíticas aisladas
Se realizó la prueba de electroforesis SDS-PAGE a los caldos de fermentación de las cepas seleccionadas para determinar el peso molecular promedio de las proteínas presentes después de 48 horas de cultivo. Los resultados se observan en la Tabla 3, donde se obtiene una variación del peso molecular de las proteínas de acuerdo con la especie; el peso molecular oscila entre 38-175 KDa coincidiendo con los datos reportados (5, 31). De los resultados obtenidos es posible que las cepas estén expresando proteínas de AMG, ya que están en rango de pesos moleculares esperados; corresponde entonces realizar la prueba de actividad enzimática para corroborar si son AMG o alfa amilasas.
Cabe anotar que, las a-amilasas microbianas oscilan entre 23-96 KDa, mientras que las amiloglucosidasas están entre 82-114 KDa. Por esta razón, no es clara la determinación si las enzimas producidas son a-amilasas o amiloglucosidasas, dada la diversidad de tamaños moleculares presentes en ellas. Se recomienda una purificación de las proteínas con membranas de diálisis para medir la actividad específica de cada una de estas enzimas.
Determinación del efecto de diferentes fuentes de carbono en la producción de enzimas para la colonia 24
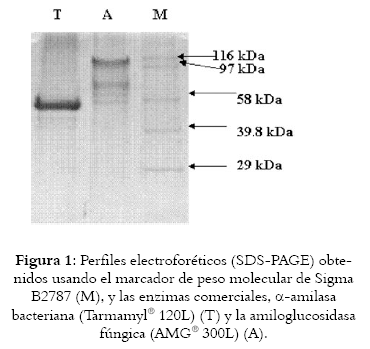
Las modificaciones de la fuente de carbono del medio estándar seleccionado utilizando almidón, glucosa y dextrina en una cantidad de 1 g/L permitieron establecer que el comportamiento de las cepas seleccionadas cuando se utiliza un medio de cultivo con almidón o dextrina como fuente de carbono es similar; es decir, al realizar la electroforesis de las respectivas muestras de las fermentaciones realizadas, se observó que las proteínas presentes en el medio de cultivo modificado con almidón son exactamente las mismas que se expresan en el medio de cultivo modificado con dextrina. Las proteínas expresadas por la cepa en el medio de cultivo modificado con glucosa, y observadas en la electroforesis no presentaron un comportamiento diferente. Estos resultados se pueden apreciar en las Figuras 1 y 2.
En la Figura 1 se pueden apreciar las enzimas patrones utilizadas y el marcador molecular con los diferentes pesos moleculares. La primera banda observada corresponde a la enzima patrón Termamy ® 120L (T), la segunda banda corresponde a la enzima patrón Amiloglucosidasa: AMG® 300L (A). El marcador (M) presenta 7 bandas que corresponden de manera descendente a los siguientes pesos moleculares: 116, 97, 58.1, 39.8, 29 KDa.
Se seleccionó la cepa 24 ya que se logró un mejor crecimiento en medio liquido y presentó el mejor perfil electroforético (mayor definición), en cambio en la cepa 115 si bien se lograron dos bandas, estas fueron muy tenues. Los perfiles electroforéticos de los diferentes aislados presentaron bandas claramente definidas a 30 y 58 KDa, acordes con los tamaños definidos para α-amilasas, como lo muestra la cepa 24 (Véase figura 2).
A partir de lo observado en la electroforesis realizada a las muestras tomadas de las fermentaciones, se aprecia que la cepa 24 presenta trazas de diferentes proteínas. Se seleccionó por lo tanto ésta cepa para realizar la determinación de actividad amilolítica y para realizar el seguimiento de la cinética de fermentación. Se eligió, además, el almidón como fuente de carbono, ya que como se observa en las electroforesis éste no inhibe la expresión de diferentes proteínas del microorganismo y el interés es obtener una enzima que degrade este sustrato.
Existen muchos factores como la temperatura, la deshidratación, la purificación, la congelación etc., que pueden desnaturalizar las proteínas y enzimas. La actividad de las enzimas depende de la concentración de éstas en el medio fermentado. La electroforesis es un medio bastante sensible, que no requiere de grandes cantidades de proteína para arrojar resultados positivos. Sin embargo, los protocolos utilizados para la determinación de las actividades, tanto, de amiloglucosidasa como alfaamilasa requieren de una concentración mínima de enzimas para llevar a cabo con éxito la cuantificación. Las fermentaciones realizadas con el medio, y el volumen establecidos en la metodología, no presentaron una concentración de enzima que fuera detectable por los métodos establecidos. Se plantea la necesidad de desarrollar un nuevo diseño experimental que permita trabajar con un volumen mayor de muestra que garantice la concentración mínima de enzima requerida para la determinación de la actividad enzimática.
CONCLUSIONES
Bajo las condiciones de referencia se logró obtener un cepario de 103 cepas amilolíticas, de las cuales 9 presentaron cualitativamente mayor actividad amilolítica y fueron identificadas bioquímicamente, éstas pertenecen a los géneros Bacillus sp ,Clostridium sp y Kurthia sp, presentes en el almidón de maíz. El peso molecular de las proteínas varia de acuerdo con la especie empleada; el peso molecular oscila entre 38-175 KDa
Se plantea la necesidad de realizar futuras investigaciones, orientadas a la experimentación con otros medios de cultivo diferentes al PCA, que proporcionen los nutrientes necesarios para el mantenimiento del microorganismo, y que estimulen la expresión de la enzima amilolítica AMG. A su vez, se puede recomendar una metodología diferente, donde se realice un seguimiento durante un mayor tiempo, con el objetivo de verificar el comportamiento del microorganismo y la producción de proteínas a períodos más largos de fermentación.
AGRADECIMIENTOS
Al profesor Juan Carlos Quintero Díaz, al Politécnico Jaime Isaza Cadavid y al CODI de la Universidad de Antioquia, a las empresas que colaboraron con las muestras para los análisis: MEKATO, Almidones la María, Industrias del Maíz.
REFERENCIAS BIBLIOGRÁFICAS
1. Quintero, R. Aplicaciones de la Biotecnología en la industria alimentaría. En: Congreso Internacional El laboratorio de salud Integral: Una perspectiva. Medellín: Universidad de Antioquia; 1998 [ Links ]
2. Montoya, D. Tecnología de enzimas. En: Primer congreso Internacional de Microbiología Industrial. Santa Fé de Bogotá: Universidad Pontificia Bolivariana; 1998. [ Links ]
3. Agudelo B, Sánchez C, Figueroa C. Producción de jarabe glucosado mediante sacarificación enzimática empleo de un reactor con membrana. Ingeniería Química 2004; 415:196-199. [ Links ]
4. James J, Lee B. Glucoamylases: Microbial Sources, Industrial Applications and Molecular Biology- A Review. J of Food Biochem.1997; 21(6):1-52. [ Links ]
5. Uhlig H. Industrial enzymes and their applications, John Wiley & sons. 1998; 33: 45-64. [ Links ]
6. Sánchez C, Figueroa C. Producción de maltodextrinas a partir de almidón de yuca, utilizando la enzima alfa amilasa. Ingeniería Química. 416: 217-219. [ Links ]
7. Fogarty WM, Kelly CT. Recent Advances in Microbial Amylases. En: Fogarty WM, Kelly CT. Microbial Enzymes and Biotechnology. 2ª ed. Londres: Elsevier Applied Science; 1990. [ Links ]
8. Lealem F, Gashe B. Amylase production by a Grampositive bacterium isolated from fermenting tef (Eraglostis tef). J. Appl. Bacteriol.1994; 77(3):348–352. [ Links ]
9. Aguero JMZ, Macêdo GR, Facciotti MCR, Schmidell W. Influência do pH na síntese e liberação de glucoamylase por A. awamori NRRL 3112 e A. niger NRRL 337. Rev. Microbiol. 1990; 21(4):350–360. [ Links ]
10. Castro et al. Amylolytic enzymes produced by B. amyloliquefaciens MIR 41 in bath and continuous culture. J. chem. technol. biotechnol. 1993; 50(3): 289–294. [ Links ]
11. Mc Tigue MA, Kelly CT, Fogarty WM, Doyle EM. Production studies on the alkaline amylases of three alkalophilic Bacillus sp. Biotechnol. Lett. 1994; 16(6):569–574 [ Links ]
12. Henrissat B, Coutinho PM, Really PJ. Reading - Frame shift in Sacharomyces glucoamylases restores catalytic base, extends sequence and improves alignment with other glucoamylases. Protein Eng. 1994; 7(11):1281-1282. [ Links ]
13. Coutinho P, Ford C. Effect on Thermostability and Catalytic Activity of Introducing Disulfide Bonds into Aspergillus awamori Glucoamylase. Protein Eng. 1998; 11(8):661-667. [ Links ]
14. Coutinho P et al. Automated Docking of alfa-(1,4) and alfa-(1,6)-Linked Glucosyl Trisaccharides in the Glucoamylase Active Site. IEC Research. 1998; 37: 2148-2157. [ Links ]
15. Wiseman A. Biotecnología de los enzimas. 1ª ed. Zaragoza: Acribia S.A.; 1991. [ Links ]
16. Figueroa C, Mazo JC, Sánchez C, Rojas IF, Ríos R. Efecto del Ca2+ sobre la actividad catalítica de glucosa isomerasa inmovilizada durante la producción de jarabes fructosados a partir de almidón de yuca. Aliment. Equipos Tecnol. 2003; 22(184):49-54. [ Links ]
17. Hang and Woodams. Thermophilic glucoamylase from T. flavus. Lett. Appl. Microbiol. 1993; 17: 156–157. [ Links ]
18. Sun Z and Henson. A quantitative assessment of the importance of barley seed α-amylase, α-amylase, debranching enzyme, and α-glucosidase in starch degradation. Arch. Biochem. Biophys. 1991; 284:298–305. [ Links ]
19. De Araújo JM, Da Silva AC, Azevedo João L. Isolation of endophytic actinomycetes from roots and leaves of maize (Zea mays.). Braz. Arch. Biol. Technol. 2000; 43(4): 447–451. [ Links ]
20. Marlida Y, Saari N, Hassan Z, Radus S, Bakar J. Purification and characterization of sago starch-degrading glucoamylase from Acremonium sp. endophytic fungus. Food Chem. 2000; 71(2): 221–227. [ Links ]
21. Saha BC, Zeikus JG. Microbial glucoamylase: biochemical and biotechnological features. Starch. 1989; 41(2): 57–64. [ Links ]
22. Suckling CJ. Enzyme chemistry impact and applications. 2ª ed. London: Chapman and Hall; 1990. [ Links ]
23. Agudelo L. Producción de una Enzima Amilolítica a partir de una Cepa Nativa. Trabajo de grado para optar al título de Ingeniera Química. Medellín Colombia: Universidad de Antioquia; 2003. [ Links ]
24. López R, Jesús M. Caracterización de la producción y actividad de un extracto enzimático con capacidad amilolítica producida por un aislado nativo de Bacillus sp. Medellín Colombia: Universidad Nacional de Colombia. Facultad de Ciencias; 2004. [ Links ]
25. Medios de cultivo. Avalable from: http://www.dsmz.de Accessed: July 18 2004
26. Ortiz B, Ramírez R. Protocolo para electroforesis e inmunotransferencia. Taller de Técnicas de separación de proteínas y modificaciones enzimáticas y no enzimáticas de proteínas. Corporación de Patologías Tropicales. Medellín: Universidad de Antioquia; 1998 [ Links ]
27. Holt JG. Bergeys Manual of Determinative Bacteriology, 9ª ed. Nueva York: Williams & Wilkins, Baltimore 1994. [ Links ]
28. Pamboukian. VI seminary of enzymatic hydrolisis of biomasa. Brasil: Universidade Estadual de Maringá; 1999. 6-10. [ Links ]
29. Sambrook J, Fritsch E, Maniatis T. Molecular Cloning: A Laboratory Manual 1,2 y 3. 2ª ed. Nueva York: Cold Spring Harbor Laboratory Press Plainview; 1989. [ Links ]
30. Duarte A. Introducción a la ingeniería bioquímica. Bogota: Universidad Nacional, Facultad de Ingenierías. 1995. [ Links ]
31. Novo Nordisk Industries, Novo method for the determination of amyloglucosidase activity. http://www.enzymes.novo.dk. Consulado en Julio de 2004. [ Links ]
32. Jennylynd A, James and Jean-Luc, Berger. Purification of Glucoamylase from Lactobacillus amylovorus ATCC 33621, Current Microbiol. 1997; 34(3):186 –191. [ Links ]