Introducción:
Staphylococcus aureus es un patógeno que puede colonizar las mucosas y la piel; y causar severas infecciones invasivas y mediadas por toxinas en el hombre y los animales.
La aparición de cepas deStaphylococcus aureusresistentes a meticilina (MRSA, Methicillin-Resistant Staphylococcus aureus), fue descrita inicialmente en 1960, poco tiempo después de la introducción de la meticilina en la práctica clínica [1]. Actualmente, la meticilina no se utiliza, fue reemplazada por la oxacilina (OXA, Oxacillin), es más estable, por lo que el término correcto seríaS. aureusresistente a oxacilina (ORSA, Oxacillin Resistant Staphylococcus aureus); debido a su rol histórico, el acrónimo MRSA es aún usado para describir estos aislamientos [2].
La meticilino-resistencia está asociada con la presencia del casete cromosómico estafilocócico mec (SCCmec, Staphylococcal Cassette Chromosome mec) que contiene el gen mecA, codifica para la proteína de unión a penicilina 2a (PBP2a, Penicillin Binding Protein 2a), variante de la PBP nativa que interviene en la síntesis del peptidoglicano [1].
La expresión fenotípica del gen mecA suele ser heterogénea, es decir, aunque todas las células porten el gen mecA, sólo 1 de 104a 108la expresan, esta condición dificulta la detección en el laboratorio clínico. Existen diversos factores que intervienen en la expresión del fenotipo resistente, tales como el uso de oxacilina incluida en el agar (6 µg/ml), la incorporación de NaCl al medio de cultivo (4%), la incubación a 30 ó 35ºC y la incubación superior a 24 horas [3].
Los sistemas automatizados de identificación como VITEK® 2 (BioMérieux), el sistema MicroScan® (Siemens) y el PHOENIX® (BD Diagnostics) permiten resultados de detección a OXA entre 4 y 11 horas aproximadamente. Adicionalmente, incluyen la detección de susceptibilidad a cefoxitina (FOX, Cefoxitine), puesto que se ha demostrado in vitro, que es mejor inductor de la producción de PBP2a que la oxacilina, en cepas deS. aureus[2].
Las infecciones por bacterias resistentes a antibióticos, especialmente en la bacteriemia, aumenta los costos de la estancia hospitalaria, el consumo de antibióticos, exámenes de laboratorio, entre otras; en un estudio realizado en hospitales de Bogotá, se encontró un incremento en el costo de la atención asociado con la resistencia a meticilina alrededor del 31% [4].
En Colombia, un estudio sobre la epidemiología de S. aureus permitió conocer los cambios a nivel de la tasa de resistencia y del genotipo de la población bacteriana circulante. El informe científico realizado por la Red Nacional de Vigilancia de la Resistencia Bacteriana y de las infecciones asociadas al cuidado de la Salud del Instituto Nacional de Salud (INS) y el Ministerio de Salud y de Protección Social (MSPS) en el año 2009 reportó un cambio en la tasa de resistencia de 28% en el período del 1997-2000 a 61% para el período del 2003-2006, adicionalmente, se encontraron tasas de resistencia entre 61-71% en las unidades de cuidado intensivo (UCIs) [5].
En nuestro país, los laboratorios de microbiología de las instituciones de salud de mediana y alta complejidad utilizan los métodos automatizados (VITEK® 2, MicroScan® y PHOENIX®) para la identificación y determinación de los perfiles de susceptibilidad de los microorganismos causantes de las infecciones, requiriendo para esto un tiempo aproximado de 3 a 5 días para la obtención del reporte; lo que en ocasiones retarda el establecimiento de tratamientos adecuados.
La prevalencia de MRSA asociada al ambiente hospitalario es alta, convirtiéndose en un problema de salud pública por el impacto clínico y económico que esto genera. En nuestra región la información sobre la epidemiología de MRSA es poca al igual que los estudios a nivel genotípico; por lo cual se consideró realizar este estudio, cuyo propósito fue comparar dos métodos para la identificación fenotípica y genotípica de aislamientos hospitalarios MRSA en centros hospitalarios de Pereira. La investigación permitirá comprender su epidemiología en la región y contribuir a la implementación de medidas costo-efectivas de control y manejo enfocadas al tratamiento y seguimiento de las infecciones causadas por MRSA.
Materiales y métodos:
Tipo de estudio y obtención de la muestra.
Se realizó un estudio transversal con un componente fenotípico y molecular en aislamientos de Staphylococcus aureus que se obtuvieron en los laboratorios clínicos de tres centros hospitalarios prestadores de servicios de salud de mediana y alta complejidad de Pereira, dichos laboratorios están dotados de equipos automatizados de alta precisión y tecnología de avanzada. El período de recolección se realizó desde junio de 2013 hasta agosto de 2014.
Tamaño de muestra.
Para el cálculo del tamaño de la muestra, se realizó una asignación de aislamientos de S. aureus proporcional para cada institución las cuales se clasificaron en Institución A, Institución B e Institución C; basada en el número de aislamientos anuales en cada institución y la prevalencia de MRSA en el país para 2012 que fue de 36%. El cálculo por institución fue: Institución A, 120 aislamientos, Institución B, 60 aislamientos e Institución C, 70 aislamientos.
Criterios de inclusión y exclusión.
Entre los criterios de inclusión se establecieron los siguientes: los aislamientos de S. aureus debían ser cultivos puros; un solo aislamiento por paciente sin importar la edad y el sexo; y todo aislamiento debía tener su reporte de identificación y perfil de susceptibilidad antimicrobiana a nivel fenotípico.
Como criterios de exclusión, no se tuvo en cuenta los aislamientos polimicrobianos y aislamientos de otras especies del género Staphylococcus.
Identificación fenotípica y perfil de susceptibilidad.
La identificación y perfil de susceptibilidad antimicrobiana se llevó a cabo por Microscan® (Siemens) o VITEK® 2 (Biomerieux) “Automated Systems for Susceptibility Testing” del área de microbiología de los laboratorios de cada una de las instituciones participantes. Se consideraron aislamientos MRSA, aquellos con MIC ≥4 µg/mL para oxacilina y cefoxitina.
Cada institución entregó el número calculado de aislamientos de S. aureus, éstos fueron llevados al Laboratorio de Microbiología de la Facultad de Ciencias de la Salud, Universidad Tecnológica de Pereira; donde fueron sembrados en agar nutritivo e incubados a 37°C durante 24 horas para confirmar su viabilidad. Posteriormente, una porción del cultivo fue almacenada en crioviales que contenían caldo BHI con glicerol al 20% para ser preservados a -80°C y otra porción se resuspendió en 1ml de amortiguador TE 1X (Tris-EDTA; pH 8), almacenada a -20°C, hasta su uso para las pruebas moleculares.
Para el análisis genotípico de la detección del gen mecA, se utilizaron todos los aislamientos que fueron resistentes a meticilina por los sistemas automatizados. Para determinar si dentro de los aislamientos MSSA pudieran existir resistentes, se tomó una muestra representativa y se sometió al mismo análisis.
Extracción de ADN.
Se siguió el protocolo de extracción CTAB/Lisostafina [6], estandarizándolo con la enzima lisostafina “AMBI BL” con algunas modificaciones [7]. La cuantificación del ADN obtenido se hizo por densidad óptica (DO260nm) y la calidad con la relación DO260/280nm empleando el NanoDrop 2000 marca BIORAD®, se almacenaron a -20°C, para su posterior análisis molecular.
Confirmación de especie y detección genética de resistencia a meticilina.
Para establecer la concordancia del método fenotípico se tomó como método de referencia la detección del gen mecA.
Se realizó una amplificación por PCR múltiple para confirmar la especie mediante la presencia del gen nuc, y para la resistencia a meticilina, el gen mecA que codifica para la proteína PBP2a. Como control positivo y negativo de la reacción, se utilizaron la cepa ATCC 43300 (MRSA) y la ATCC 29213 (MSSA). Como control interno se usó un par de iniciadores Rib-1 y Rib-2 que reconocen una región del ADN ribosomal 16S [8].
Declaración Bioética.
El proyecto fue avalado por el Comité de Bioética de la Universidad Tecnológica de Pereira, y se contó con el aval del Comité de Ética y/o el aval por el custodio de la información de los pacientes, para este caso el gerente de cada una de las instituciones que aceptaron voluntariamente participar en el estudio. El proyecto fue exonerado de solicitar consentimiento informado de los sujetos de investigación para procesar sus resultados.
Análisis estadístico.
La información se recopiló en una base de datos de Excel. Se procesó la información en STATA Versión 14,0 donde se aplicó pruebas exactas. Se consideraron estadísticamente significativos, valores de probabilidad (p) ≤ 0,05 con un intervalo de confianza del 95%.
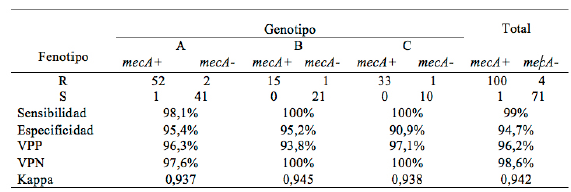
Se determinó la sensibilidad (SEN), especificidad (ESP), valor predictivo positivo (VPP), valor predictivo negativo (VPN), empleando la presencia del genmecA, como método de referencia. Se utilizó el índice kappa como coeficiente de concordancia para medir la confiabilidad y validez del diagnóstico.
Resultados:
Prevalencia fenotípica de la resistencia a meticilina, MRSA.
Se obtuvo un total de 241 aislamientos de Staphylococcus aureus que cumplieron los criterios de inclusión y exclusión, de los cuales el 44,4% presentaron resistencia fenotípica a meticilina (MRSA). Se determinó que la mayor prevalencia fenotípica se encontró en la Institución A con 48,7%. Sin diferencia estadísticamente significativa entre Instituciones.
Prevalencia genotípica por detección del gen mecA.
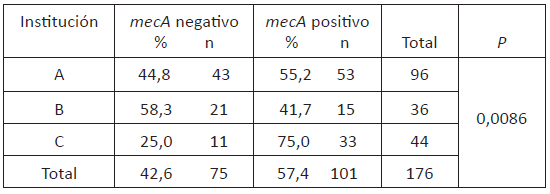
Para la detección del gen mecA se analizaron 183 aislamientos de S. aureus: De 107 con fenotipo MRSA, se detectó el gen mecA en 100 aislamientos (93,5%) y de 134 aislamientos con fenotipo MSSA se estudió una muestra representativa de 76 aislamientos, encontrando que 1,3% (1/76) era portador del gen mecA. Estos resultados establecieron que el total de aislamientos de MRSA fue de 101. La prevalencia genotípica encontrada fue 57,4% en la población general y en la tabla 1 se observan los datos para cada institución encontrando diferencia estadísticamente significativa entre instituciones.
Tabla 1 Prevalencia genotípica de MRSA en instituciones de salud de mediana y alta complejidad de Pereira, Risaralda. Junio 2013- agosto 2014.
El figura 1 muestra los amplicones para diferentes genes detectados y la banda de 162pb corresponde a la amplificación del gen mecA.
Comparación de los métodos fenotípico y genotípico en la identificación de MRSA.
El método fenotípico presenta una sensibilidad y especificidad de 99,0% y 94,7%, respectivamente, cuando se compara con el método de referencia, que para el estudio fue la detección del gen mecA por PCR. La institución B fue la de mayor sensibilidad (100%) y especificidad (95,2%). El índice kappa fue 0,942, indicando esto una alta concordancia entre los métodos comparados. Tabla 2.
Discusión:
La resistencia bacteriana es un problema de salud pública global. El tratamiento de las infecciones producidas porS. aureuses cada vez más complicado, ya que a pesar de la existencia de una gran variedad de antibióticos activos in vitrocontra este patógeno, su habilidad natural para desarrollar nuevos mecanismos de resistencia, compromete su utilidad terapéutica [9].
En los últimos años se había observado un incremento de aislamientos MRSA, sin embargo, en la actualidad a sufrido una leve disminución, en promedio, hasta un 35% de los aislamientos hospitalarios son debido a esta bacteria; con todas las implicaciones a nivel clínico y económico que esto conlleva para el sector salud [4,10].
En esta investigación se encontró una prevalencia promedio de 51%, determinada por métodos fenotípico y genotípico. Se considera alta, al compararla con la tasa de 38,3% encontrada en un estudio previo realizado por este mismo grupo de investigación [11]. Estos valores se encuentran dentro de los rangos reportados previamente para otras ciudades de nuestro país [5]. En el 2009 se reportó una tasa de 41% para Latinoamérica, siendo más baja que la nuestra [12], y prevalencias similares fueron descritas por Stefani et al, en una revisión de epidemiología global de MRSA [13].
Con respecto a la comparación de la detección de MRSA por el método fenotípico frente al método de referencia, se encontraron valores de sensibilidad, especificidad, valor predictivo positivo y valor predictivo negativo superiores a 95% al igual que un índice kappa de 0,942. Estos valores demuestran una buena concordancia entre los métodos comparados y ratifican su utilidad en el diagnóstico de las infecciones de MRSA.
Cabe resaltar, que la eficiencia de estos métodos dependerá de la calidad de los equipos e insumos, y más aún, de la experticia del profesional de microbiología encargado de ejecutar las técnicas. Los resultados obtenidos corroboran que los sistemas automatizados ofrecen un excelente nivel de identificación bacteriana y que los profesionales de microbiología de las instituciones participantes son idóneos y calificados para la realización de esta importante labor.
Se debe tener en cuenta que, aunque el gold estándar, que es la detección del gen mecA por PCR, si bien detecta el tipo de resistencia más frecuentemente encontrado entre las cepas deS. aureus, no incluye todos los mecanismos de resistencia a meticilina que pueden encontrarse en estos microorganismos. Existen cepas deS. aureusresistentes a meticilina que no poseen el genmecAy que deben su resistencia a la modificación de los genes que codifican las PBP normales, a la sobre-expresión de las PBP normales o a la hiperproducción de ß-lactamasa estafilocóccica. Estas cepas presentan un nivel de resistencia limítrofe a oxacilina (4 a 8 µg/ml) y no producen PBP2a [9,14]. En este estudio se encontraron cuatro cepas resistentes a oxacilina por el método fenotípico, que no portaban el gen mecA, esto sugiere que podrían presentar alguno de los mecanismos mencionados anteriormente. Será necesario hacer ensayos adicionales para corroborar cual mecanismo es el responsable de estas variantes. La caracterización de este tipo de cepas es importante, tanto por sus implicaciones terapéuticas como de laboratorio. Estudios clínicos ein vitro, indican que infecciones por este tipo de cepas responden al tratamiento con ß-lactámicos y que los pacientes afectados pueden no requerir medidas de aislamiento y [9,15].
Como conclusión del estudio se pudo determinar que: la prevalencia de aislamientos MRSA en las instituciones de salud de alta complejidad de Pereira fue alta, los métodos fenotípicos usados por las instituciones participantes tienen una excelente sensibilidad y especificidad, que los avala en el diagnóstico de las infecciones por MRSA.
Es recomendable realizar ensayos adicionales a los cuatro aislamientos mecA-/Oxa+ para establecer el mecanismo de resistencia a meticilina. La presencia de este tipo aislamientos nos lleva a plantear ciertas inquietudes: con qué frecuencia se pueden presentar? y en la rutina, cómo clasificarlos cuando se utilicen los métodos que se evaluaron en este estudio?