Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Biotecnología
Print version ISSN 0123-3475
Rev. colomb. biotecnol vol.13 no.1 Bogotá Jan./June 2011
ARTÍCULO DE INVESTIGACIÓN
Incidencia de potyvirus y caracterización molecular de PVY en regiones productoras de papa (Solanum tuberosum L.) de Colombia
Incidence of potyvirus and molecular characterization of PVY in potato (Solanum tuberosum L.) growing regions of Colombia
Título corto: Incidencia de potyvirus en cultivos de papa de Colombia
José Fernando Gil 1 , José Miguel Cotes 2 , Mauricio Marín 3.
1 Investigador, M.Sc., Laboratorio de Biología Celular y Molecular, Facultad de Ciencias, Universidad Nacional de Colombia, Sede Medellín. josefergil@gmail.com
2 Profesor Asociado, Ph.D., Departamento de Ciencias Agronómicas, Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia, Sede Medellín. jmcotes@unal.edu.co
3 Profesor Asociado, Ph.D., Laboratorio de Biología Celular y Molecular, Facultad de Ciencias, Universidad Nacional de Colombia, Sede Medellín. mamarinm@unal.edu.co
Recibido: enero 13 de 2011 Aprobado: mayo 30 de 2011
Resumen
Los problemas virales reducen los rendimientos y la calidad del tubérculo semilla en cultivos de papa de todo el mundo. Esta investigación se planteó con el fin de evaluar los niveles de incidencia de potyvirus en diez de las principales regiones cultivadoras de papa de los departamentos de Antioquia, Boyacá, Cundinamarca y Nariño (Colombia), y las características genotípicas del virus Y de la papa (Potato virus Y, PVY), seleccionado por ser el potyvirus más limitante de este cultivo. Para la evaluación de la incidencia se utilizaron pruebas de Elisa con anticuerpos que reconocen epítopes comunes a los potyvirus, mientras que las pruebas moleculares incluyeron el análisis filogenético de secuencias parciales del gen de la cápside viral de 33 aislamientos, así como la secuenciación de una porción de los extremos 5´ y 3´del genoma de dos cepas colombianas de este virus. Los resultados confirmaron la presencia de potyvirus en los cultivos de los cuatro departamentos evaluados, con una incidencia promedio del 72%, siendo este nivel superior al 56% en todas las zonas evaluadas. Los análisis moleculares del PVY, permitieron asociar las cepas colombianas estudiadas con las razas PVYN y la variante PVYNTN, esta última responsable de la enfermedad conocida en el mundo como PTNRD (Potato tuber necrotic ringspot disease).
Palabras clave: cápside, Elisa, RT-PCR, secuenciación.
Abstract
Potato viruses are responsible for significant reductions in seed quality and crop yields around the world. In this study, we evaluate the levels of incidence of potyvirus in ten potato growing regions of Colombia from the provinces of Antioquia, Boyacá, Cundinamarca and Nariño. As PVY is the most limiting potyvirus in potato farming, a molecular characterization of Colombian PVY strains was also performed. Incidence was evaluated by ELISA using general potyvirus antibodies. Phylogenetic analysis were made on the partial sequence of the capsid gene from 33 isolates. A portion of the 5´ and 3' genome ends was obtained from two Colombian strains. Results confirmed the presence of potyvirus in the four provinces with an average incidence of 72%. The lowest incidence value was 56%. Molecular analysis clustered all Colombian isolates with strains PVYN and PVYNTN, the latter responsible for the disease known as PTNRD (Potato tuber necrotic ringspot disease).
Key words: Capsid, Elisa, RT-PCR, sequencing.
Introducción
Desde tiempo atrás se han venido registrando los efectos que tienen los virus en el cultivo de la papa y principalmente en su rendimiento (Sánchez de Luque et al., 1991; Salazar, 1995). Guerrero y Martínez (1980) reportaron pérdidas del orden del 61% en el rendimiento de la variedad Puracé en Colombia, como resultado de la infección conjunta de Potato virus X (PVX), Potato virus Y (PVY) y Potato leafroll virus (PLRV). Además de estos virus, en las últimas décadas ha cobrado importancia el Potato yellow vein virus (PYVV) (Guzmán et al., 2004; Zapata, 2004), gracias a su eficiente diseminación por parte de Trialeurodes vaporariorum (Salazar, 1995). Más recientemente, se ha reportado en Colombia el Potato mop-top virus (PMTV), cuyo vector, el plasmodiofórido Spongospora subterranea f. sp. subterranea, causa la sarna polvosa de la papa, una enfermedad de gran impacto económico en la agroindustria de este país suramericano (Vélez, 2007; Carreño, 2009). Uno de los grupos virales más limitantes en el cultivo de la papa son los potyvirus, con las especies Wild potato mosaic virus (WPMV), Potato virus A (PVA), Potato virus V (PVV) y PVY. Este último representa el virus de mayor importancia económica del grupo y, además, es la especie tipo del género Potyvirus (Salazar, 1995; Büchen-Osmond, 2010).
El género Potyvirus posee 143 especies aprobadas, lo que lo convierte en el más numeroso de los siete géneros de la familia Potyviridae (Buchen-Osmond, 2010). Las partículas virales tienen una longitud de 680 a 900 nm y un genoma de ARN de cadena sencilla con alrededor 9700 nucleótidos (nt), provisto con una proteína VPg hacia su extremo 5’ y poliadenilación hacia el extremo 3’ (Dijkstra y Khan, 2006).
La morfología del PVY consiste en una partícula con cápside no envuelta, elongada con simetría helicoidal, flexuosa y una longitud de 684 nm a 730 nm (Ogawa et al., 2008). La proteína de la cápside (CP) es el principal componente del virión con aproximadamente 2000 unidades monoméricas. Históricamente, los aislamientos del PVY se han dividido en tres razas principales: PVYO, PVYN y PVYC, de acuerdo con los síntomas inducidos en Nicotiana tabacum cv. Samsun y Solanum tuberosum ssp. tuberosum (Singh et al., 2008). Más recientemente, se han incorporado en la clasificación de las variantes de esta especie viral los criterios del tipo de antigenicidad de su proteína de cápside, los genes de resistencia que vence en el hospedante y la presencia de regiones recombinantes; tal es el caso del aislamiento denominado PVYZ, el cual está serológicamente clasificado como PVYO, pero sobrepasa los genes Nytbr y Nc y elicita el gen Nz en las plantas (Singh et al., 2008). Otro grupo es denominado PVYN-Wilga (PVYN-Wi), que presenta las propiedades biológicas de los aislamientos PVYN, pero está serológicamente clasificado como PVYO, lo que permite deducir que presenta un genoma recombinante entre las secuencias de los dos tipos de aislamientos (Glais et al., 2002). Los síntomas inducidos por las razas PVYO y PVYC incluyen mosaicos suaves a severos, rugosidad de hojas, defoliación, enanismo y finalmente reducción en el rendimiento de las plantas. PVYN y PVYN-Wi generalmente causan síntomas más leves en hojas, pero también se han encontrado aislamientos causando necrosis de tubérculos (Shubert et al., 2007). En los últimos años se ha detectado una variante denominada PVYNTN, serológicamente relacionada con PVYN, pero que causa anillos necróticos en tubérculos de papa, enfermedad conocida como PTNRD y que puede ocasionar pérdidas hasta del 100% en este cultivo (Kogovsek et al., 2008). Estudios del genoma del virus han encontrado que las regiones 5´ NTR (región no traducible del 5’) y el gen P1 corresponden a las secuencias más variables en la especie PVY, siendo responsable hasta de un 28% de la variabilidad total existente entre los grupos PVYO y PVYN (Marie-Jeanne-Tordo et al., 1995; Owaga et al., 2008). Diferentes trabajos indican que es posible que hacia el interior de la región CP (Glais et al., 2002; Ohshima et al., 2000), o del gen HC-Pro (Schubert et al., 2007), se encuentren los motivos responsables de los síntomas asociados a PTNRD. De otra parte, las regiones NIb y 3’-NTR constituyen puntos de variación y recombinación, y pueden servir para diferenciar las razas o los patotipos del virus a partir del estudio de sus secuencias (Kogovsek et al., 2008). Sin embargo, Singh et al. (2008) consideran que hasta el momento no es posible atribuir a un motivo o recombinación particular en el genoma de PVY la causa directa del fenotipo asociado a PTNRD.
El PVY es transmitido de forma mecánica, por injerto y por áfidos de manera no persistente, principalmente por la especie Myzus persicae, aunque también se han reportado Aphis fabae y Macrosiphum euphorbiae, entre otros (Büchen-Osmond, 2010).
En Colombia, con contadas excepciones (Guerrero y Martínez, 1980; Sánchez de Luque et al., 1991; Guzmán et al., 2004), es escasa la información de los niveles de incidencia, severidad y el efecto de cada uno de los virus y de su infección combinada en los cultivos de papa. Más limitado es el conocimiento sobre las características genotípicas de los principales virus que afectan este cultivo, siendo el PYVV del que se dispone de un mayor número de secuencias en las bases de datos moleculares (Offei et al., 2004; Guzmán et al., 2006; Guzmán et al., 2009). Debido a la falta de información actualizada sobre la presencia de potyvirus en los cultivos de papa de Colombia y sus características genéticas, se planteó el presente estudio. Se determinó la incidencia de potyvirus mediante pruebas de Elisa en cultivos de papa de 10 regiones de los cuatro principales departamentos productores de este tubérculo en Colombia, y se secuenció una región parcial del gen de la cápside (CP) en 33 aislamientos de PVY, además de los extremos parciales de 5´y 3´ del genoma de dos cepas procedentes de los municipios de Carmen de Víboral y La Ceja (Antioquia). Todo esto con el fin de definir sus afinidades filogenéticas con las razas y variantes que se han detectado en esta especie viral en diferentes países del mundo.
Materiales y métodos
Incidencia de potyviridae en cultivos de papa de Colombia
Para esta evaluación se realizaron muestreos en ocho cultivos de papa de diferentes variedades ubicados en tres zonas productoras del departamento de Antioquia (zona 1: Oriente —La Unión [05°58′38′′N, 75°21′54′′W], Sonsón [05°42′44′′N, 75°18′50′′W]—, zona 2: Oriente cercano —Santuario [06°08′18′′N, 75°15′51′′W], Marinilla [06°10′36′′N, 75°20′21′′W], Carmen de Víboral [06°05′06′′N, 75°20′19′′W]—, zona 3: Norte Antioqueño —Santa Rosa de Osos [06°38′56′′N, 75°27′48′′W], Don Matías [06°29′23′′N, 75°25′46′′W] —); tres zonas en Cundinamarca (zona 4: Zipaquirá [05°01′42′′N, 74°00′21′′W], zona 5: Villapinzón [05°13′00′′N, 73°36′00′′W], zona 6: Occidente —Madrid [04°26′47′′N, 74°09′25′′W], Cota [04°49′00′′N, 74°06′00′′W]—); dos zonas en Boyacá (zona 7: Turmequé [05°19′00′′N, 73°30′00′′W], Ventaquemada [05°22′09′′N, 73°31′30′′W], zona 8: Tunja [05°32′07′′N, 73°22′04′′W], Siachoque [05°30′00′′N, 73°14′00′′W]), y dos zonas en Nariño (zona 9: Pasto [01°12′49′′N, 77°16′52′′W], zona 10: Ipiales [00°49′49′′N, 77°38′40′′W]). En cada cultivo se realizó una colección aleatoria de cinco muestras conformadas por dos foliolos jóvenes para generar una muestra compuesta (bulk). Una vez en el laboratorio, las 400 muestras (5 muestras x 8 cultivos x 10 zonas) se evaluaron mediante pruebas de ACP-Elisa con anticuerpos monoclonales de la compañía Agdia (Indiana, EE.UU.) para la detección de potyviridae transmitidos por áfidos, siguiendo las instrucciones del fabricante. Los resultados colorimétricos fueron cuantificados en un equipo Multiscan (Labsystem, Finlandia), incluyendo en cada prueba un control positivo (suministrado en forma liofilizada), y un control negativo. Los pozos fueron considerados con reacción positiva cuando la lectura de absorbancia a 405 nm presentó un valor mínimo del doble de la lectura obtenida en el control negativo, siguiendo el criterio de Matthews (1993).
Para el análisis de los datos se consideraron d departamentos (i = 1,2,…,d); mi zonas dentro de departamentos (j = 1,2,…,b; donde b=Σmi), Fij lotes dentro de zonas y departamentos (k = 1,2,…,c; donde c=ΣΣFij), y n observaciones totales. Así, para el vector y de observaciones de presencia/ausencia de potyviridae en la planta l del lote k dentro de la zona j del departamento d se consideró el siguiente modelo lineal generalizado (McCulloch et al., 2008; Littell et al., 2006): donde, en su orden, 1μ+Z1u1+Z2u2+Z3u3+e de, 1 y μ son el vector de unos de tamaño n y la media general; Z1 y u1 son la matriz de incidencia n × a y el vector a× 1 de efectos aleatorios de departamento; Z2 y u2 son la matriz de incidencia n × b y el vector b× 1 de efectos aleatorios de zonas dentro de departamentos; Z3 y u3 son la matriz de incidencia n × c y el vector c× 1 de efectos aleatorios de lotes dentro de zonas y departamentos, y e es el vector de efectos residuales. En este estudio se supone que el vector y sigue una distribución de Bernoulli, y que los efectos de departamentos, zonas dentro de departamentos, y lotes dentro de zonas y departamentos siguen una distribución normal con media cero y varianza σ2u1, σ2u2 y σ2u3.
Para la predicción de la incidencia (I) se utilizó el mejor predictor lineal insesgado (BLUP, por sus siglas en inglés) para el efecto respectivo. Así, por ejemplo, para predecir la incidencia del efecto de los departamentos se sigue la siguiente expresión:
Î=1ˆµ + Z1 û1
Para el análisis estadístico de utilizó el procedimiento Glimmix del programa estadístico SAS versión 9.1.3.
Definición de afinidades filogenéticas de aislamientos de PVY
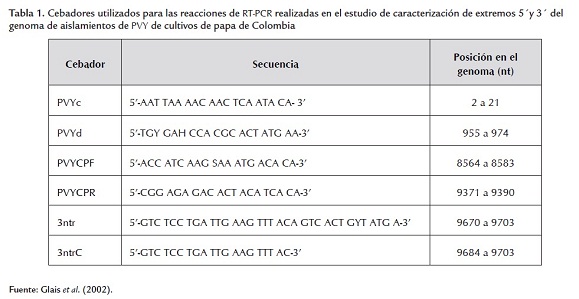
El ARN molde necesario para la técnica de RT-PCR se obtuvo a partir de extracciones de ARN total de foliolos de papa con síntomas de mosaico rugoso, mediante el kit RNeasy plant mini kit (Qiagen, EE.UU.). Para este procedimiento se maceraron 100 mg de tejido foliar utilizando 450 μL de buffer RLT y 4,5 μL de Β-mercaptoetanol, siguiendo las instrucciones del fabricante. Las reacciones de RT-PCR se realizaron en dos pasos (Two-Steps RT-PCR) con los cebadores PVYF (5' ACG TCC AAA ATG AGA ATG CC 3') y PVYR (5' TGG TGT TCG TGA TGT GAC CT 3') (Nie y Singh, 2001). La retrotranscripción se realizó con la enzima M-MuLV Transcriptasa Reversa (20 U/µl) (Fermentas, Lituania) a 37 °C por 60 min y el programa de PCR consistió en 95 °C por 30 s, seguido de 40 ciclos de 95 °C por 30 s, 53 °C por 45 s, 72 °C por 1 min y una extensión final a 72 °C por 5 min. Los amplicones del tamaño esperado fueron purificados directamente del gel mediante el kit QIAquick Gel Extraction (Qiagen), para proceder a su secuenciación en ambos sentidos utilizando los mismos cebadores del RT-PCR en un secuenciador ABI Prism 3730xl (PE Applied Biosystems) de la compañía Macrogen (Corea del Sur).
Las secuencias obtenidas con cada cebador fueron editadas mediante el software BioEdit 6.0.6, construyéndose secuencias consenso y confirmándose su identidad con genes virales por comparación mediante BLASTn con las bases de datos moleculares. El análisis filogenético incluyó las secuencias obtenidas en el estudio y aquellas depositadas en el GenBank representando las razas y variantes principales de PVY. Para esto se alinearon las secuencias mediante el software Clustal W y la matriz generada fue utilizada para obtener un árbol filogenético basado en Máxima Parsimonia utilizando la opción Heuristic search del programa PAUP 4.0b (Swofford, 1998). El soporte de la topología interna de los dendrogramas fue determinado por análisis de Bootstrap con 1000 remuestreos (Felsenstein, 1985). Los árboles fueron caracterizados a partir de los índices de Homoplasia (HI), Consistencia (CI) y Retención (RI), que miden el grado de homoplasia de los caracteres individuales y del árbol completo, a partir del número de cambios de estado observado en un árbol particular y del número mínimo de cambios que se pueden producir (RI), así como de la relación entre la longitud del árbol y su longitud mínima (CI). Como grupo externo de análisis se utilizó la secuencia del potyviridae Pepper mottle virus (PepMV).
Secuencias parciales de los extremos 5´ y 3´ del genoma de PVY
Con el fin de obtener un mayor nivel de información sobre las características de los genomas de los virus bajo estudio se evaluaron seis pares de cebadores (tabla 1) dirigidos a los extremos 5´y 3´del PVY; estas regiones fueron elegidas debido a sus altos niveles de variación (Owaga et al., 2008). Para esto se siguieron los procedimientos de extracción de ARN, RT-PCR y secuenciación descritos anteriormente, pero utilizando dos aislamientos de PVY del departamento de Antioquia (Carmen y La Ceja) seleccionados por la disponibilidad de material vegetal. Para realizar el ensamblaje de los genomas parciales, las secuencias se editaron mediante Chromas, generándose los consensos con ambos cebadores usando Bioedit. Posteriormente, se verificó el marco de lectura correcto utilizando el servidor de Expasy y se procedió a su alineamiento con respecto a genomas completos de PVY disponibles en el GenBank. Las regiones alineadas fueron seleccionadas para evaluar sus niveles de identidad en nucleótidos (nt) y aminoácidos (aa) con la herramienta de matriz de identidad del programa Bioedit. Con las secuencias disponibles para cada aislamiento se generó un ensamble en contigs mediante ContigExpress del software vector NTI (versión 11.0, Invitrogen).
Resultados y discusión
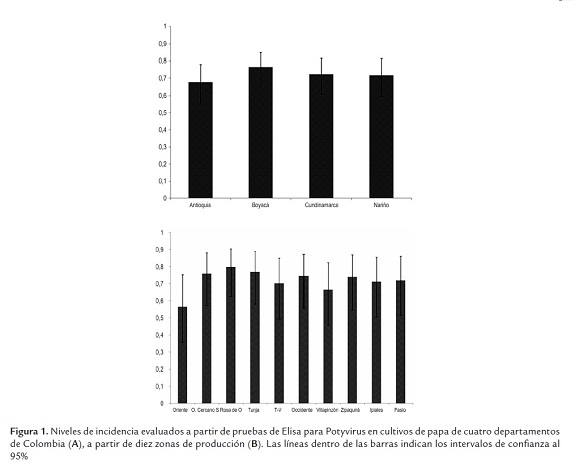
Los resultados de la detección serológica de potyviridae indican su presencia generalizada en las zonas productoras de papa de Colombia, con un promedio de incidencia de 72%, no presentándose diferencias significativas entre los cuatro departamentos evaluados (figura 1A). El análisis de la incidencia potyviral en cada una de las diez zonas evaluadas indicó que las regiones de Santa Rosa de Osos (Antioquia), seguida por Tunja (Boyacá) y Oriente Cercano (Antioquia) presentaban los mayores niveles de detección de este virus, con 80, 77 y 75%, respectivamente; mientras que las demás zonas presentaron valores superiores al 56% en todos los casos (figura 1B). En Colombia se ha reportado la presencia de los potyviridae PVY y PVA (Zapata, 1994), aunque la cuantificación de sus efectos individuales sobre la producción de papa no han sido plenamente determinados. Zapata (2004) indicó que el PVY puede disminuir la cosecha hasta un 80%, y causar la destrucción total del cultivo cuando está asociado con PVA y PVX. Un estudio realizado en el país entre 1984 y 1986 por Sánchez de Luque et al. (1991) en zonas agroecológicas localizadas a diferente altitud, demostró que la prevalencia de PVX, PVY, PVS, PLRV evaluados por métodos serológicos varió entre virus, semestres de cultivo y zonas agroecológicas. La incidencia de PVY en el segundo ciclo de cultivo del primer semestre fue de 4, 11 y 47% para la zona de páramo (3200 msnm), media (2600) y baja (2150), respectivamente, y de 18, 24,5 y 42,5% para los cultivos del segundo semestre. Este estudio también indicó que la infección conjunta de dichos virus podría reducir el rendimiento del cultivo de papa hasta en un 50% en las zonas bajas y en 30% en la región media (Sánchez de Luque, 1991). Por otra parte, Guzmán et al. (2004) reportan que en evaluaciones mediante inmunoimpresión de 800 muestras de campo de diferentes variedades de papa de Nariño y Cundinamarca, el nivel de incidencia de este virus fue del 72%, mientras que en un banco de germoplasma de S. phureja dicho nivel alcanzó el 18,85%. Similarmente, Franco et al. (2009), utilizando esta misma técnica, determinaron que de 44 accesiones de la Colección Central Colombiana de Papa (CCCP), 17 resultaron positivas para PVY (38,7%). Estos reportes y los resultados del presente estudio, sugieren que los programas de manejo fitosanitario viral en Colombia deben ser reforzados, y que a pesar de que el país cuenta con un Programa Nacional de Certificación de Semilla de Papa cuyas tolerancias mínimas para el PVY corresponden al 1% en semilla básica, 2% en semilla registrada y 5% en semilla certificada, dichas exigencias no se ven reflejadas en la situación actual de infección viral de los cultivos, siendo además muy alto el porcentaje de agricultores que aún no utilizan material de siembra certificado.
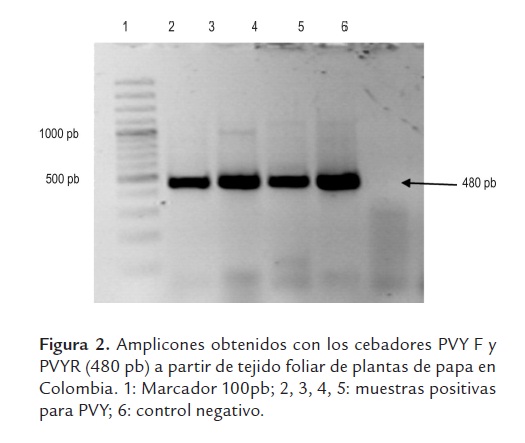
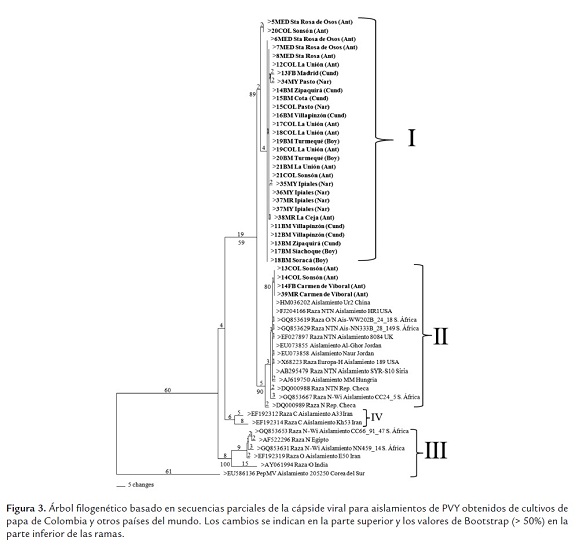
Las pruebas de RT-PCR con los cebadores PVYF y PVYR arrojaron los amplicones del tamaño esperado de ~480 pb (figura 2). El análisis utilizando 33 secuencias de este virus y 20 secuencias de referencia de cepas de PVY representando diferentes razas, variantes y orígenes geográficos, indicó niveles de identidad para la región estudiada superiores al 89%, lo que demuestra que todas las cepas pertenecen a la misma especie, según los criterios taxonómicos definidos para el género Potyvirus (Adams et al., 2005). Es interesante destacar la relación observada entre la mayoría de cepas obtenidas en este estudio y aquellas clasificadas como PVYN y PVYNTN, con las cuales compartieron niveles de identidad superiores al 96%. Por otra parte, las identidades entre cepas colombianas de PVY fueron superiores al 95% en todos los casos, con una gran proporción de cepas compartiendo niveles superiores al 98%. Las cepas de referencia con menores niveles de identidad (89 al 92%) con respeto a las cepas colombianas fueron aquellas representantes de las razas PVYO y PVYC, aunque también ocurrió esto con algunas secuencias de PVYNWi y PVYN. El análisis filogenético utilizó 454 posiciones, 291 de las cuales resultaron constantes, 92 variables pero no informativas y 71 informativas para el método de máxima parsimonia. El dendrograma presentó bajos valores de homoplasia (CI: 0,78, RI: 0,89 y HI: 0,22), lo cual soporta la composición interna de los grupos y la separación de las cepas bajo análisis en tres clados (I, II, III), fuertemente soportados por valores de Bootstrap superiores al 89%. Adicionalmente se presentó un cuarto clado conteniendo dos cepas de Irán, pero con un bajo soporte de Bootstrap (59%). El grupo I incluyó 29 cepas de PVY obtenidas en este estudio de cultivos de papa de Colombia, presentando un muy bajo número de cambios entre sus integrantes (<9); mientras que el clado II agrupó a cuatro cepas de PVY obtenidas en cultivos del oriente de Antioquia con el conjunto principal de cepas de referencia de las razas PVYNTN y PVYN, además de un aislamiento del tipo H procedente de EE.UU. El tercer grupo no incluyó ninguna cepa secuenciada en este estudio, y estuvo conformado por cepas de referencia de las razas PVYO, PVYC, PVYN y PVYNWi, pero no PVYNTN. Finalmente, la secuencia del PepMV presentó valores de identidad cercanos al 70%, lo cual confirmó su utilidad como grupo externo para el análisis (figura 3). Las secuencias fueron depositadas en el GenBank bajo los números HQ335222 a HQ335254 y HQ335258 a HQ335263.
Las reacciones de RT-PCR dirigidas a los extremos 5´ y 3´ del genoma de PVY para dos aislamientos de Antioquia permitieron obtener los amplicones del tamaño esperado con los tres pares de cebadores empleados. Las secuencias generadas se compararon con 11 genomas completos de PVY depositados en el GenBank. Así, para la región P1 y parte del 5’NTR de la cepa Ceja, se encontraron identidades del 99% en nt con cepas de las razas PVYN y PVYNTN y superiores al 99% para aa. Para el mismo aislamiento se obtuvieron dos secuencias diferentes de la región CP, que compartieron identidades superiores al 95% en nt y 97% en aa, con cepas de las razas PVYNTN y PVYN de Europa, EE.UU. y Canadá. El análisis de la cepa Carmen mostró dos tipos de asociación con secuencias de referencia para la región CP. La primera (CP y 3’NTR) se dio con secuencias de las razas PVYNTN, presentando identidades del 99% para nt y del 98% en aa. El segundo fragmento de CP, al igual que el fragmento correspondiente a la región P1 y 5’NTR (figura 4), se asoció con secuencias de referencia de la raza PVYN de EE.UU. y Canadá, con identidades para CP del 96% para nt y del 97% para aa. En el caso de la región genómica P1, se encontraron identidades del 94% para nt con respecto a una cepa canadiense de PVYN, y del 93% para aa, lo cual era de esperarse pues la región 5´del genoma de PVY es la más variable en esta especie (Ogawa et al., 2008), pudiendo incluso presentarse niveles de variación tan bajos como 63% en la región 5'NTR y del 63% para el gen P1 (Ramírez-Rodríguez, 2009). Estos resultados permiten identificar a las cepas colombianas de PVY incluidas en el análisis filogenético y de las secuencias parciales de P1, CP y los UTR 5’ y 3’como pertenecientes a la raza PVYN y dentro de esta, algunas cepas comparten una alta identidad con la variante PVYNTN, lo cual puede ser un indicativo de la posible ocurrencia de la enfermedad PTNRD en el país. Dicha enfermedad se caracteriza por la presencia de anillos necróticos en los tubérculos, que pueden aparecer cuando estos se cosechan, pero más comúnmente cuando se almacenan (Glais et al., 2002).
Debido a que en Colombia el almacenamiento de tubérculos no es una práctica usual, los síntomas de la presencia de la enfermedad PTNRD pueden ser confundidos o verse enmascarados por otros factores, como los cambios climáticos repentinos y los agrietamientos ocasionados por la herramienta de labranza e incluso por otros patógenos del tubérculo. Según Salazar (2006), los síntomas que causan las razas necrosantes observados en variedades de papa nativas de los Andes son diferentes a las reportadas en otros lugares del mundo, siendo más frecuente la aparición de agrietamientos reticulados de la superficie, es decir, de daños similares a los ocasionados por algunos hongos del suelo y por Streptomyces sp. Al parecer los síntomas foliares de la raza PVYNTN son más severos que los producidos por PVYN, produciendo también anillos en la lámina foliar (Kogovsek et al., 2008). Esta enfermedad ya ha sido detectada en diferentes países latinoamericanos incluyendo Brasil (Fonseca et al., 2005), México (Ramírez-Rodríguez et al., 2009) y Perú (Salazar, 2006). Sin embargo, es necesario disponer de mayor información biológica que permita confirmar la inducción de necrosis venal en N. tabacum por parte de dichos aislamientos, y evaluar los eventos recombinatorios que pudieran presentar, para direccionar los estudios tendientes a identificar la sintomatología y su efecto en los programas de certificación de semilla y mejoramiento genético que se realizan en el país.
Conclusiones
Los análisis serológicos de potyviridae en cultivos de papa de 10 regiones de Colombia indican altos niveles de incidencia de este grupo viral en el país, con un promedio de 72%. Esta situación sugiere la necesidad de revisar los programas de certificación de tubérculo-semilla, así como implementar programas de manejo integrado de enfermedades virales, que contemplen las diferentes variables epidemiológicas que afectan el establecimiento y la dispersión de patógenos virales en los cultivos de papa.
Los análisis filogenéticos basados en las secuencias del gen de la cápside de aislamientos de PVY obtenidos en cultivos de papa de Colombia permitieron asociar dichas cepas con las razas PVYN y PVYNTN de diferentes países del mundo.
Agradecimientos
Esta investigación se realizó gracias al apoyo económico y técnico del Ministerio de Agricultura y Desarrollo Rural de Colombia (proyecto 090-2007S4527-87-08), de la Universidad Nacional de Colombia, Sede Medellín, Fedepapa y Fritolay.
Referencias bibliográficas
1 Adams, M. J., Antoniw, J. F., Fauquet, C. M. 2005. Molecular criteria for genus and species discrimination within the family Potyviridae. Archives of Virology, 150: 459-479. [ Links ]
2 Büchen-Osmond, C. The Universal Virus Database of the International Committee on Taxonomy of Viruses. Disponible en: http:// www.ictvdb.org. [Fecha de consulta: 10 de julio de 2010]. [ Links ]
3 Carreño, A. J. 2009. Evaluación de la variabilidad genética de Spongospora subterranea f.sp. subterranea mediante la comparación de regiones ITS del ADN ribosomal de cepas procedentes de las regiones productoras de papa en Colombia. Tesis Maestría en Ciencias Agrarias, Facultad de Agronomía, Universidad Nacional de Colombia, Sede Bogotá. [ Links ]
4 Dijkstra, J., Khan, J. A. 2006. Handbook of Plant Virology. New York: Haworth Press. [ Links ]
5 Felsenstein, J. 1985. Confidence limits on phylogenies: an approach using the bootstrap. Evolution, 39: 783-791. [ Links ]
6 Fonseca, L. N., Inoue-Nagata, A. K., Nagata, T., Singh, R. P., Ávila, A. C. 2005. Diferenciação de estirpes de Potato virus Y (PVY) por RT-PCR. Horticultura Brasileira, 23: 904-910. [ Links ]
7 Franco-Lara, L., Soto, C. A., Guzmán, M. 2009. Detección de los virus PVX, PVS, PVY y PLRV en la Colección Central Colombiana de papa por medio de la técnica de inmunoimpresión (IMI). Revista Facultad de Ciencias Básicas, 5: 130-139. [ Links ]
8 Glais, L., Tribodet, M., Kerlan, C. 2002. Genomic variability in Potato potyviridae Y (PVY): evidence that PVYNW and PVYNTN variants are single to multiple recombinants between PVYO and PVYN isolates. Archives of Virology, 147: 363-378. [ Links ]
9 Guerrero-Guerrero, O., Martínez-López, G. 1980. Evaluación de pérdidas ocasionadas en la variedad de papa Puracé por los virus "Potato Virus X", "Potato Virus Y" y "Potato Leafroll Virus". Fitopatología Colombiana, 9: 3-40. [ Links ]
10 Guzmán, M., Caro, M., García, Y. 2004. Validación de la técnica serológica de inmunoimpresión para detección de diferentes virus que afectan el cultivo de papa. In: Memorias, I Taller Nacional sobre patógenos del suelo, virus e insectos plagas diferentes a Tecia solanivora. Bogotá: Cevipapa. [ Links ]
11 Guzmán, M., Ruiz, E., Arciniegas, N., Coutts, A. 2006. Occurrence and variability of Potato yellow vein virus in three departments of Colombia. Journal of Phytopathology, 154: 748-750. [ Links ]
12 Guzmán, M., Franco, L., Rodríguez, P. A. 2009. Molecular detection of Potato yellow vein virus in leaves and shoot-tubers of Solanum phureja from Colombia. Phytopathology, 99: 189. [ Links ]
13 Kogovsek, P., Gow L., Pompe-Novak, M., Gruden, K., Foster, G.D., Boonham, N., Ravnikar, M. 2008. Single-step RT real-time PCR for sensitive detection and discrimination of potato virus Y isolates. Journal of Virological Methods, 149: 1-11. [ Links ]
14 Littell, R. C., Miliken, A., Stroup, W. W., Wolfinger, R. D., Schabenberger, O. 2006. SAS® for mixed models. 2 ed. EUA: SAS Institute Inc. [ Links ]
15 Marie-Jeanne-Tordo, V., Chachulska, A. M., Fakhfakh, H., Le Romancer, M., Robaglia, C., Astier-Manifacier, S. 1995. Sequence polymorphism in the 5"-NTR and in the P1 coding región of potato virus Y genomic RNA. Journal of General Virology, 76: 939-949. [ Links ]
16 Matthews, R. E. F. 1993. Diagnosis of plant virus diseases. Boca Ratón: CRC Press. [ Links ]
17 McCulloch, C. E., Searle, S. R., Neuhaus, J. M. 2008. Generalized Linear, and Mixed Models. EUA: John Wiley & Sons. [ Links ]
18 Nie, X., Singh, R. P. 2001. A novel usage of random primers for multiplex RT-PCR detection of virus and viroid in aphids, leaves, and tubers. Journal of Virological Methods, 91: 37-49. [ Links ]
19 Offei, S. K., Arciniegas, N., Muller, G., Guzmán, M., Salazar, L. F., Coutts, R. H. A. 2004. Molecular variation of Potato yellow vein virus isolates. Archives of Virology, 149: 821-827. [ Links ]
20 Ogawa, T., Tomitaka, Y., Nakagawa, A., Ohshimab, K. 2008. Genetic structure of a population of Potato virus Y inducing potato tuber necrotic ringspot disease in Japan; comparison with North American and European populations. Virus Research, 131: 199-212. [ Links ]
21 Ohshima, K., Sako, K., Nakagawa, A., Matsuo, K., Ogawa, T., Shikata, E., Sako, N. 2000. Potato tuber necrotic ringspot disease occurring in Japan: its association with potato virus Y necrotic strain. Plant Disease, 84: 1109-1115. [ Links ]
22 Ramírez-Rodríguez, V., Aviña-Padilla, K., Frías-Treviño, G., Silva-Rosales, L., Martínez-Soriano, J. P. 2009. Presence of necrotic strains of Potato virus Y in Mexican potatoes. Virology Journal, 6: 1-7. [ Links ]
23 Salazar, L. F. 1995. Los virus de la papa y su control. Lima: Centro Internacional de la Papa (CIP). [ Links ]
24 Salazar, L. F. 2006. Emerging and Re-emerging Potato Diseases in the Andes. Potato Research, 49: 43-47. [ Links ]
25 Sánchez de Luque, C., Corzo, P., Pérez, O. 1991. Incidencia de virus en papa y su efecto sobre rendimientos en tres zonas agroecológicas de Colombia. Revista Latinoamericana de la Papa, 4: 36-51. [ Links ]
26 Schubert, J., Fomitcheva, V., Sztangret-Wisniewska, J. 2007. Diferentiation of Potato virus Y strains using improved sets of diagnostic PCR primers. Journal of Virological Methods, 140: 66-74. [ Links ]
27 Singh, R. P., Valkonen, J. P. T., Gray, S. M., Boonham, N., Jones, R. A. C., Kerlan, C., Schubert, J. 2008. Brief review: The naming of potato virus Y strains infecting potato. Archives of Virology, 153: 1-13. [ Links ]
28 Swofford, D. L. 1998. PAUP: Phylogenetic analysis using parsimony (* and other methods). Version: 4. Sunderland: Sinauer Associates. [ Links ]
29 Vélez, P. B. 2007. Detección e identificación del Potato Mop - top virus (PMTV) en áreas de producción de papa donde se encuentra Spongospora subterranea en dos departamentos de Colombia. Tesis de Maestría en Ciencias Agrarias, Facultad de Agronomía, Universidad Nacional de Colombia, Sede Bogotá. [ Links ]
30 Zapata, J. L. 2004. Algunos aspectos sobre los virus de la papa en Colombia. En: Memorias I Taller Nacional sobre patógenos del suelo, virus e insectos plagas diferentes a Tecia solanivora, 1. Bogotá: Cevipapa. [ Links ]