Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Biotecnología
Print version ISSN 0123-3475
Rev. colomb. biotecnol vol.18 no.2 Bogotá July/Dec. 2016
https://doi.org/10.15446/rev.colomb.biote.v18n2.52206
DOI: http://dx.doi.org/10.15446/rev.colomb.biote.v18n2.52206
ARTÍCULO CORTO
Diseño in silico y evaluación funcional de genes semisintéticos que confieran tolerancia a fosfinotricina
In silico design and functional assessment of semisynthetic genes that confer tolerance to phosphinothricin
Jenny Paola Jiménez*, Alejandro Chaparro-Giraldo**
* Bióloga, MSc., Universidad Nacional de Colombia - sede Bogotá, Facultad de Ciencias, Departamento de Biología & Instituto de Genética, Grupo de Ingeniería Genética de Plantas.
jenny.jimb@gmail.com
** Ingeniero Agrónomo, MSc., PhD., Universidad Nacional de Colombia - sede Bogotá, Facultad de Ciencias, Departamento de Biología & Instituto de Genética, Grupo de Ingeniería Genética de Plantas.
achaparrog@unal.edu.co
Recibido: febrero 10 de 2016 Aprobado: octubre 22 de 2016
Resumen
La tolerancia a herbicidas es una de las características más usadas en los cultivos GM, con resultados positivos para los agricultores y el ambiente. El punto de partida, es el desarrollo de casetes de expresión que expresen la característica de interés, inicialmente construidos mediante técnicas de biología molecular convencionales. Actualmente, con herramientas de bioinformática y biología sintética, es posible diseñar y probar el constructo in silico, para luego contratar su síntesis. Esta aproximación, permite optimizar la expresión mediante la modificación del uso codónico. En este trabajo se diseñaron y evaluaron en Nicotiana benthamiana versiones semisintéticas de genes que confieren tolerancia al herbicida fosfinotricina. Se realizó un análisis de libertad de operación, con el fin de asegurar que los constructos diseñados no violen derechos de propiedad intelectual en Colombia. Se obtuvieron dos casetes de expresión con libertad de operación, que expresan versiones del gen bar.
Palabras clave: Cultivos GM, libertad de operación, tolerancia a herbicidas, uso codónico.
Abstract
Herbicide tolerance is one of the features most used in GM crops, which has shown positive results for farmers and the environment. The starting point is the development of expression cassettes that express the characteristic of interest, they are initially constructed by standard molecular biology techniques. Currently, by bioinformatics and synthetic biology tools, it is possible to design and test the construct in silico, and then hire their synthesis. This approach allows optimizing expression by modifying the codon usage. In this work there were designed and evaluated semi-synthetic versions of genes in Nicotiana benthamiana, these genes confer tolerance to the herbicide phosphinothricin. It was made an analysis of freedom to operate in order to ensure that the designed constructs not violate intellectual property in Colombia. There were obtained two expression cassettes with freedom to operate, which express versions of the bar gene.
Key words: GM Crops, freedom to operate, herbicide tolerance, codon usage.
Introducción
Una de las tecnologías que ha permitido mejorar la eficiencia en el control de malezas y al mismo tiempo aumentar la productividad en diversos cultivos y en diferentes países, es el uso de variedades Genéticamente Modificadas (GM) con tolerancia a herbicidas (Brookes & Barfoot, 2015). Esta metodología se basa en la introducción de genes foráneos que confieren resistencia a herbicidas no selectivos de amplio espectro, permitiendo que el agroquímico se pueda utilizar en cualquier etapa de la cosecha, sin ocasionar daños al cultivo. El primer paso para el desarrollo de una línea GM, es la obtención de un constructo génico que exprese el gen de interés en la planta. Estos casetes de expresión se han desarrollado mediante técnicas de biología molecular convencionales, sin embargo, gracias a herramientas de bioinformática y biología sintética, es posible diseñar y probar el constructo in silico, para luego contratar su síntesis (Diazgranados et al., 2013).
El diseño in silico de genes semi-sintéticos, constructos genéticos o casetes de expresión, es una herramienta versátil que permite al investigador manejar fácilmente variables en la secuencia de ADN y de los constructos que con el uso de técnicas moleculares convencionales sería muy dispendioso. Esta metodología permite la libre escogencia de secuencias promotoras, terminadoras, genes, regiones no traducidas (UTR), enhancers, péptidos señal, entre otros; insertar o eliminar sitios de restricción que faciliten manipulaciones en el laboratorio y realizar modificaciones en la secuencia codificante como optimizaciones de uso codónico y remoción de secuencias indeseables, factores que mejoran significativamente los niveles de expresión genética (Perlak et al.,1991; Lannacone etal.,1997; Li etal., 2013).
En este trabajo se realizó el diseño in silico de versiones de casetes de expresión que confieren tolerancia al herbicida fosfinotricina o glufosinato de amonio, realizando las optimizaciones necesarias para favorecer su expresión en soya (Glycine max), y se evalúo su funcionalidad mediante transformación de la especie modelo Nicotiana benthamiana. Se efectuó un estudio de libertad de operación para Colombia de todas las secuencias involucradas, con el fin de asegurar que los constructos diseñados no violen derechos de propiedad intelectual, puesto que los resultados se van a usar en un proyecto para la generación comercial de líneas GM de soya a partir de variedades colombianas de esa especie.
Materiales y métodos
Diseño "in silico" de genes semisintéticos
Se realizó el diseño in silico de dos casetes de expresión que confieren la característica de tolerancia a fosfinotricina. Para ello, se descargaron las secuencias de interés del banco genético del NCBI y/o de patentes según se describe a continuación: gen bar (ID X17220.1), promotor 35S (gb|HQ698853.1|:1967-2514), promotor FMV (US 5378619 SEQ ID No. 11). El promotor 35S se aisló del virus del mosaico de coliflor (Brassica oleracea var. Botrytis) o CaMV y corresponde a un ARN 35S. El promotor FMV se aisló del virus del mosaico de escrofularia (Ranúnculus ficaria). Se determinó la secuencia codificante u ORF del gen bar y fue optimizada de acuerdo a los siguientes criterios: modificaciones de uso codónico para la soya, remoción de sitios crípticos de splicing y sitios de terminación prematuros; este proceso se realizó con el programa Visual Gene Developer 1.3 (Jung & McDonald, 2011). Para las modificaciones codónicas, se descargó el uso codónico de la soya (Glycine max) de la base de datos kazusa (Nakamura et al., 2000) y se acopló al software. Mediante cambios iterativos el programa reemplaza los codones originales con otros manteniendo la identidad de la secuencia de aminoácidos. La selección de la secuencia optimizada se realizó teniendo en cuenta parámetros como el índice de adaptación codónico -CAI (Sharp & Li, 1987), el Número efectivo de codones Nc (Wright, 1990) y el contenido de GC. Todos los cambios realizados sobre la secuencia nucleotídica son cambios silenciosos, es decir no modifican la secuencia final de la proteína. El ensamblaje de los genes y de los elementos reguladores de los constructos se realizó con el programa Gene Designer 2.0 (Villalobos et al., 2006), con este mismo software se insertaron sitios de reconocimiento para enzimas de restricción. El análisis de traducción in silico se realizó a nivel de estructura primaria. Las predicciones se realizaron mediante la herramienta ORF Finder del NCBI. Como vector de transformación se seleccionó el pCAMBIA 1301, del cual se eliminaron los genes de selección vegetal y el gen reportero. La síntesis de los constructos y su clonación en el vector de transformación se contrató con la compañía Shanghai Generay Biotech Co., Ltd.
Validación funcional de los constructos
Con el fin de evaluar la funcionalidad de los casetes de expresión, se transformó la planta modelo Nicotiana benthamiana con la cepa LBA4404 de Agrobacterium tumefaciens previamente transformada con uno de los constructos. También se realizaron curvas de selección in vitro para N. benthamiana con fosfinotricina (PPT), con el objetivo de determinar la concentración adecuada para utilizar este agente en el medio de selección. Para todos los ensayos descritos a continuación se utilizaron plántulas de cuatro semanas de edad, y el material vegetal se mantuvo bajo condiciones controladas, con fotoperiodo 16/8 a 28°C. Para la curva de selección se sembraron explantes de hoja sobre medio de regeneración R (sales MS 1X, vitaminas Gamborg 1X, sacarosa 30 g/l, BAP 1 mg/l, agar PTC 7g/l y pH 5.8) con la concentración adecuada del herbicida. En este caso se usó PPT para cultivo de tejidos de Phytotechnology®, a las siguientes concentraciones: 0.2 mg/l, 0.4 mg/l, 0.6 mg/l, 0.8 mg/l, 1 mg/l, 3 mg/l, 5 mg/l, y 7 mg/l. Posteriormente, se transformó N. benthamiana con el constructo diseñado. Para este fin, se cultivó la bacteria en medio LB líquido con acetosiringona 200 µM a 28°C y 200 rpm hasta que alcanzó una DO = 0.6. Se infectaron explantes cuadrados de hoja (1cm2 aprox.) utilizando una fase de cocultivo líquido con la suspensión bacteriana durante dos minutos, y una fase de cocultivo en medio solido durante 24h (medio R con acetosiringona 200 µM). Posteriormente, se realizaron cinco lavados consecutivos en agitación a 100 rpm y temperatura ambiente, con el fin de eliminar la bacteria; finalmente los explantes se secaron y se sembraron sobre medio de selección (medio R con cefotaxime 400mg/l y PPT). Se utilizaron dos concentraciones de selección 0.8mg/l y 3 mg/l de PPT. Las plantas putativamente transformadas fueron evaluadas mediante PCR con el kit KAPA BYOSYSTEMS®. Se diseñaron primers específicos para la secuencia de interés. FW: GCGTCCTGCCGATATTAGG y RV: TCTGTAACGGGCAATACGG. El programa utilizado fue el siguiente: denaturación (95°C 3 min x 1 ciclo), anillamiento (95°C 20 segundos, 66°C 15 segundos, 72°C 30 segundos x 35 ciclos), elongación (72°C 30 segundos x 1 ciclo). Sobre las plantas PCR positivas, se realizó RT-PCR, para evaluar la expresión a nivel de ARN. Para ello se hizo la extracción de RNA a partir de hojas, utilizando el Kit "RNA/DNA/protein purification"® de Norgen biotek corp. Se eliminó la contaminación con ADN, utilizando el kit "DNaseI RNA free"® de Thermo Scientific; se comprobó que la muestra no tenía remanentes de ADN mediante PCR. Luego, se realizó síntesis de ADNc con el kit "First Strand cDNA synthesis"® de Thermo Scientific, y finalmente una prueba de PCR con primers específicos para el gen bar.
Análisis de libertad de operación
Se realizó la búsqueda de patentes nacionales e internacionales. Se utilizaron tres bases de datos internacionales: The Lens (https://www.lens.org/lens/), Patentscope: (http://www.wipo.int/patentscope/en/), Spacenet (http://www.epo.org/), y la base de datos nacional de la superintendencia de industria y comercio (SIC) (http://www.sic.gov.co/es/banco-patentes), todas de acceso público. En las bases de datos internacionales, las búsquedas se realizaron mediante palabras clave dentro de las reivindicaciones. Una vez identificadas las patentes que afectaban el uso de los genes y promotores, se realizó la búsqueda en la base de datos nacional, utilizando palabras clave en los campos, inventor, propietario, y título. Se analizaron los documentos encontrados, y se determinó si los genes de interés, estaban o no cubiertos por derechos de propiedad intelectual en Colombia. Las búsquedas están actualizadas hasta junio del 2014.
Resultados y discusión
Selección de las secuencias
La fosfinotricina (PPT) es el ingrediente activo de varios herbicidas comerciales, se comporta como un inhibidor competitivo de la enzima glutamina sintetasa. La obtención de plantas transgénicas tolerantes a PTT, se ha logrado básicamente mediante la introducción de uno de dos genes: el gen bar de Streptomyces. hygroscopicus (Thompson et al., 1987) o el gen pat de S. viridochromogenes (Wohlleben et al.,1988). Estos genes codifican para la proteína fosfinotricina n-acetil transferasa (PAT); esta proteína acetila el grupo NH2 de la PPT dando lugar a la formación de N-acetil fosfinotricina, que no tiene actividad herbicida (Dröge et al., 1992). Para los constructos diseñados en este trabajo se seleccionó el gen bar, ya que hay varios reportes del uso del mismo como marcador de selección específicamente en plantas de soya (Zhang et al., 1999; Paz et al., 2004; Zeng et al., 2004; Paz et al., 2006). Como elementos promotores de los constructos, se seleccionaron promotores de expresión constitutiva, ya que se requiere que el gen se exprese en todos los tejidos de la planta transformada. Se seleccionaron los promotores 35S (Odell et al., 1985) y FMV (Sanger et al., 1990; Maiti et al., 1997). El p35S es el promotor constitutivo más utilizado en transformación genética de plantas. Y el promotor FMV, ha sido utilizado en varios eventos de transformación genética mostrando una buena expresión del gen heterólogo (ej. MON89788, GTSB77, GT73 entre otros). Como secuencias terminadoras se utilizaron la región terminadora 35S y la secuencia terminadora del gen Rubisco E9 (rbcs, subunidad pequeña de ribulosa-1,5-bifosfato carboxilasa/oxigenasa) de la arveja o guisante (Pisum sativum), que ha sido utilizada en varias líneas GM comerciales (ej, GTSB77, MON89788 entre otras).
Diseño "in silico" de genes semisintéticos
Una de las mayores ventajas del diseño in silico de casetes de expresión, es que permite realizar cambios en las secuencias nucleotídicas que pueden mejorar significativamente la expresión del gen, en el genoma que se va a introducir. Dentro de estos cambios las modificaciones de uso codónico han demostrado ser uno de los factores más influyentes para lograr buenos niveles de expresión (Perlak et al., 1991; Lannacone et al., 1997; Laguía-Becher et al., 2010; Yan et al., 2011; Kucho et al., 2013).
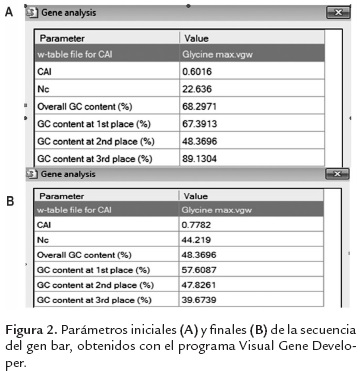
En este trabajo se diseñaron dos casetes de expresión (figura 1). En el primero, denominado Bar-IGP se realizaron modificaciones de uso codónico sobre la secuencia codificante original (gen bar de Streptomyces hygroscopicus), con el fin de acercarlo al uso codónico de la soya. Para este constructo se utilizó el promotor FMV y el terminador E9. Se eliminaron secuencias cripticas de splicing y sitios de poliadenilación prematuros del ORF. La secuencia del gen bar es de origen bacteriano, y tiene un alto contenido de GC en el tercer codón (89 %), que es un valor más alto que el reportado para genomas vegetales (Kawabe & Miyashita, 2003). Las modificaciones de uso codónico permitieron cambiar el contenido de GC a 40 %, que es un valor más cercano al observado en el genoma de la soya (45.69 %). Se cambiaron 153 pares de bases (27.72 %) y 132 codones (71.74 %). La figura 2 muestra una comparación de los parámetros mencionados entre la secuencia inicial y la secuencia final.
Para el segundo constructo (Bar-2) se utilizó la versión nativa del gen, en conjunto con el promotor y el terminador 35S. Así, los dos constructos, uno con modificaciones de uso codónico, y uno con la secuencia nativa del gen, permitirán evaluar el efecto de la optimización codónica en futuros experimentos.
La herramienta ORF Finder demostró que los casetes de expresión generan una proteína de 183 aminoácidos (figura 3). Mediante alineamiento pareado con el algoritmo de Smith-Waterman se determinó un 100 % de identidad entre la proteína predicha y la proteína esperada, indicando que los constructos efectivamente codifican para la fosfinotricina acetil transferasa (P16426).
Evaluación de la funcionalidad de los constructos
Nicotiana benthamiana se ha utilizado ampliamente como sistema modelo en diferentes áreas de la biología, puede ser transformada genéticamente y regenerada con buena eficiencia en tiempos cortos, en comparación con otras especies (Clemente, 2006; Goodin et al., 2008). Con el fin de determinar que los constructos diseñados efectivamente se expresan, se realizaron ensayos de transformación genética en dicha planta. Con el objetivo de usar la tolerancia a fosfinotricina como marcador de selección in vitro, se evaluó la respuesta regenerativa de N. benthamiana frente a este agente. Con todas las concentraciones menores a 1mg/l los explantes mostraron un aspecto muy semejante al tratamiento control. En los tratamientos 0.2, 0.4 y 0.6 mg/l se registró una leve disminución en su capacidad regenerativa, mientras que a 0.8 mg/l la regeneración cayó a 20% (figura 4). Según Lutz et al. (2001), los explantes de Nictiana tabacum cultivados sobre medio con concentraciones de PPT superiores a 4 mg/l se tornan blancos, para N. benthamiana este efecto se observó desde 1 mg/l. Se realizaron experimentos de transformación utilizando dos concentraciones 0.8 mg/l (concentración a la cual el explante conserva su capacidad regenerativa) y 3 mg/l (concentración letal).
Para ello, se transformaron explantes de hoja de N. benthamiana con una cepa de A. tumefaciens LBA4404 previamente transformada con el vector Bar-IGP. Como control negativo se sembraron explantes sin transformar sobre medio de regeneración con PPT. Los explantes sometidos a transformación con A. tumefaciens mostraron una apariencia saludable en contraste con los controles negativos (figura 5), siendo este un primer indicio que el constructo confiere tolerancia a PPT.
Los explantes produjeron regenerantes en la cuarta semana. De los experimentos de transformación se obtuvieron cuatro plántulas en medios de selección con el herbicida, que se evaluaron para la presencia del transgén. Las pruebas de PCR mostraron la presencia del amplicón de 531 pb predicho para el gen bar (figura 6A y 6B carriles 1, 2, 3, 4) en todas las plántulas tolerantes a PPT, mientras que en la planta control no transformada no se presentó el amplicón específico. Se realizó una prueba de RT-PCR sobre una plántula positiva para PCR, con su control correspondiente. Para ello se realizó una PCR sobre el ADNc obtenido a partir del ARN aislado. La figura 7C, muestra que se obtuvo la banda de 531 pb esperada (carril 1), en tanto que para el control negativo (ADNc de planta no transformada) no hubo amplificación. De esta manera se demostró tanto la presencia, como la expresión del transgén en las plántulas transformadas.
Estudio de libertad de operación
Se identificaron las patentes relacionadas con cada uno de los elementos genéticos de los constructos diseñados. Las patentes del gen bar están asignadas a Plant Genetic Systems, N.V., (Bayer CropScience actualmente) y Biogen Inc. La solicitud de patente para este gen fue presentada mediante el mecanismo previsto por el Tratado de Cooperación en materia de Patentes o PCT (por sus siglas en inglés) con el número WO1987/005629, con 19 países designados, dentro de los cuales no se incluye a Colombia. Las patentes estadounidenses más importantes relacionadas con el uso del gen bar, están identificadas con los números US 5561236 (Leemans et al., 1996), US 5646024 (Leemans et al., 1997a), y US 5648477 (Leemans et al., 1997b). En ellas se protege, células vegetales transformadas con el gen que codifica para la acetil transferasa con la capacidad de inactivar inhibidores de la glutamina sintetasa; el proceso de producción de plantas tolerantes a PPT o a cualquier compuesto que tenga alguna fracción de PPT mediante procesos de integración del gen bar de S. hygroscopicus o S. viridochromogenes en el genoma de células vegetales; vectores de transformación genética que comprendan el gen bar; modificaciones en la secuencia de nucleótidos o aminoácidos del gen. La última patente concedida en relación a estos genes tiene el número US 7112665 (Leemans et al., 2006), en esta se amplía la protección del gen bar, a cualquier cambio posible en la secuencia de ADN que pueda producir un polipéptido con las mismas propiedades para generar resistencia a glufosinato de amonio. Está patente fue reasignada mediante la patente US RE44962. La búsqueda de patentes en la base de datos nacional mostró que estas patentes no fueron solicitadas en Colombia.
Las patentes estadounidenses relacionadas con el uso del promotor 35S están identificadas con los números US 5352605 (Fraley et al., 1994), US 5530196 (Fraley et al., 1996), US 5858742 (Fraley et al, 1999) y US 6255560 B1 (Fraley et al., 2001). De acuerdo a la fecha de solicitud, las dos primeras debería haber vencido en el 2014, sin embargo en las bases de datos consultadas sólo se encontraron reportes de pago de la cuota de mantenimiento hasta el 2007, por lo que podrían haber vencido antes. Las patentes US 5858742 y US 6255560 B1 expiraron por falta de pago de la cuota de mantenimiento en el 2007. Para el promotor FMV se identificaron las patentes US 5378619 (Rogers, 1995) y US 5994521 (Maiti & Shepherd, 1999). La búsqueda en la base de datos nacional indica que ninguna de estas patentes fue solicitada en Colombia. No se encontraron patentes que protegieran específicamente el uso de las secuencias terminadoras. Estos resultados sugieren que los elementos genéticos de estudio pueden ser usados en el territorio colombiano sin violar derechos de terceros.
Conclusiones
Se realizó el diseño in silico de constructos que confieren tolerancia a fosfinotricina. El análisis traduccional in silico, demostró que los constructos diseñados efectivamente codifican para la proteína PAT. Los explantes de N. benthamiana cocultivados con la bacteria que porta el vector BAR-IGP, mostraron una respuesta fenotípica diferencial con respecto a los explantes control, indicando que el constructo diseñado podría conferir tolerancia a la fosfinotricina. Las pruebas de PCR y RT-PCR demostraron que las plantas fueron transformadas con el gen de interés, y que el gen se transcribe a ARNm. Estos resultados son una evidencia preliminar de la funcionalidad de los constructos diseñados, indicando que la metodología utilizada para el diseño de genes permite la obtención de constructos funcionales. Una validación funcional completa, podría permitir la utilización de los constructos en experimentos de transformación genética de variedades colombianas de soya. Finalmente, los elementos de los casetes de expresión diseñados no están cubiertos por patentes en Colombia, lo que es importante, para el desarrollo de líneas GM con fines comerciales en el país.
Agradecimientos
Los autores agradecen al Fondo Nacional de la Soya - Federación Nacional de Cultivadores de Cereales y Leguminosas (FENALCE), y a la Universidad Nacional de Colombia por la financiación de este trabajo.
Referencias bibliográficas
Brookes, G., & Barfoot, P. (2015). Global income and production impacts of using GM crops technology 1996 - 2013. GM Crops & Food. 6, 13-146. [ Links ]
Clemente, T. (2006) Nicotiana (Nicotiana tobaccum, Nicotiana benthamiana). En K. Wang (ed). Agrobacterium protocols (pp. 143-153) New Jersey, Humana press. [ Links ]
Diazgranados, C., Sandoval, A.M., & Chaparro-Giraldo, A. (2013). Diseño de un gen semisintético cry1Ac y análisis de la estructura de la proteína traducida. Biotecnología en el Sector Agropecuario y Agroindustrial. Edición especial No. 2, 155-164. [ Links ]
Dröge, W., Broer I., & Pühler, A. (1992). Transgénic plants containing the phosphinothricin-N-acetyltransferase gene metabolize the herbicide l-phosphinothricin (glufosinate) differently from untransformed plants. Planta. 187(1), 142-151. [ Links ]
Fraley, R. T., Horsch, R. B., & Rogers, S. G. (1994). US Patent No. US 5352605. [ Links ]
Fraley, R. T., Horsch, R. B., & Rogers, S. G. (1996). US Patent No. US 5530196. [ Links ]
Fraley, R. T., Horsch, R. B., & Rogers, S. G. (1999). US Patent No. US 5858742. [ Links ]
Fraley, R. T., Horsch, R. B., & Rogers, S. G. (2001). US Patent No. US 6255560. [ Links ]
Goodin, M., Zaitlin, D., Naidu, RA., & Lommel, SA.(2008). Nicotiana benthamiana: Its History and Future as a Model for Plant-Pathogen Interactions. Mol Plant Microbe Interact., 21 (8), 1015-1026. [ Links ]
Lannacone, R., Grieco, P., & Cellini, F. (1997). Specific sequence modifications of a cry3B endotoxin gene result in high levels of expression and insect resistance. Plant Molecular Biology, 34(3),485-496. [ Links ]
Jung, S.K., & McDonald, K. (2011). Visual gene developer: a fully programmable bioinformatics software for synthetic gene optimization. BMC Bioinformatics 12(1), 1-13. [ Links ]
Kawabe, A., & Miyashita, N. (2003). Patterns of codon usage bias in three dicot and four monocot plant species. Genes & Genetic Systems, 78(5), 343-352. [ Links ]
Kucho, K., Kakoi, K., Yamaura, M., Iwashita, M., Abe, M., & Uchiumi, T. (2013). Codon-optimized antibiotic resistance gene improves efficiency of transient transformation in Frankia. Journal of Biosciences, 38(4), 713-717. [ Links ]
Laguía-Becher, M., Martín, V., Kraemer, M., Corigliano, M., Yacono, M., Goldman, A, & Clemente, M. (2010). Effect of codon optimization and subcellular targeting on Toxoplasma gondii antigen SAG1 expression in tobacco leaves to use in subcutaneous and oral immunization in mice. BMC Biotechnology, 10(1), 1-14. [ Links ]
Leemans, J., Botterman, J., De Block, M., Thompson, C., & Mouva, R. (1996). US Patent No. US 5561236. [ Links ]
Leemans, J., Botterman, J., De Block, M., Thompson, C., & Mouva, R. (1997). US Patent No. US 5646024. [ Links ]
Leemans, J., Botterman, J., De Block, M., Thompson, C., & Mouva, R. (2006). US Patent No. US 7112665. [ Links ]
Leemans, J., Botterman, J., Thompson, C., & Mouva, R. (1997). US Patent No. US 5648477. [ Links ]
Li, X., Li, S., Lang, Z., Zhang, J., Zhu, L., & Huang, D. (2013). Chloroplast-targeted expression of the codon-optimized truncated cry1Ah gene in transgénic tobacco confers a high level of protection against insects. Plant Cell Reports, 32(8), 1299-1308. [ Links ]
Lutz, K.A., Knapp, J.E., & Pal, M. (2001). Expression of bar in the Plastid Genome Confers Herbicide Resistance. Plant Physiology, 125(4), 1585-1590. [ Links ]
Maiti, I., & Shepherd, R. J. (1999). US Patent No. US 5994521. [ Links ]
Maiti, I.B., Gowda, S., Kiernan, J., Ghosh, S.K., & Shepherd, R.J. (1997). Promoter/leader deletion analysis and plant expression vectors with the figwort mosaic virus (FMV) full length transcript (FLt) promoter containing single or double enhancer domains. Transgénic Research, 6(2), 143-156. [ Links ]
Nakamura, Y., Gojobori, T., & Ikemura, T. (2000). Codon usage tabulated from international DNA sequence databases: status for the year 2000. Nucleic Acids Research, 28(1), 292. [ Links ]
Odell, J.T., Nagy, F., & Chua, N.H. (1985). Identification of DNA sequences required for activity of the cauliflower mosaic virus 35S promoter. Nature, 313(6005), 810-812. [ Links ]
Paz, M., Martinez, J., Kalvig, A., Fonger, T., & Wang, K. (2006). Improved cotyledonary node method using an alternative explant derived from mature seed for efficient Agrobacterium-mediated soybean transformation. Plant Cell Reports, 25(3), 206-213. [ Links ]
Paz, M., Shou, H., Guo, Z., Zhang, Z., Banerjee, A., & Wang, K. (2004). Assessment of conditions affecting Agrobacterium-mediated soybean transformation using the cotyledonary node explant. Euphytica, 136(2), 167-179. [ Links ]
Perlak, F.J., Fuchs, R.L., Dean, D.A., McPherson, S.L., & Fischhoff, D.A. (1991). Modification of the coding sequence enhances plant expression of insect control protein genes. Proceedings of the National Academy of Sciences, 88(8), 3324-3328. [ Links ]
Rogers, S. (1995). US Patent No. US5378619. [ Links ]
Sanger, M., Daubert, S., & Goodman, R.M. (1990). Characteristics of a strong promoter from figwort mosaic virus: comparison with the analogous 35S promoter from cauliflower mosaic virus and the regulated mannopine synthase promoter. Plant Molecular Biology, 14(3), 433-443. [ Links ]
Sharp, P.M., & Li, W.H. (1987). The codon Adaptation Index-a measure of directional synonymous codon usage bias, and its potential applications. Nucleic Acids Research, 15(3), 1281-1295. [ Links ]
Thompson, C.J., Movva, N.R., Tizard, R., Crameri, R., Davies, J.E., Lauwereys, M., & Botterman J. (1987). Characterization of the herbicide-resistance gene bar from Streptomyces hygroscopicus. EMBO Journal, 6(9), 2519-2523. [ Links ]
Villalobos, A., Ness, J., Gustafsson, C., Minshull, J., & Govindarajan, S. (2006). Gene Designer: a synthetic biology tool for constructing artificial DNA segments. Biomed Central Bioinformatics, 7, 1-8. [ Links ]
Wohlleben, W., Arnold, W., Broer, I., Hillemann, D., Strauch, E., & Punier, A. (1988). Nucleotide sequence of the phosphinothricin N-acetyltransferase gene from Streptomyces viridochromogenes Tü494 and its expression in Nicotiana tabacum. Gene, 70(1), 25-37. [ Links ]
Wright, F. (1990). The 'effective number of codons' used in a gene. Gene, 87(1), 23-29. [ Links ]
Yan, H-Q., Chang, S-H., Tian, Z-X., Zhang, L., Sun, Y-C., Li, Y., Wang, J., & Wang, Y-P. (2011). Novel AroA from Pseudomonas putida confers tobacco plant with high tolerance to glyphosate. Plos One, 6(5), 1-7. [ Links ]
Zeng, P., Vadnais, D.A., Zhang, Z., Polacco, J.C. (2004). Refined glufosinate selection in in Agrobacterium-mediated transformation of soybean [Glycine max (L.) Merrill]. Plant Cell Reports, 22(7), 478-482. [ Links ]
Zhang, Z., Xing, A., Staswick, P., & Clemente, T.E. (1999). The use of glufosinate as a selective agent in Agrobacterium-mediated transformation of soybean. Plant Cell, Tissue and Organ Culture, 56(1), 37-46. [ Links ]