1. Introducción
El componente biológico del suelo es de vital importancia cuando se habla del potencial agrícola de una zona de producción ya que, gracias a esta riqueza, tanto taxonómica como funcional, suceden procesos biológicos importantes como el ciclaje de nutrientes, siendo este uno de los servicios ecosistémicos que permiten el soporte, aprovisionamiento y regulación ambiental (Aguilar-Paredes, et al., 2023). Particularmente el ciclo del nitrógeno es indispensable para el mantenimiento de la vida en la tierra, las plantas dependen de este proceso para adquirir nutrientes esenciales para su desarrollo. El nitrógeno es necesario para todos los organismos vivos, es el cuarto elemento más abundante en la biomasa celular, representando del 1 al 4% de las células vivas formando parte esencial de aminoácidos y ácidos nucleicos. En las plantas un déficit de nitrógeno afecta el crecimiento y desarrollo, mientras que un exceso puede ser perjudicial para animales, seres humanos y el medio ambiente (Pashaei et al., 2022; Aczel, 2019).
El ciclo biogeoquímico del nitrógeno se atribuye a seis procesos de transformación: asimilación, amonificación, nitrificación, desnitrificación, oxidación anaeróbica del amonio (Anammox) y fijación de nitrógeno (Kuypers et al., 2018). En esta investigación nos centramos en el proceso de nitrificación, el cual consiste en un proceso aeróbico de dos pasos; el primer paso corresponde a la oxidación del amonio a nitrito (NH3 → NO2-), y posteriormente, el nitrito se oxida a nitrato (NO2- → NO3-), siendo esta una de las formas asimilables por las plantas (Andrade et al., 2015). Hasta hace poco este proceso se atribuía principalmente a bacterias oxidantes del amonio (AOB) de los géneros: Nitrosomonas (β-proteobacteria), Nitrosospira (β-proteobacteria), Nitrospira (β-proteobacteria) y Nitrosococcus (γ-proteobacteria), sin embargo, a través de técnicas independientes de cultivo como la metagenómica y la metatranscriptómica, se ha identificado una gran abundancia de comunidades de archaeas oxidantes de amonio (AOA), en muchos casos superando en número a las AOB en diferentes ambientes (Cerón et al., 2012; Leininger et al., 2006), descubrimiento que evidenció la complejidad de estas comunidades en el medio que habiten (Hatzenbichler, 2012).
Las AOA también catabolizan el primer paso del ciclo de nitrificación. Si bien en medios marinos de aguas superficiales, zonas costeras y mar abierto, se presenta la mayor riqueza descrita de linajes de AOA, donde pueden representar del 20% al 30% de la población de microorganismos (Karner et al., 2001; Herndl et al., 2005), no son exclusivos de estas zonas, ya que como describe Tourna et al. (2011), microorganismos como Nitrososphaera viennensis EN 76, son originarios del suelo y pueden ser cultivados a un pH inferior a 5.5, condición predominante en el 30% de la superficie terrestre libre de hielo y asociada a altas tasas de nitrificación (Lehtovirta-Morley et al., 2011).
Durante el proceso de nitrificación las AOB y AOA activan el amonio oxidándose a hidroxilamina, reacción catalizada por la enzima amonio monooxigenasa (AMO) y codificada por el gen amo-A (Kuypers et al., 2018), dicho gen se utiliza como marcador molecular en el análisis de la identificación de la presencia y abundancia de microorganismos a través de técnicas de qPCR (Andrade et al., 2015). La enzima AMO pertenece a una familia diversa de monooxigenasas asociadas a membranas que contienen cobre (CuMMO) (Tavormina et al., 2011).
Si bien durante mucho tiempo este tipo de estudios en las comunidades de AOB y AOA fueron escasos debido a las limitaciones que presentan las técnicas de cultivo tradicional, para el estudio de comunidades dentro de los ecosistemas terrestres, el desarrollo de la transcriptómica, abrió un mundo de posibilidades ya que, el análisis se realiza por medio de moléculas de RNA que se copian a cDNA y de este modo se puede evaluar indirectamente la producción de proteínas en un medio, esto con ayuda de técnicas moleculares como PCR en tiempo real (qPCR), PCR con transcriptasa reversa (RT-PCR) y PCR digital (dPCR) (Colin, 2000). Estas técnicas aportan información clave para entender cómo la información del genoma se traduce en funciones celulares y como la traducción responde a diferentes condiciones en el entorno (Fang et al., 2012).
Como parte de esta investigación se realizó un análisis en muestras de suelo extraídas de un cultivo de arroz (Oryza sativa) ubicado en el departamento del Meta (Villavicencio). Este cultivo es el tercer cereal con mayor producción en el mundo, el cual ascendió a un total de producción para el año 2020 de 756 Mt y cuyo mayor productor es el continente asiático (90.5%) (FAOSTAT, 2020). Para el caso colombiano, en el año 2021 se alcanzó una producción de 3.3 Mt, siendo la zona Llanos orientales la de mayor aporte, seguido de la zona Centro (Fedearroz, 2021a), logrando así que las importaciones de arroz se disminuyeran en un 94%, generando una gran oportunidad para incentivar a los productores en la siembra de este cereal, sin dejar de lado las buenas prácticas agrícolas y técnicas de agricultura de precisión que se vienen incentivando a través de programas como “Adopción Masiva de Tecnología - AMTEC” promovidos por la Federación Nacional de Arroceros (Fedearroz), enfocado en generar mejores resultados de producción con un menor costo, teniendo en cuenta las necesidades edafoclimáticas de cada zona (Fedearroz, 2021b)
Estos programas plantean el sistema de agricultura por ambientes en donde los suelos destinados a la producción agrícola son entendidos como unidades heterogéneas, tanto espacial como temporalmente (Tiki et al., 2015). Dicho sistema consiste en la toma de decisiones y la asignación de recursos de forma eficiente a partir del análisis integral de las características edáficas (biológicas, físicas y químicas) presentes dentro del lote, siendo esta una herramienta para la optimización de procesos productivos, la diferenciación de labores de manejo agronómico y la conservación de la diversidad microbiológica del suelo (Scudiero et al., 2018).
De este modo se espera que, la información generada con el uso de técnicas moleculares como la transcriptómica, en combinación con la caracterización por ambientes en suelos arroceros del Meta, contribuya a generar planes de manejo integrado diferenciados que mejoren la productividad y eficiencia del cultivo de arroz en Colombia. Por tal razón, el objetivo de esta investigación fue realizar la cuantificación de los transcritos del gen amo-A en poblaciones de AOB y AOA presentes en un lote arrocero caracterizado por ambientes, empleando técnicas de PCR cuantitativa.
2. Materiales y métodos
2.1 Área de estudio y recolección de muestras de suelo
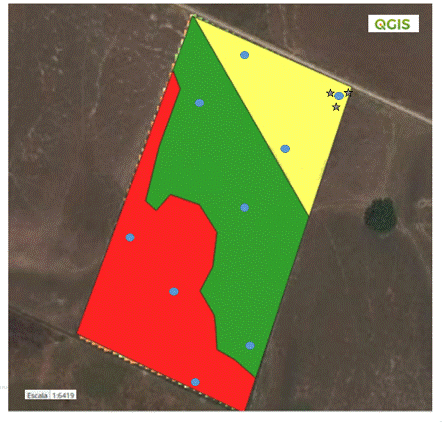
En total se recolectaron 18 muestras de suelo (9 de suelo de soporte y 9 de suelo rizosférico) de un lote de arroz (Oryza sativa) de la finca “La Arabia” ubicada en la vereda “Las delicias”, en el municipio de Cabuyaro del departamento del Meta - Colombia (Coordenadas 4° 22’ 43.17” Norte, 72° 58’ 26.14” Oeste). El área total de estudio correspondió a un lote comercial arrocero de 33 ha con sistema de riego y manejo de fertilización tradicional. Esta área se delimitó en tres ambientes diferentes (alto, medio y bajo) teniendo en cuenta el rendimiento de las plantas de arroz y un análisis de imágenes espectrales de un histórico de 10 años. En cada ambiente se definieron al azar 3 puntos de muestreo y en cada punto se tomó una muestra compuesta del suelo de soporte y una de la capa rizosférica. Estas muestras se tomaron ochos días después del último suministro de fertilización en el ciclo del cultivo (etapa de floración) (S1), para la toma de muestras se utilizaron implementos y herramientas libres de RNAsa para garantizar la integridad del ácido ribonucléico (RNA), la recolección de muestras del suelo de soporte se realizó una limpieza superficial de la zona y con ayuda de un barreno se extrajo una muestra a una profundidad no mayor de 15 cm. Las muestras suelo rizosférico se tomaron a tres plantas de arroz por punto, de las cuales se recolectó el suelo adherido a la zona radical. Las muestras se almacenaron en hielo seco para ser transportadas y luego se almacenaron a -80°C en laboratorio para su posterior análisis molecular.
2.2. Cuantificación de lecturas de DNA con homología al gen amo-A de bacterias y archaeas
Con el fin de asegurar la presencia de las comunidades de AOB y AOA en las muestras de suelo del lote arrocero, se realizó la exploración de datos metagenómicos obtenidos a partir de las mismas muestras, empleando la plataforma KBase (Arkin et al, 2018) donde se realizó la alineación entre librerías metagenómicas del gen amo-A frente a secuencias reportadas en el GenBank para cada comunidad a través del programa Bowtie2 v2.5.1 (Langmead & Salzberg, 2012), junto con la secuenciación previamente realizada (datos no mostrados). Finalmente se obtuvo el número de bases de nucleótidos y genes asociados con el gen de interés para cada comunidad.
2.3. Cuantificación de la expresión del gen amo-A de bacterias y archaeas por RT-qPCR y RT-dPCR
Una vez se confirmó la presencia de las comunidades AOB y AOA, se procedió a la cuantificación del gen amo-A utilizando las técnicas de RT-qPCR y RT-dPCR. Las dos técnicas son de cuantificación absoluta, sin embargo, la RT-qPCR requiere del desarrollo de curvas estándar.
2.3.1. Selección y diseño de cebadores de amplificación
Inicialmente se hizo una revisión bibliográfica y se seleccionaron dos conjuntos de cebadores que flanquean la secuencia del gen amo-A de las comunidades de AOB y AOA, en donde se seleccionaron los cebadores específicos reportados por Rotthauwe et al. (1997) para AOB y Francis et al. (2005) para AOA (S2).
Adicionalmente, para la comunidad de AOB se diseñó un par de cebadores que se utilizaron en una PCR anidada con el fin de incrementar la sensibilidad y detección de la técnica, donde inicialmente se amplificó una región más grande y luego se utilizaron los cebadores específicos de Rotthauwe et al. (1997). El diseño y termodinámica de estos cebadores se realizó a través de la plataforma “Integrated DNA Technologies Inc.” utilizando secuencias genómicas de especies bacterianas oxidantes de amonio presentes en el GenBanK.
2.3.2. Extracción de Ácido ribonucleico (RNA) de las muestras de suelo y purificación
El RNA se extrajo a partir de 2 g de suelo utilizando el Kit de RNA PowerSoil® de Mo Bio, Laboratories, Inc. (12866-25), de acuerdo con el protocolo establecido por el fabricante donde se utilizó fenol:cloroformo:alcohol isoamílico para la desnaturalización de proteínas, eliminación de lípidos y la separación de la fase acuosa y orgánica. Los rendimientos del procedimiento de extracción de RNA fueron determinados por fluorometría usando el kit QubitTM RNA HS Assay (InvitrogenTM) y el equipo QubitTM.
Una vez cuantificado el RNA se procedió a la purificación de este, donde se realizó un tratamiento con la enzima DNAsa I® (Invitrogen®), utilizando 8.6 μl de RNA y adicionando 0.4 μl de la enzima DNAsa (1 U/μl) y 1 μl de Buffer de reacción (10x), se incubó durante 10 min a 37 ºC; luego se adicionó 1 μl de EDTA (25 mM) y finalmente se inactivó la enzima llevando la solución a 65°C durante 10 min.
2.3.3. Síntesis de cDNA
La síntesis de cDNA a partir de RNA extraído de las muestras de suelo se realizó mediante el kit Transcriptor First Strand cDNA Synthesis (Roche®) para un volumen final de 20 μl. Para la etapa de anillamiento de oligonucleótidos se agregó al RNA, tratado con DNAsa, 2 μl de cebadores de hexámeros aleatorios (10 μM) y 1 μl de agua grado PCR, para después hibridar a 65 °C durante 10 min y luego se transfirió inmediatamente a hielo. Para la síntesis de la primera cadena se adicionaron a la solución previa 4 μl de Buffer (5x), 2 μl del mix de desoxinucleótidos (10 mM), 0.5 μl del Protector Inhibidor de RNAasa (40 U/μl) y 0.5 μl de Transcriptasa Reversa (20 U/μl), se incubó a 55 °C durante 30 min y posteriormente se inactivó a 85 °C durante 5 min, finalmente se transfirió inmediatamente a hielo por 1 min.
Una vez terminado este proceso se procedió a cuantificar los cDNAs por fluorometría usando el kit Quant-iTTM ssDNA Assay (Invitrogen®) con el equipo QubitTM. Posterior a este análisis se almacenaron las muestras a -80 ºC hasta el momento de la amplificación por qPCR.
2.3.4. Curvas estándar
Para realizar las curvas de cuantificación se emplearon dos estrategias, la primera consistió en la amplificación de una muestra de DNA de suelo y el segundo a través de un plásmido.
Método I: Amplificación muestra de DNA de suelo (Templete).
Se realizó la extracción de DNA de una muestra de suelo de jardín y luego se llevó a cabo una amplificación con PCR convencional utilizando los cebadores reportados por Francis, et al. (2005) para AOA y para AOB los cebadores diseñados en esta investigación (Tabla 1), donde se utilizó el termociclador T100TM Thermal Cycler (Bio-Rad). La mezcla de reacción para AOA y AOB (12.5 μl) contenía 2.5 μl del GoTaq® Flexi Buffer (5x), 4.7 μl de agua grado PCR, 0.13 μl de cada primer (20 mM), 0.125 μl de dNTPs (20 mM), 0.5 μl de solución de MgCl2 (25 mM), 2.2 μl de albúmina de suero bovino - BSA (20 mg/μl), 0.2 μl de GoTaq® DNA Polimerasa (5 U/μl) y 2 μl de DNA (134 ng/μl). El ciclo térmico para AOB fue el siguiente: 95 ºC durante 10 min y 40 ciclos (94 ºC, 1 min; 60 ºC, 1 min y 72 ºC, 1 min) y un ciclo final de 72°C por 7 min. El ciclo térmico para AOA fue el siguiente: 95 ºC durante 10 min y 40 ciclos (95 ºC, 45 seg; 55 ºC, 45 seg y 72 ºC, 15 seg) y un ciclo final de 79°C por 10 min. El tamaño de los amplímeros se confirmó en gel de agarosa al 1.5%. Luego se cuantificaron las concentraciones de los fragmentos del DNA amplificado por fluorometría, usando el kit QubitTM dsDNA BR Assay (Invitrogen) con el equipo QubitTM. Finalmente se determinó el número de copias utilizando la concentración en ng de la PCR convencional y el tamaño del fragmento flanqueado a través de la herramienta “Calculator for determining the number of copies of a template” de URI Genomics & Sequencing Center.
Luego se realizó una PCR en tiempo real para AOB y AOA (10 μl), respectivamente, utilizando diluciones seriadas en base diez, 109, 107, 105, 103 y 101 de cada uno de los amplimeros de la PCR convencional, con la siguiente mezcla de reacción: 5 μl del Mix DNA Green Master (2x), 2.1 μl de albúmina de suero bovino - BSA (20 mg/μl), 0.105 μl de cada primer (20 mM), 0.7 μl de agua grado PCR y 2 μl de cada dilución de amplimero. El ciclo térmico fue el mismo empleado para la PCR convencional ajustado de acuerdo con las recomendaciones del equipo qTower3G para SYBR GREEN.
Método II: Plásmido.
De curvas estándar se generaron utilizando un plásmido (TOPO TA), donde se insertó al plásmido los amplímeros que se obtuvieron previamente mediante PCR convencional con los cebadores reportados por Rotthauwe, et al. (1997) y Francis, et al. (2005). Igualmente, se determinó el número de copias y se realizaron diluciones seriadas en base 10. Finalmente se utilizaron las mismas condiciones de mezcla y ciclos térmicos utilizados para hacer las curvas estándar de la estrategia de templete.
2.3.5. Cuantificación de la expresión del gen amo-A por PCR en tiempo real (RT-qPCR)
El número de transcritos del gen amo-A de AOB y AOA se determinó mediante PCR en tiempo real utilizando el Kit FastStart Essential DNA Green Master (Roche) con el tinte fluorescente SYBR-Green y termociclador qTower3G (Analytik Jena). Todas las muestras de cDNA fueron ajustadas a una misma concentración colocando 12 ng para cada reacción de 10 μl. La mezcla de reacción para AOB y AOA (10 μl) contenía 5 μl del Mix DNA Green Master (2x), 2.1 μl de albúmina de suero bovino - BSA (20 μg/μl), 0.105 μl de cada primer (20 mM) y 0.7 μl de agua grado PCR. Cada muestra se analizó por triplicado empleando las condiciones de termociclaje de la PCR convencional.
2.3.6. Cuantificación de la expresión del gen amo-A por PCR digital (RT-dPCR)
El número de transcritos del gen amo-A de AOB y AOA también se determinó mediante PCR digital utilizando el Kit QIAcuity EG PCR (QIAGEN) con el tinte fluorescente EvaGreen en el termociclador QIAcuity One (QIAGEN).
La mezcla de reacción para AOB y AOA (12 μl) contenía 4 μl del Mastermix QIAcuity EvaGreen PCR (3x), 0.24 μl de cada primer (20 mM), 5.5 μl de agua grado PCR y 2 μl de cDNA (12 ng). El ciclo térmico empleado correspondió al recomendado por el kit QIAcuity EG PCR utilizando las temperaturas de anillamiento de 60 °C y 55 °C para AOB y AOA, respectivamente. Cada muestra se analizó con una sola repetición, pero cabe destacar que en esta técnica cada muestra se particionó en 8500 nanoceldas y en cada una se generó una reacción de amplificación del gen de interés.
2.4. Análisis estadístico
En el análisis metagenómico se emplearon pruebas paramétricas (ANOVA), donde se revisaron los supuestos de normalidad, homogeneidad de las varianzas e independencia, con un posterior análisis post-hoc HSD de Tukey para ver si hubo diferencias entre los ambientes, el tipo de muestra de suelo y la comunidad de microorganismos. Mientras que para el análisis del número de transcritos se hizo por medio de pruebas no paramétricas (Kruskal-Wallis). Por otra parte, se empleó el análisis por componentes principales (PCA) para identificar las asociaciones entre las propiedades químicas y el número de transcritos del gen amo-A. El programa que se utilizó fue R Studio (R Core Team, 2022).
3. Resultados y discusión
El proceso de nitrificación es de gran importancia en la agricultura ya que por medio de este las plantas tienen acceso al N, esta transformación es posible gracias al papel funcional de los microorganismos presentes en el suelo, sin embargo, las prácticas agrícolas y la alteración del hábitat de estos organismos pueden ocasionar una expresión variable según las condiciones del medio y el cultivar presente (Barrios, 2007).
3.3. Números de bases y genes asociados al gen amo-A de bacterias y archaeas
No se encontraron diferencias en el número de bases de nucleótidos (B) del gen amo-A en las muestras de suelo de soporte y rizosférico en AOB de los ambientes (alto, medio y bajo). Sin embargo, sí hubo diferencias entre el número de B según el tipo de muestra, siendo mayor en las muestras de suelo rizosférico (8882 B) respecto al de soporte (4975 B) (Tabla 1). Por otra parte, los genes asociados presentaron el mismo comportamiento de la variable B, pues se encontró en el suelo rizosférico un total de 20 genes asociados respecto al de soporte que presentó 12 genes asociados (Tabla 1). En un estudio realizado por Wu et al. (2022) evaluaron la diversidad de abundancia de AOB en suelos rizosféricos de un cultivo de arroz, donde encontraron una amplia diversidad de bacterias presentes en la zona radical que contribuyen a diferentes procesos en la planta como la toma de nutrientes o el sistema de defensa, esta relación simbiótica entre los microorganismos y las raíces es importante para el crecimiento y desarrollo del cultivo, influyendo en la calidad y en el rendimiento del mismo. Se resalta que las comunidades que contribuyen al ciclo del nitrógeno pueden variar de acuerdo a diferentes condiciones del medio como tipo del suelo, el estado de desarrollo de la planta, las interacciones con otros microorganismos y el suministro exógeno de N al suelo (Kuypers et al., 2018).
Tabla 1 Recuento de bases (B) y genes asociados al gen amo-A de AOB y AOA a partir de datos metagenómicos. Los valores corresponden a los promedios de las sumatorias de las B y el recuento de genes asociados de todas las muestras en cada ambiente (error estándar). El promedio y letras diferentes del promedio en las tablas hace referencia a la comparativa entre comunidades, mientras que, en la misma columna, letras diferentes indican diferencias entre cada tipo de suelo o ambiente (HSD de Tukey, p<0,05).
Para el caso de AOA no se encontraron diferencias estadísticas respecto al número total de (B) y genes asociados a la subunidad enzimática encargada de síntesis de la amo-A en los distintos ambientes, ni tampoco entre las muestras de suelo de soporte o rizosférico, esto pone en evidencia una estructura poblacional con poca varianza, tanto en la muestra compuesta de suelo de soporte como en la muestra del suelo rizosférico, si bien como se observa en la tabla 1, en la muestra de suelo de soporte el ambiente clasificado como bajo, muestra un número elevado de B (11047 B), este valor se debe a un dato atípico, el punto donde se realizó el muestreo pertenecía a una zona que presentaba un nivel de humedad bajo respecto a los otros puntos por características topográficas, lo que pudo favorecer un microclima diferencial en esta zona. Estudios previos han demostrado que la producción de N2O subproducto de la oxidación desde la NH2OH a NO del proceso de nitrificación (Schreiber et al., 2012), se ve afectado en cultivos de arroz paddy durante el ciclo de secano y durante la inundación del cultivo (Xing et al., 2009) lo que podría suponer un cambio de la estructura poblacional de AOA durante cada una de las etapas del manejo del cultivo, esto podría influenciar directamente el número de genes asociados en el ambiente catalogado como bajo, pues en este punto se muestra un mayor número (33), lo que directamente estaría mostrando una mayor diversidad poblacional respecto a los demás puntos del lote tal como se observa en la (Figura 1).

Figura 1 Heatmap de bases de nucleótidos del gen amo-A de poblaciones de AOA y AOB presentes en un lote arrocero.
Respecto al comportamiento entre especies, se encontró que en la variable de número de B no hubo diferencias significativas en las muestras de suelo de soporte, no obstante, en las muestras de suelo rizosférico las AOB (8882) son mayores comparadas con las AOA (3846). Por otra parte, en la variable de número de genes asociados se encontraron diferencias en las muestras de suelo de soporte, donde las AOA (23) aumentaron respecto a las AOB (12) y el mismo comportamiento se observó en las muestras de suelo rizosférico, donde las AOB (20) fueron mayor respecto a las AOA (12) (Tabla 1 y Figura 1).
Este comportamiento en el suelo de soporte en donde se observó mayor diversidad poblacional de arqueas respecto a las bacterias es el esperado, ya que como estableció Leininger, (2006) las AOA son más abundantes en suelos que su contraparte las AOB, esto debido al amplio rango de adaptabilidad de estos organismos en el suelo (Ramirez, 2006) comparado con las AOB. Mientras que el comportamiento observado en el suelo rizosférico en donde se encontró favorecida la diversidad de AOB respecto a las AOA obedece a la interacción planta-organismo en donde la producción de exudados puede favorecer el establecimiento de comunidades AOB (Azziz et al., 2016).
3.4. Abundancia de transcritos de poblaciones oxidantes de amonio
La cuantificación de la expresión del gen amo-A se realizó tomando como punto de partida el RNA presente en las muestras de suelo para luego ser sintetizado en DNA complementario (cDNA) y finalmente realizar la amplificación por PCR. En esta investigación la cuantificación se realizó con dos técnicas de PCR, la primera PCR cuantitativa (RT-qPCR) donde se calculó el número de copias del gen a partir de curvas de calibración (templete y plásmido), pero al no observar resultados confiables para las AOB, se realizó una segunda cuantificación a través de una PCR digital (RT-dPCR). A continuación, se discuten los resultados obtenidos para cada técnica empleada.
3.4.3. Técnica PCR en tiempo real (RT-qPCR)
Para el análisis de AOB como para AOA se emplearon dos tipos de curvas de calibración (Figuras 2 y 3), la primera correspondió a un fragmento de DNA, utilizado como templete, donde el rango de lectura del número de copias del gen amo-A oscilaba entre los 7300 y los 7.3 x 109, con una eficiencia en la curva de 1.02 y un R2 0.9874 (Figura 2A). En esta curva el número de transcritos amplificados de las muestras de suelo del lote arrocero se encontraron en el rango de 6 a 39 número de copias (Figura 2C), valores que se localizaron fuera del rango de confianza de lectura de la curva, por lo cual, no fue concluyente debido a que esta cuantificación puede deberse a la formación de dímeros a partir de los cebadores. En cuanto a la curva realizada por plásmido (Figura 2B), el rango de lectura se encontró entre 1310 a 1.31 x 109 número de copias del gen amo-A con una eficiencia de 100% y R2 0.97966, sin embargo, la amplificación de las muestras de suelo del lote arrocero estuvo entre 19 a 103 número de copias (Figura 2C), reflejando la misma situación de la curva de calibración del templete. Por esta razón se determinó que la técnica no fue confiable para la evaluación de este gen en las muestras de suelo de esta investigación, por lo cual, se procedió a la amplificación del gen amo-A empleando la técnica de PCR digital para dilucidar los resultados obtenidos.
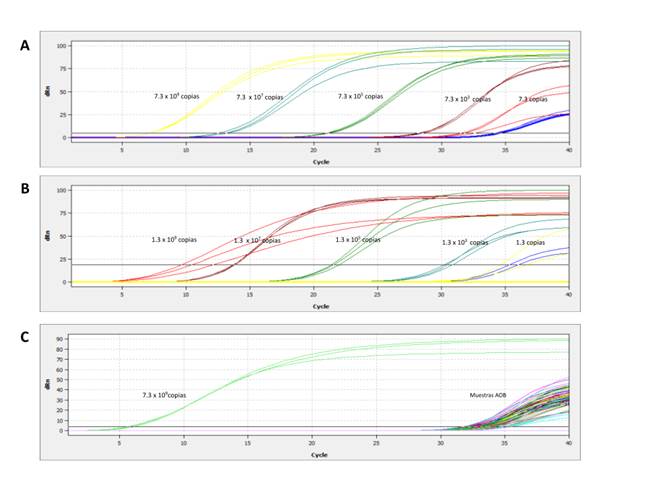
Figura 2 A. Curva estándar de amplificación del número de copias del gen amo-A de AOB por RT-qPCR, usando como templete diluciones seriadas de un producto de PCR de 911 pb. B. Curva estándar de amplificación del número de copias del gen amo-A de AOB por RT-qPCR, usando como templete diluciones seriadas del plásmido TOPO A-AOB. C. Amplificación del número de transcritos del gen amo-A en la comunidad de AOB presente en los suelos de los tres ambientes por el método de RT-qPCR.
Se observa en la tabla 2 que el número de copias de las AOA, no presentó diferencias significativas entre ambientes ni entre tipo de suelo, sin importar la curva estándar utilizada (plásmido o fragmento). Las comunidades AOA de esta investigación son poco variables en el medio sin importar el tipo de suelo en donde se encuentran (soporte o rizosférico), o el tipo de ambiente, gracias a esto se pudo establecer que ninguna de estas variables influenció la expresión del gen.
Ambas variantes de la qPCR mostraron una buena capacidad de lectura para establecer el número de copias del gen, para el caso de la qPCR con la curva de calibración obtenida por la dilución del fragmento o templete, estableció un rango de lectura de 6.76 x 109 a 67 copias con una eficiencia de 0.91% y R2 0.98368, por su parte, el método del plásmido mostró mejor sensibilidad al momento de realizar la amplificación, presentando un rango de lectura más amplio, de 7.3 x 109 hasta 7.3 copias con una eficiencia de 0.98% y R2 0.98848. No se presentaron diferencias estadísticas entre las distintas estrategias empleadas para las curvas estándar, dando resultados similares para ambas por lo cual se podrían tener en consideración para realizar cuantificación del gen amo-A en AOA.

Figura 3 A. Curva estándar de amplificación del número de copias del gen amo-A de AOA por RT-qPCR, usando como templete diluciones seriadas de un producto de PCR de 639 pb. B. Curva estándar de amplificación del número de copias del gen amo-A de AOA por RT-qPCR, usando como templete diluciones seriadas del plásmido TOPO A-AOA. C. Amplificación del número de transcritos del gen amo-A en la comunidad de AOA presente en los suelos de los tres ambientes por el método de RT-qPCR.
Tabla 2 Número de transcritos del gen amo-A de AOB y AOA según el ambiente, el tipo de suelo y la técnica empleada en la amplificación de RT-qPCR. Los valores corresponden a los promedios de todas las muestras (error estándar). En la misma columna, letras diferentes indican diferencias (HSD de Tukey, p<0.05).
| AOB | AOA | ||||
|---|---|---|---|---|---|
| Ambiente | Fragmento de PCR | Plásmido | Fragmento de PCR | Plásmido | |
| Soporte | Alto | - | - | 749.76 (88.2) a | 581.53 (60.1) a |
| Medio | - | - | 654.10 (111.6) a | 506.27 (76.7) a | |
| Bajo | - | - | 585.84 (91.8) a | 450.49 (62.6) a | |
| Rizosférico | Alto | - | - | 676.65 (53.8) a | 520.31 (43.4) a |
| Medio | - | - | 939.83 (237.6) a | 758.18 (197.5) a | |
| Bajo | - | - | 1440.71 (472.3) a | 1199.8 (409.6) a | |
3.4.4. Técnica dPCR
El número de transcritos del gen amo-A en comunidades de AOB se encuentra en la tabla 3. Allí se observa que no hubo diferencias entre los ambientes ni el tipo de suelo. Los valores con esta técnica oscilaron entre 1 y 2 números de copias (Figura 4A), los cuales se consideraron negativos. En conclusión, se encontró que no hay expresión de este gen por parte de la comunidad de AOB en las muestras de suelo del lote arrocero bajo las concentraciones y técnicas empleadas en esta investigación. Este resultado puede estar asociado con el tipo de fertilización que se ha realizado en el lote en los últimos años, según los reportes de manejo se han suministrado fertilizantes químicos para la compensación de nutrientes, ciclo tras ciclo. Wang et al. (2022), encontró que en un lote de maíz al suministrar constantemente dosis de N (Urea) al suelo disminuyó notablemente la expresión del gen amo-A en las comunidades de AOB, inhibiendo el proceso de nitrificación.

Figura 4 A. Número de transcritos del gen amo-A en la comunidad de AOB presentes en las muestras de suelo de los tres ambientes por el método de RT-dPCR. Celdas analizadas. Diluciones de ADN de suelo seriadas: A1, B1; ADN copia de muestras de suelo provenientes de los tres ambientes A2, A3 B2, B3, C1, C2, C3, D1, D2, D3, E1, E2, E3, F1, F2, F3, G1, G2; ADN de suelo de jardín como control positivo: G3; Control negativo: H3. Y B. Número de copias del gen amo-A en la comunidad de AOA presentes en las muestras de suelo de los tres ambientes por el método de dPCR. Celdas analizadas. Diluciones de ADN de suelo seriadas: A1, B1; ADN copia de muestras de suelo provenientes de los tres ambientes A2, A3 B2, B3, C1, C2, C3, D1, D2, D3, E1, E2, E3, F1, F2, F3, G1, G2; ADN de suelo de jardín como control positivo: G3; Control negativo: H3.
En las AOA no se observó diferencias significativas entre ambientes o tipo de suelo tal como se estableció anteriormente, si bien, como se ve en la Tabla 4 las muestras del ambiente medio mostraron un valor más alto respecto a los otros dos ambientes (Figura 4B) esto se debe a un dato atípico en los datos de análisis ocasionada por la topografía del terreno.
El número de copias del gen amo-A se vio reducido frente al número de copias obtenido por la otra técnica, esto se debe a que la dPCR es una técnica absoluta que no depende de una calibración externa de una curva (Lewintre, 2021), por lo cual no se tiene sobreestimaciones frente a la cuantificación, mientras que para el caso de la RT-qPCR se presenta las limitaciones que se verán más adelante, generando estas sobreestimaciones.
En la tabla 3 se encuentra la comparación entre las dos comunidades (AOA y AOB) donde se observó que solo se obtuvo número de transcritos en la comunidad de AOA, tanto para muestras de suelo de soporte como rizosférico. Esta técnica de RT-dPCR fue concluyente con el resultado de bacterias, donde se encontró que no hay expresión de este gen en las muestras de suelo del lote arrocero bajo las concentraciones y técnicas empleadas en este trabajo, descartando la incertidumbre de la técnica que generó la PCR en tiempo real.
Tabla 3 Número de transcritos del gen amo-A de bacterias y archaeas según el ambiente y el tipo de suelo. Los valores corresponden a los promedios de todas las muestras (error estándar). En la misma columna, letras diferentes indican diferencias estadísticamente significativas (HSD de Tukey, p<0.05).
| Ambiente | AOB | AOA | |
|---|---|---|---|
| Soporte | Alto | 0.0 (0.0) a | 73.64 (6.8) a |
| Medio | 0.4 (0.2) a | 189.17 (78.8) a | |
| Bajo | 0.5 (0.3) a | 51.88 (0.6) a | |
| Rizosférico | Alto | 0.7 (0.4) a | 41.80 (2.9) a |
| Medio | 0.4 (0.2) a | 299.88 (120) a | |
| Bajo | 0.0 (0.0) a | 47.06 (4.1) a |
Tabla 4 Comparación de número de transcritos entre técnicas para la comunidad de AOA
| RT-qPCR con templete | RT-qPCR con Plásmido | RT-dPCR |
|---|---|---|
| 685.7 (195) a | 504.38 (79.4) ab | 53 (3.2) b |
La comparación entre técnicas de cuantificación se hizo exclusivamente para las AOA debido a que estos organismos lograron ser cuantificados por cada una de las técnicas aplicadas en esta investigación. Para este caso como se observa en la tabla 4 se evidenció que no se presentó diferencias estadísticas entre las técnicas empleadas, pero sí se observó diferencias entre las estrategias de RT-qPCR, esto debido a una menor sensibilidad de la estrategia de templete frente a la de plásmido, en donde se obtuvo el grupo a y grupo b, respectivamente. Se observó que el número de copias obtenido a partir de la curva de calibración con el plásmido comparte medias entre ambas técnicas por ende se establece que para este método las diferencias estadísticas no son significativas.
Se parte de la idea que las tres metodologías se basan en cuantificación absoluta, aunque las de RT-qPCR se pueden definir como relativa ya que, son absolutas en términos de cuantificación, pero son relativas respecto a la curva estándar, (Lewintre,2021). En las técnicas realizadas con RT-qPCR se logró obtener el número de copias gracias a las curvas realizadas, estas son altamente reproducibles permitiendo datos específicos y de alta sensibilidad, sin embargo requieren absoluta exactitud pues la cuantificación de la expresión genética en la RT-qPCR depende exclusivamente de la precisión de los estándares empleados (Pfaffl, 2008) mientras que la RT-dPCR se basa en la fracción de reacciones negativas y su recuento, esto quiere decir que no necesita curvas ya que divide la muestra en distintas nanoceldas en reacciones en paralelo, las celdas donde ocurra una reacción emitirá fluorescencia y serán contadas en el recuento arrojando el número absoluto de copias haciéndola más sensible.
Como se observa en la figura 3A la curva estándar del fragmento llego a presentar problemas frente a la estandarización de la dil 5 (Color amarillo) y la dil 7 (Color azul aguamarina), lo que llevo a que estos resultados fueran altos en exactitud, pero con una precisión media, esto permitió la identificación del gen de interés ya que se obtuvo el rango de lectura para la expresión del gen, pero limito la sensibilidad de la RT-qPCR ya que el número de copias pudo variar debido a esto. Por su parte la curva estándar con el plásmido (Figura 3B) presentó una alta exactitud y precisión lo que ayudó a la sensibilidad de la prueba arrojando valores de cuantificación más precisos y exactos (Figura 3C).
Es de importancia establecer las limitaciones que tiene la qPCR en cuanto a la calidad de los datos obtenidos, debido a que se requiere de un exhaustivo proceso de optimización para garantizar resultados confiables, entre estos procesos se encuentra la especificidad de los primers y el gen diana, la sensibilidad de la prueba la cual detecta la reacción, la eficiencia de la qPCR y por último la reproducibilidad (Edwards, 2004). En donde si bien la mayor parte de las variables se manejaron de la manera adecuada el no realizar un fragmento anidado pudo favorecer la degradación de la secuencia y disminuir la sensibilidad.
3.5. Interacción entre las propiedades químicas del suelo y los transcritos de poblaciones oxidantes de amonio
Para el análisis multivariado se revisaron los resultados de variables químicas como Capacidad de Intercambio Cationico Real (CICA), pH, Conductividad Eléctrica (CE), Materia Orgánica (MO), Carbono Orgánico (CO), Capacidad de Intercambio Catiónico Efectivo (CICE), Acidez (Al+H) y la concentración disponible de Cobre (Cu) para determinar si habían diferencias entre cada ambiente (Tabla 5). En este lote se encontró que hay un nivel bajo del contenido de MO, CICA y CICE, y presenta suelos con un pH de fuerte a extremadamente ácido. Además, solo hay diferencias entre ambientes en las variables de CICE, CICA, MO, CO y Cu, siendo mayor en el lote con rendimiento alto, respecto a los demás.
Tabla 5 Propiedades químicas del suelo para cada ambiente en un lote arrocero. Los valores corresponden a los promedios de todas las muestras (error estándar). En la misma columna, letras diferentes indican diferencias estadísticamente significativas (HSD de Tukey, p<0.05).
| CICA (cmol(+)/kg) | pH | CE (dS/m) | MO (g/100g) | CO (g/100g) | CICE (cmol(+)/kg) | Acidez (cmol(+)/kg) | Cu disponible (mg/kg) | |
|---|---|---|---|---|---|---|---|---|
| Alto | 14.7 (a) | 5.0 (a) | 0.15 (a) | 1.9 (a) | 1.1 (a) | 7.8 (a) | 4.1 (a) | 5.1 (a) |
| Medio | 14.8 (a) | 4.9 (a) | 0.21 (a) | 1.4 (b) | 0.8 (b) | 6.8 (a) | 3.3 (a) | 3.7 (ab) |
| Bajo | 9.3 (b) | 4.8 (a) | 0.16 (a) | 1.6 (ab) | 0.9 (ab) | 4.4 (b) | 2.4 (a) | 1.6 (b) |
El análisis multivariado para la comunidad de AOA (Figura 5) de todos los componentes muestra que el 68% de la variabilidad de los datos es dada por la suma de los 2 primeros vectores, el PC1 el cual es influenciado mayoritariamente por CU y CICE, el PC2, el cual está influenciado por CICA, MO y CO. No se encontró relación entre las variables edáficas evaluadas y la expresión del gen amo-A en arqueas.

Figura 5 Análisis de componentes principales obtenido con los transcritos de AOA y propiedades químicas del suelo evaluado.
Respecto a mayor expresión del gen amo-A en AOA comparado con AOB, Azziz et al. (2016) reporta que el pH del suelo influye en la expresión de las dos comunidades, ya que AOA era 10 veces más abundante que AOB en suelos de arroz, sin embargo, AOB es más abundante en la capa superficial del suelo. También se reporta que las condiciones de pH ácido tienen un efecto positivo en la abundancia de AOA; por lo tanto, el pH puede ser un factor que explique las diferencias en la abundancia de AOA en nuestros resultados. Este pH puede ser afectado por diferentes factores como mayor contenido de N exógeno, calidad del agua del riego, las características del material parental del suelo, variables climáticas, entre otros (Neina, 2019)
CONCLUSIONES
El recuento de número de bases alineadas con el gen amo-A a nivel metagenómico demostró la presencia de arqueas y bacterias en el suelo. Fue mayor la representación de las comunidades bacterianas en el suelo rizosférico, mientras que en el suelo de soporte las arqueas dominaron. No se encontraron diferencias entre arqueas y bacterias en el número de bases asociadas al gen amo-A por ambientes caracterizados.
No se detectó expresión del gen amo-A asociada a la comunidad de bacterias en las muestras de suelo analizadas, con ninguna de las dos técnicas utilizadas en este trabajo (RT-qPCR y RT-dPCR).
El número de transcritos del gen amo-A en la comunidad de arqueas obtenidos por RT-qPCR mediante la estrategia de curvas estándar empleando plásmidos fue más confiable que la cuantificación mediante la estrategia de curvas estándar usando templete de PCR; en esta última, se observó pérdida de sensibilidad y reproducibilidad.
El número de transcritos del gen amo-A en la comunidad de arqueas obtenidos por RT-dPCR fue menor que por RT-qPCR. Al considerar la robustez de la dPCR, se concluyó que estos datos fueron más exactos y se seleccionaron para el análisis estadístico multivariado.
No se detectaron diferencias en la expresión del gen amo-A en la comunidad de arqueas en los ambientes caracterizados (alto, medio, bajo rendimiento), ni entre tipos de muestra de suelo analizados (soporte y rizosférico).
El análisis de componentes principales no mostró asociaciones entre las propiedades químicas del suelo y el número transcritos del gen amo-A de la comunidad de arqueas.