Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista U.D.C.A Actualidad & Divulgación Científica
Print version ISSN 0123-4226
rev.udcaactual.divulg.cient. vol.15 no.2 Bogotá July/Dec. 2012
CIENCIAS AGROPECUARIAS - Artículo Científico
CARACTERIZACIÓN MOLECULAR DE ACCESIONES DE ÑAME (Dioscorea alata L.) DE LA REGIÓN CARIBE COLOMBIANA
MOLECULAR CHARACTERIZATION OF YAM (Dioscorea alata L.) ACCESSIONS FROM THE COLOMBIAN CARIBBEAN REGION
Hernando Rivera-Jiménez1, Andrés Álvarez2, Juan Palacio-Mejía3, Alba Ochoa4
1 Ingeniero Agrónomo, M.Sc. Universidad de Córdoba. Colombia. autor para correspondencia: hriveraj@gmail.com , hernandorivera@sinu.unicordoba.edu.co - Ciudad Universitaria Carrera 6 No. 76-103 Código Postal 354. Montería - Córdoba, Colombia
2 Ingeniero Agrónomo, M.Sc., Universidad de Córdoba. M.Sc. Montería - Córdoba, Colombia. andresalvarez864@hotmail.com
3 Ingeniero Agrónomo, M.Sc. Estudiante de doctorado The University of Texas at Austin, jdpalacio@utexas.edu, EUA
4 Tecnólogo Agrícola. Universidad de Córdoba. Colombia.
Rev. U.D.C.A Act. & Div. Cient. 15(2): 323 - 330, 2012
RESUMEN
El ñame (Dioscorea spp.) hace parte esencial de la dieta de la Costa Norte colombiana, donde es cultivado, llegando, incluso, a ser parte de su identidad cultural. Como estrategia para la conservación, el conocimiento y el uso de esta agrobiodiversidad, la Universidad de Córdoba ha logrado consolidar una colección de germoplasma de ñame, dentro de la que se destacan 14 accesiones de la especie Disocorea alata L., la especie más representativa de la colección. El conocimiento de esta colección comenzó con una caracterización morfológica, que es ahora complementada con la presente caracterización molecular. El objetivo de esta investigación fue evaluar la variabilidad genética de D. alata y cómo se relaciona con la caracterización morfológica, previamente realizada, utilizando para ello los marcadores moleculares AFLP. Los datos fueron analizados mediante los métodos de agrupación de análisis correspondencia múltiple y análisis de similaridad de Dice, estableciendo los niveles de confiabilidad de los grupos genéticos formados mediante remuestreos. La variabilidad genética de las 14 accesiones de ñame fue alta, permitiendo diferenciar cuatro grupos y dos sub-grupos, mediante un patrón de organización estructural bien definido, con una asociación de caracteres moleculares, botánicos y morfológicos, al interior de cada grupo.
Palabras clave: Ñame, germoplasma, variabilidad genética, recursos fitogenéticos, AFLP.
SUMMARY
Yam (Dioscorea Spp) is an essential part of the diet of the Colombian North Coast, where it is cultivated, even becoming part of their cultural identity. As a strategy to enhance the conservation, use and knowledge of its agrobiodiversity, the University of Cordoba has established a germplasm collection of yam, which stores 14 accessions of the species Disocorea alata, the most representative of the collection. The goal of this study was to evaluate the genetic variability of the collection and its relationship to the morphological characterization previously performed, using AFLP molecular markers. The data were analyzed by clustering methods of multiple correspondence analysis and Dice similarity, establishing levels of reliability of genetic groups by resampling. The genetic variability of the 14 yam accessions was high, allowing to-differentiate four groups and two subgroups through a pattern of well-defined organizational structure with a combination of molecular data, botanical and morphological characteristics.
Key words: Yam, germplasm, genetic variability, plant genetic resources, AFLP.
INTRODUCCIÓN
El ñame (Dioscorea spp.) es un alimento de importancia económica para los habitantes del Oeste de África, de Asia, del Lejano Oriente, del Pacifico y del Caribe (Obidiegwu et al. 2009). El ñame es un rizoma de amplia distribución en la región Caribe colombiana, siendo D. alata, la especie más ampliamente distribuida, debido a su potencial productivo. A pesar de la importancia de D. alata en la dieta de miles de personas en la región Caribe y su participación en la economía regional (Álvarez, 2000), varios cultivares presentan susceptibilidad a plagas y enfermedades, en especial, a la antracnosis, causada por el hongo Colletotrichum gloeosporioides Penz. Estos problemas crean la necesidad de desarrollar variedades mejoradas que combinan altos rendimientos, tolerancia a la antracnosis y de buena aceptación en el mercado (Campo et al. 2009).
La Universidad de Córdoba tiene en su colección accesiones caracterizadas morfológicamente y evaluadas agronómicamente, de interés directo para los agricultores. Complementando estas con caracterización molecular, se puede obtener una información sobre la diversidad del germoplasma, para ser utilizado por los fitomejoradores, además, se proporciona datos útiles para la selección de progenitores de la población básica y es aplicada, frecuentemente, como una herramienta para estimar variabilidad genética en una especie (Bonin et al. 2007).
La accesiones de ñame conservadas en el banco de germoplasma de la Universidad de Cordoba presenta una amplia diversidad genética, tanto a nivel inter como intraespecífico (Rivera et al. 2011). Los intentos de caracterizar el ñame empleando marcadores morfológicos (Hamon & Touré, 1990b) e isoenzimaticos (Hamon & Touré, 1990a; Dansi et al. 2000), no han revelado resultados concluyentes, debido a su alto grado de variabilidad (Tamiru et al. 2007). Estudios de diversidad mostraron diferencias, tanto en especies de ñame de origen silvestre como cultivadas en el sur de Etiopia y especies cultivadas en África occidental, como D. alata L., D. bulbifera L., D cayenensis L. y D. rotundata Poir (Tamiru et al. 2007). La utilización de marcadores moleculares, como la técnica de polimorfismo de longitud del fragmentos amplificados (AFLP) (Vos et al. 1995), se ha utilizado en ñame para estudios taxonómicos, filogenéticos y de diversidad genética (Ramser et al. 1997; Mignouna et al. 1998; Dansi et al. 2000; Rivera et al. 2011).
El objetivo de esta investigación fue determinar la variabilidad genética de 14 accesiones de ñame de la colección de germoplasma de la Universidad de Córdoba, mediante la técnica de marcadores AFLP.
MATERIALES Y MÉTODOS
Para la obtención del ADN, se colectó tejido foliar de 14 accesiones de D. alata, pertenecientes a la colección de germoplasma de ñame de la Universidad de Córdoba (Tabla 1), las cuales, fueron colectadas en los departamentos de Córdoba, de Sucre, de Bolívar y del Magdalena (Colombia). El tejido foliar, se almacenó en bolsas plásticas de cierre hermético, con 50g de sílica gel, suficientes para mantener la estabilidad del ADN, de hasta 5g de tejido. Posteriormente, el tejido se maceró con nitrógeno líquido y se almacenó, a una temperatura de -196°C.

La caracterización molecular, se realizó en el Laboratorio de Biología Molecular del Instituto de Investigación de Recursos Biológicos "Alexander von Humboldt". Para la extracción del ADN, se tomó tejido foliar de las diferentes accesiones maceradas con nitrógeno líquido. La extracción de ADN, se realizó utilizando el kit comercial de Qiagen® 2006, modificando condiciones, como la reducción de la temperatura de incubación a 60°C y tiempo de centrifugado, a 5min. La concentración de ADN, se determinó mediante el uso del marcador de peso de ADN Lambda 20-bp., mediante electroforesis en gel de agarosa al 1%, teñido con bromuro de etidio y visualizados en luz UV. Finalmente, las extracciones fueron diluidas en buffer TE, obteniendo una concentración final de 50ng μL-1, por muestra.
Marcadores AFLP y amplificación por PCR: El ADN aislado de cada una de las accesiones, se sometió a digestión previa con enzimas de restricción EcoRI y MseI, de acuerdo a las instrucciones del kits comercial de Invitrogen, 2003 (Life Technologies®); posteriormente, fueron adicionados dos adaptadores a los fragmentos de ADN generados en el proceso de restricción, para ser amplificados mediante el uso de PCR (Polymerase Chain Reaction). Se utilizaron cebadores complementarios a la secuencia de los adaptadores, se hizo una preamplificación +1/+1 con nucleótidos adicionales; los fragmentos de ADN se amplificaron, a partir de los fragmentos generados en la reacción de digestión de cada una de las muestras en estudio (Tabla 2). Para la amplificación selectiva +3/+3 nucleótidos, se evaluaron, inicialmente, cinco genotipos de ñame, tomados al azar, con seis combinaciones de cebadores, reportados por Tamiru et al. (2007), para seleccionar las tres combinaciones más polimórficas y de mayor resolución, que serían usados para evaluar todos los genotipos de ñame.

El programa de PCR para el inicio del ciclo +3/+3 fue el siguiente: un ciclo a 94°C, por 30s; 65°C, por 30s y 72°C, por 60s; se bajó la temperatura de apareamiento 0,7°C en cada ciclo, durante 12 ciclos y se realizaron 23 ciclos así (tiempo total: 2h, 2min): 94°C, por 30s, 56°C, por 30s y 72°C, por 60s; el producto amplificado, se almacenó, finalmente, a 4°C. Las reacciones de amplificación fueron visualizadas en un gel de poliacrilamida al 6% teñido con plata, de donde se capturó la distribución de los loci.
Para el análisis de datos, debido a la naturaleza dominante de los marcadores AFLP, se construyeron matrices binarias originadas en la amplificación de los fragmentos digeridos. Con la matriz de datos, se calculó el índice de similitud de Dice, adaptado por Nei & Li (1979), para datos moleculares. El índice promedia los valores de similitud por par de individuos, mediante la siguiente ecuación: Sij= 2a / (2a+b+c); donde, Sij= similitud entre los individuos i y j; a= número de loci compartidos por i y j; b= número de loci presentes en i, pero ausentes en j, y, c= número de loci presentes en j, pero ausentes en i. Las matrices y los dendrogramas de similitud, se construyeron con el programa NTSYS-PC, versión 2.02i (Rohlf, 1998), mediante el método UPGMA y el agrupamiento SAHN, respectivamente. También, se analizaron las relaciones entre individuos, mediante el Análisis de Correspondencia Múltiple (ACM) con toda la población, para obtener una representación gráfica de la distancia entre las accesiones. Para estimar la confiabilidad de los grupos genéticos designado en los dendrogramas, se realizó la confirmación del análisis de conglomerados y de los grupos de diversidad, mediante remuestreo de 1000 permutaciones, para lo cual, se utilizó el software WINBOOT (Yap & Nelson, 1996).
RESULTADOS Y DISCUSIÓN
Las combinaciones de cebadores empleados en esta investigación presentaron patrones altamente polimórficos entre las accesiones, mostrando así perfiles de loci diferentes, garantizando un buen poder de discriminación, que permitió la identificación de grupos genéticos disímiles dentro de la especie D. alata. El número total de loci, que se obtuvo en todo el estudio, fue de 184, que se extendieron en un rango de 68 loci, para la combinación del cebador E-AAC/M-CAC, hasta 53 loci, para la combinación del cebador E-ACA/M- CAT, con un promedio de 61,33 loci, por par de cebadores. La combinación del cebador E-AAG/M-CTC (Figura 1), que se utilizó en el presente trabajo, muestra porcentajes de fragmentos polimórficos de 95,23%, muy similar a la reportada por Tamiru et al. (2007); la combinación del cebador E-AAC/M-CAC y E-ACA/M-CAT presentaron un porcentaje polimorfismo, de 77,94% y 62,26% (Tabla 3), respectivamente. Estos porcentajes son diferentes a los reportados por Tamiru et al. 2007, quien reportó porcentajes polimórficos para estas mismas combinaciones de cebadores, de 98,7 y 100%, respectivamente. Lo antes anotado, evidencia que el marcador es lo suficientemente sensible, para mostrar diferencias entre las accesiones estudiadas.


El análisis de correspondencia múltiple (ACM) estimó la variación de los individuos en tres dimensiones (ejes x, y, z) (Figura 2), mostrando un patrón de información similar a la obtenida en el dendrograma de similitud; las muestras que se encuentran más alejadas en cada coordenada, se deben separar como muestras diferenciadas genéticamente, en tanto que aquellas que se agrupan, se deben considerar muy cercanas, desde el punto de vista genético.

El estudio muestra cuatro grupos principales con cierto grado de variabilidad genética. El grupo 1 está conformado por ocho genotipos colectados en el departamento de Córdoba y uno colectado en el departamento de Bolívar. Se separa de las demás accesiones un primer sub-grupo, representado por los genotipos 9506-027 y 0406-100 (Figura 3-A), caracterizado por presentar flores masculinas y presencia en el tallo alas de color verde, rizomas subterráneos de color de pulpa blanca; este grupo presenta un valor promedio de 0,976 de similaridad. Un segundo subgrupo conformado por los genotipos 0406-002, 0406-009, 0406-037, 0406-098, 0406-062, 0406-094 y 0406-130, representado con un valor promedio de 0,972 de similitud; se caracteriza por presentar floración femenina, tallo cuadrado y presentar alas muy pequeñas de color morado, material completamente rústico y crecimiento vigoroso. Las hojas presentaron ápice agudo y sus flores son de color amarilla, simples en forma de racimo; los rizomas mostraron forma irregular, vellosidad y muchas raíces de fijación.
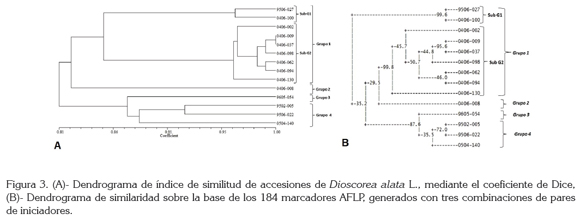
En la región Caribe, el ñame es muy conocido con diferentes nombres vulgares, entre los cuales, están 0406-002 (Ñame pepita), 0406-009, 0406-037 y 0406-094 (Mampuján), 0406- 062 (Ñame manteco) y el 0406-130 (Te encontré). De acuerdo a los resultados mostrados por el análisis de agrupamiento UPGMA (Figura 3-A), utilizando la combinación de cebadores E-ACA/M-CAT, muestra que las accesiones 0406-009, 0406- 037 es el mismo genotipo, lo cual, es comprobado por tener un coeficiente de similaridad, lo que implica que estas dos accesiones son clones, al compartir la misma información genética. Esto se evidencia, debido a las semejanzas morfológicas, como la coloración verde y morado en el tallo, las hojas simples de forma acorazonada y ápice agudo, las flores de color amarillas, simples y compuestas, en forma de racimo, rizomas de gran tamaño con vellosidad. Estas accesiones poseen resistencia a antracnosis (Campo et al. 2009).
El grupo 2 conformado por la accesión 0406-008 (Ñame peludo), colectado en el departamento de Córdoba (Colombia), presentó un coeficiente promedio de similaridad de 0,82. Tiene como característica principal la forma cuadrada del tallo, su color es verde con pigmentos marrón, presentó alas muy pequeñas, que son pliegues que se encuentran localizadas a lo largo del tallo, su crecimiento es indeterminado y alcanza de cuatro a seis metros de largo; presenta enrollamiento de forma dextral, es decir, en sentido de las manecillas del reloj. Las hojas son de forma larga y ancha (sagitada) y lobulado poco profundo, separado ápice muy agudo. Sus flores son femeninas, los rizomas son ramificados, la pulpa es de color blanca y textura lisa, el rizoma posee vellosidad y raíces de fijación de color marrón oscuro, su forma es irregular, también posee tubérculos aéreos, de forma redonda.
El grupo 3 (Figura 3-A), conformado por la accesión 9605-054 (Ñame seda), colectado en el departamento de Magdalena (Colombia), presenta un coeficiente alto de similaridad de 0,871. Como característica principal de esta accesión presentó ausencia de bulbillos aéreos y ausencia de raíces secundarias en los rizomas, también precocidad y espinas en la base de los tallos jóvenes. El grupo 4 (Figura 3-A), conformado por las accesiones 9502-005, colectado en el departamento de Sucre (Colombia) y la accesión 9506- 022 y 0504-140, colectado en el departamento de Córdoba (Colombia), con un coeficiente de similaridad de 0,92, se encuentran compartiendo las características de formación de bulbillos aéreos y presencia de raíces secundarias en los rizomas; estas accesiones muestran resistencia al hongo C. gloeosporioides (Campo et al. 2009). Estas relaciones se ven soportadas por el análisis de conglomerados (Figura 3-B) de los valores de confianza de la matriz de distancia que fue de 99,8%, para la relación entre el primer y el segundo grupo, para el tercer y el grupo cuatro, con un 87,6% de confiabilidad.
No se presentó correspondencia geográfica entre cultivares procedente del departamento de Córdoba, de Sucre, de Bolívar y del Magdalena y las relaciones de similaridad de las accesiones, indicando que estos materiales que son introducidos han sido ampliamente distribuidos e intercambiados por productores en todo el Caribe. Estos resultados son consistentes con los reportados por Sonibare et al. (2010), encontrando tres grupos genéticos de D. dumetorum, en varios países de África Central y Occidental, donde estas accesiones no siguen de manera estricta una correlación entre la variabilidad genética observada y la distribución geográfica de colecta. Esto es atribuible a la dispersión biológica o a las actividades humanas, que conducen a intercambio de germoplasma entre los agricultores, a través de los diferentes lugares donde se toman las muestras (Scarcelli et al. 2006); de igual forma, Obidiegwu et al. (2009) encontraron resultado similares en D. alata, concluyendo que el alto grado del intercambio y de transferencia de cultivares, por parte de los agricultores locales, inciden en la no correlación entre la variabilidad genética y la distribución geográfica.
En este estudio, se reveló una importante variación genética dentro de la especie D. alata en la región Caribe colombiana, mediante la utilización de la técnica AFLP; esta técnica tiene la suficiente sensibilidad para detectar polimorfismo intraespecifico, en D. alata (Petro et al. 2011). Estos resultados son coincidentes con los manifestados por Rivera et al. (2011), quienes reportaron alta variabilidad genética, mediante un patrón de organización estructural bien definido en D. alata y una correspondencia entre la caracterización molecular y la morfológica previa, consistente en la asociación de caracteres botánicos y morfológicos. Los niveles de variabilidad entre grupos taxonómicos en Dioscorea spp., también ha sido estudiada por Lebot et al. (1998), quienes indican la distinción de cultivares de D. alata, distribuidos en Asia, en África y en Melanesia. Malapa et al. (2005) revelaron que D. alata, es una especie heterogénea, encontrando diferencias en tres grupos principales de genotipos dentro de D. alata y con diferentes niveles de ploidía, mediante la utilización de marcadores AFLP. El nivel relativamente alto de la variación genética mostrada por estas accesiones podría ser atribuible a la adaptación a diferentes condiciones ambientales locales (Scarcelli et al. 2006).
Los cuatros grupos genéticos comparten características, como forma de crecimiento, presencia o ausencia de alas y espinas en tallos, forma, color de hojas y rizomas, además, se presenta agrupación de algunas accesiones, como los genotipos 0406-002, 0406-009 y 0406-037, que presentaron una característica importante de resistencia a antracnosis; este resultado tiene gran importancia agronómica, ya que dichas accesiones se pueden tener en cuenta para futuros trabajo de fitomejoramiento, que generen cultivares mejorados resistentes al hongo C. gloeosporioides (Campo et al. 2009). Esta información generada puede ser de gran utilidad para la conservación de la especie y para realizar estrategias de colecta, que permitan ampliar la colección de germoplasma.
Recomendaciones: Es importante para futuras investigaciones trabajar con marcadores codominantes o marcadores de polimorfismo funcional EST-SSRs y SNPs, que permitan identificar accesiones promisorias, contrastantes y fértiles, que contribuyan para el desarrollo de estrategias, mediante hibridaciones en futuros programas de fitomejoramiento.
Agradecimientos: Los autores expresan sus agradecimientos a la Universidad de Córdoba, por la financiación de esta investigación, a través del proyecto "Obtención de genotipos de ñame Dioscorea alata L, resistentes a la antracnosis, Colletotrichum gloeosporioides Penz y Sacc, en la Costa Norte colombiana", así como al personal del grupo técnico de producción de Ñame del programa de Ingeniería Agronómica y al personal del laboratorio de Biología Molecular del Instituto de Investigación de Recursos Biológicos "Alexander von Humboldt". CIAT. Palmira, Valle del Cauca - Colombia. Conflictos de intereses: El manuscrito fue preparado y revisado con la participación de todos los autores, quienes declaramos que no existe ningún conflicto de intereses, que ponga en riesgo la validez de los resultados presentados.
BIBLIOGRAFÍA
1. ÁLVAREZ, A. 2000. Prácticas agronómicas para el cultivo del ñame. En: Ñame, producción de semilla por biotecnología. Editorial Unibiblos Bogotá, Colombia. Universidad Nacional de Colombia. p.33-39. [ Links ]
2. BONIN, A.; EHRICH, D.; MANEL, S. 2007. Statistical analysis of amplified fragment length polymorphism data: a toolbox for molecular ecologists and evolutionists. Molecular Ecology. 16:3737-3758. [ Links ]
3. CAMPO, R.; LUNA, J.M.; JIMÉNEZ, Y. 2009. Selección de genotipos de ñame Dioscorea spp., resistentes a la antracnosis (Colletotrichum gloeosporiodes Penz). Fitopat. Col. 33(1):7-10. [ Links ]
4. DANSI, A.; MIGNOUNA, H.D.; ZOUNDJIHEKPON, J.; SANGARE, A.; ASIEDU, R.; AHOUSSOU, N. 2000. Using isozyme polymorphism to assess genetic variation within cultivated yams (Dioscorea cayenensis/Dioscorea rotundata complex) of the Republic of Benin. Genet. Resour. Crop Evol. 47:371- 383. [ Links ]
5. HAMON, P.; TOURÉ, B. 1990a. Characterization of traditional yam varieties belonging to the Dioscorea cayenensis-rotundata complex by their isozymic patterns. Euphytica. 46:101-107. [ Links ]
6. HAMON, P.; TOURÉ, B. 1990b. The classification of cultivated yams (Dioscorea cayenensis-rotundata complex) of West Africa. Euphytica 47:179-187. [ Links ]
7. LEBOT, V.; TRILLES, B.; NOYER, J.L.; MODESTO, J. 1998. Genetic relationships between Dioscorea alata L. cultivars. Genet. Resour. Crop Evolut. 45:499-508. [ Links ]
8. MALAPA, R.; ARNAU, G.; NOYER, J.L.; LEBOT, V. 2005. Genetic diversity of the greater yam (Dioscorea alata L.) and relatedness to D. nummularia Lam. and D. transversa Br. as revealed with AFLP markers. Genet. Res. Crop Evol. 52:919-929. [ Links ]
9. MIGNOUNA, H.D.; ELLIS, N.T.H.; KNOX, M.R.; ASIEDU, R.; NG, Q.N. 1998. Analysis of genetic diversity in Guinea yams (Dioscorea spp.) using AFLP fi nger printing. Trop. Agric. (Trinidad). 75:224-229. [ Links ]
10. NEI, M.; LI, W.H. 1979. Mathematical Model for Studying Genetic Variation in Terms of Restriction Endo Nucleases. Proc. Nal Acad. Sci. of the United States of America. 76(10):5269-5273. [ Links ]
11. OBIDIEGWU, J.E.; ASIEDU, R.; ENE-OBONG, E.E.; MUONEKE, C.O.; KOLESNIKOVA-ALLEN, M. 2009. Genetic characterization of some water yam (Dioscorea alata L.) accessions in West Africa with simple sequence repeats. J. Food, Agr. & Environm. 7(3&4):634-638. [ Links ]
12. PETRO, D.; ONYEKA, T.J.; ETIENNE, S.; RUBENS, S. 2011. An intraspecific genetic map of water yam (Dioscorea alata L.) based on AFLP markers and QTL analysis for anthracnose resistance. Euphytica. 179:405-416. [ Links ]
13. RAMSER, J.; WEISING, K.; LÓPEZ-PERALTA, C.; TERHALLE, W.; TERAUCHI, R.; KAHL, G. 1997. Molecular marker based taxonomy and phylogeny of Guinea yam (Dioscorea rotundata-D. cayenensis). Genome. 40:903-915. [ Links ]
14. RIVERA-JIMÉNEZ, H.; ÁLVARES, A.; PALACIO, J.; BARRIOS, D.; LOPEZ, D. 2011. Diversidad genética intra e inter-especifica de ñame (Dioscorea spp.) de la región Caribe colombiana mediante marcadores AFLP. Acta Agr. 60(4):328-338. [ Links ]
15. ROHLF, F. 1998. NTSYSpc. Numerical taxonomic and multivariated analysis system, vol 2.0, Exeter Software, Setauket, New York, USA. [ Links ]
16. SCARCELLI, N.; TOSTAIN, S.; MARIAC, C.; AGBANGLA, C.; Da, O.; BERTHAUD, J.; PHAM, J. 2006. Genetic nature of yams (Dioscorea sp.) domesticated by farmers in Benin (West Africa). Gen. Res. Crop Evol. 53:121-130. [ Links ]
17. SONIBARE, M.A.; ASIEDU. R.; ALBACH, D.C. 2010. Genetic diversity of Dioscorea dumetorum (Kunth) Pax using Amplified Fragment Length Polymorphisms (AFLP) and cpDNA. Biochem. Syst. Ecol. 38:320- 334. [ Links ]
18. TAMIRU, M.; BECKER, H.; MAASS, B. 2007. Genetic diversity in yam germplasm from Ethiopia and their relatedness to the main cultivated Dioscorea species assessed by AFLP markers. Crop Sci. 47:1744-1753. [ Links ]
19. VOS, P.; HOGERS, R.; BLEEKER, M.; REIJANS, M.; VAN DE LEE, T.; HORNES, M.; FRIJTERS, A.; POT, J.; PELEMAN, J.; KUIPER, M.; ZABEAU. M. 1995. AFLP: a new technique for DNA fingerprinting. Nucleic Acids Res. 23:4407-4414. [ Links ]
20. YAP, I.V.; NELSON, R.J. 1996. WINBOOT: A program for performing bootstrap analysis of binary data to determine the confidence limit of UPGMA based dendrograms. IRRI Discussion Paper Ser. 14. IRRI, Los Baños, Philippines. [ Links ]
Recibido: Agosto 25 de 2011 Aceptado: Junio 10 de 2012













