Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Infectio
Print version ISSN 0123-9392
Infect. vol.13 no.3 Bogotá July/Sept. 2009
1 Universidad Colegio Mayor de Cundinamarca, Facultad de Ciencias de la Salud, Programa de Bacteriología y Laboratorio y Clínico.
2 Universidad Nacional de Colombia sede Bogotá, Facultad de Medicina, departamentos de Obstetricia, Ginecología y de Pediatría.
3 Universidad El Bosque, Instituto de Genética Molecular Bacteriana.
Recibido: 12/02/2009; Aceptado: 02/08/2009
Resumen
Objetivo. Determinar la presencia y secuencia del integrón clase I en un aislamiento clínico de Staphylococcus epidermidis proveniente de un neonato con diagnóstico de sepsis.
Materiales y métodos. En una cepa de S. epidermidis, aislada de una muestra de hemocultivo de un neonato, se realizaron las pruebas de identificación microbiológica, susceptibilidad antimicrobiana y la caracterización molecular de los genes aac6´-aph2´´, mecA, el gen de la integrasa intI1 y el gen casete aac6´.
Resultados. Se identificó el gen de la integrasa intl1 y mediante secuenciación de nucleótidos se encontró una homología del 78% con las secuencias reportadas en la base de datos del NCBI para bacterias Gram negativas. También se evidenció el casete genético aac6´ presente dentro del integrón clase 1, el cual acetila los aminoglucósidos para conferir resistencia a este antibiótico; como parte de la caracterización molecular se encontró la presencia del gen (aac6´-aph2´´), que codifica para una enzima bifuncional que inactiva a los aminoglucósidos, y el gen mecA que confiere resistencia a los ß-lactámicos. En las pruebas de susceptibilidad se encontró resistencia a ampicilina, oxacilina y gentamicina.
Conclusiones. Se reporta por primera vez en Colombia, la secuencia del gen de la integrasa intl1 en un aislamiento de S. epidermidis, proveniente de un recién nacido con sepsis neonatal de la unidad de cuidados intensivos en un hospital de tercer nivel de Bogotá. Esta integrasa ha sido relacionada sólo en integrones clase 1, los cuales se han asociado a multirresistencia en aislamientos clínicos en bacterias Gram negativas.
El hallazgo de este mecanismo de resistencia, en el presente estudio realizado en una bacteria Gram positiva, sugiere la relación con transferencia horizontal de genes de resistencia entre especies, efectuado mediante la combinación de elementos genéticos móviles. Lo anterior resalta la importancia de la caracterización molecular de cepas causantes de sepsis para el control epidemiológico de la infección y el tratamiento en neonatos.
Palabras clave: Staphylococcus epidermidis, acetilación, aminoglucósidos, casete genético, integrón, integrasa, resistencia microbiana.
Abstract
Objective: To determine the presence and the sequence of the class one integron in a Staphylococcus epidermidis strain isolated from a septic neonatal patient.
Materials and methods: The S. epidermidis strain was isolated from a blood culture of a newborn. The microbiological identification, test of sensitivity and molecular characterization of the integrase gene int/1, the cassette gene aac6; aac6´aph2´´ resistant gene to aminoglycoside antibiotics and mecA gene resistant to ß-lactamics were realized.
Results: The nucleotide sequence of the integrase gen intl1 in S. epidermidis is reported, which showed a 78% similarity to the reported sequence of the NCBI data base in Gram negative bacteria. The gene cassette aac6 aac6´ (aminoglycoside acetilation) was also identified within the class one integron confering aminoglycoside resistance. Through molecular characterization we also found the aminoglycoside ß-lactamics resistance genes (aac6´aph2´y mecA ´); the susceptibility tests showed resistance to ampicilin, oxacilin and gentamicin.
Conclusions: The nucleotide sequence of the integrase gen int/1 is reported for the first time in Colombia in a S. epidermidis strain isolated from a septic neonatal patient, at the neonatal care unit of a third level hospital in Bogotá. This integrase in class one integron was reported, which has shown multiresistance association in clinical isolates in Gram negative bacteria.
The resistance mechanism found during this approach realized in Gram negative bacteria showed the evidence of an interspecies horizontal transfer especially by gene transfer or by moving elements such as integrons and gene cassettes. The molecular character of the causing agent of sepsis is important for epidemiological control of the infection and treatment in the newborn.
Key words: Staphylococcus epidermidis, acetilation, aminoglycoside, integron, integrasa, microbial resistance.
Introducción
En las unidades neonatales de cuidados intensivos un número importante de sepsis es causado por Staphylococcus epidermidis; se ha encontrado un incremento de la resistencia a aminoglucósidos y ß-lactámicos, en parte, como consecuencia de la presión selectiva a la que son sometidas por la exposición prolongada a antibióticos (1), que puede llegar a estimular la incorporación de elementos genéticos móviles, como los integrones, nuevo mecanismo de resistencia bacteriana descrito recientemente, el cual favorece la propagación y la diseminación de genes en las especies bacterianas y entre ellas (2,3).
La transferencia de genes requiere que el ADN sea incorporado en la célula huésped por procesos como transformación, conjugación y transducción, y de la capacidad de la célula para replicarse y expresar ADN foráneo en la población bacteriana, mediante selección natural. Los integrones se consideran uno de los elementos móviles más importantes en dicho proceso (4).
Existen, por lo menos, tres clases de integrones relacionados con la resistencia bacteriana que difieren en la secuencia de aminoácidos de sus integrasas. El integrón clase 1 ha sido el más estudiado y mejor caracterizado por su importancia clínica en la diseminación de resistencia a antibióticos en ambientes hospitalarios (5,6). En su estructura básica, los extremos 5´y 3´ son secuencias conservadas y entre éstas se encuentra una región muy variable en la que se pueden insertar casetes genéticos.
El extremo 5´conservado es un segmento de ADN formado por el gen intIl que codifica para la integrasa encargada de catalizar la recombinación específica de sitio y la integración y escisión de genes casete. La integrasa reconoce la secuencia attI y participa en la incorporación del casete de resistencia, dándole direccionalidad (7,8). Los dos promotores de orientación opuesta Pc, dirigen la transcripción de los casetes genéticos y el promotor PI controla la transcripción del gen de la integrasa (9).
El extremo 3´ es el menos conservado entre los distintos integrones; se encuentra formado por el gen qacE?1, el cual genera resistencia a compuestos de amonio cuaternario presentes en algunos antisépticos y desinfectantes; el gen sulI que determina resistencia a las sulfonamidas y que corriente abajo se inserta un fragmento de lectura abierta (orf 5) cuya función se desconoce hasta el momento (10).
Los casetes genéticos constituyen un grupo diverso de pequeños elementos móviles discretos que no poseen promotor propio, el cual es proporcionado por el integrón. Usualmente, contienen un sólo marco de lectura abierta, una región de codificación (orf) y una región de recombinación específica o sitio attC, que es reconocido por la integrasa para la inserción y escisión del casete genético (11).
Se pueden localizar desde 1 hasta 4 casetes genéticos separados por pequeñas regiones entre genes de, aproximadamente, 10 pb, las cuales facilitan la formación de una unidad de transcripción controlada por Pc. Su expresión depende de la ubicación con respecto al promotor, es decir, entre más cerca se encuentre del promotor la expresión será mayor, lo cual se relaciona con la expresión fenotípica de la bacteria (12). Estos elementos móviles se han asociado a resistencia a aminoglucósidos,ßlactámicos, macrólidos, sulfonamidas y quinolonas, entre otros (13).
El objetivo del presente trabajo es reportar, por primera vez en nuestro medio, la presencia del integrón clase I, su secuencia de nucleótidos y su relación con el casete genético aac6´ (acetilación de aminoglucósidos), los genes aac6´-aph2´´y mecA, que confieren resistencia a aminoglucósidos y ß-lactámicos, respectivamente, en un aislamiento de S. epidermidis proveniente de un hemocultivo de un neonato con diagnóstico de sepsis, en un hospital de tercer nivel de Bogotá.
Materiales y métodos
Historia clínica. Se trata de un recién nacido de sexo femenino, fruto de la primera gestación, 2° gemelo, con 34 semanas de edad de gestación, peso de 1.320 g, talla de 40,5 cm y perímetro cefálico de 26,5 cm. Fue necesario suministrarle respiración mecánica asistida durante 24 horas, administración de surfactante y bolo de cristaloide por hipotensión, fototerapia y plasmaféresis por policitemia.
Al tercer día de vida se encontró fractura de la diáfisis del fémur izquierdo, que se inmovilizó. Se adicionó manejo para el dolor. Se inició estímulo entérico mínimo y nutrición parenteral. A los 12 días de vida, presentó apneas profundas por lo que se reinició la respiración mecánica asistida durante 24 horas; se descartó sepsis bacteriana. Se cambió el catéter epicutáneo.
A los 19 días de vida se aumentó la vía oral; a los 22 días de vida presentó fiebre, se tomaron muestras para exámenes de laboratorio y, ante la sospecha de una infección, se administró vancomicina durante 10 días. La evolución fue satisfactoria y a los 38 días se le dio salida con un peso de 1.900 g. En el segundo hemocultivo de un examen seriado, se aisló S. epidermidis resistente a ampicilina, oxacilina y gentamicina, y sensible a vancomicina y ciprofloxacina. La identificación y las pruebas de susceptibilidad a antimicrobianos se llevaron a cabo por el sistema de Vitek (BioMérieux Francia).
Extracción de ADN. Se realizó mediante el kit Wizard® Genomic DNA Purification A1123, (Promega, Madison, WI, USA), siguiendo las indicaciones del fabricante. El ADN para los experimentos se utilizó a una concentración de 200 ng/µl.
Determinación de los genes 16S (especie específico), mecA y aac6´-aph2´´. Para su identificación se usaron los oligonucleótidos y las condiciones de amplificación descritos por Martineau et al. (14).
Análisis por bioinformática. La búsqueda de la secuencia consenso para el gen intl1 se llevó a cabo en la base de datos del National Center Biotechnology Information (NCBI) a partir de las secuencias reportadas de integrones en bacterias de los géneros Klebsiella spp., Salmonella spp., Serratia spp., Acinetobacter spp. y Citrobacter spp. Utilizando los programas Multalin y Primer3, se obtuvo la región más conservada para el diseño de oligonucleótidos. Los oligonucleótidos utilizados fueron el 5´CGAACCGAACAGGCTTATGT3´ y el 5´AGCACCTTGCCGTAGAGAAGAA 3´, y se generó un amplicón de 188 pb.
Por otra parte, se diseñaron los oligonucleótidos específicos para casetes genéticos que inactivan los aminoglucósidos por medio de la enzima acetil-transferasa (aac6¨), teniendo en cuenta las secuencias más conservadas reportadas en el NCBI para este gen, en las diferentes especies, así como, la longitud, la temperatura de fusión y el porcentaje de guanina-citosina, utilizando el programa Primer3. Los oligonucleótidos diseñados fueron el 5´GGTATGCCCAGTCGTACGTT3´ y 3´CGTTTGGATCTTGGTGACCT5´ para un amplicón de 218 pb.
Determinación del gen intl1. La mezcla contenía: GoTaq polimerasa 1U, oligonucleótidos 1 µM, dNTPs 50 µM, ADN 200 ng, en un volumen final de 20 µl. La desnaturalización inicial se hizo durante 4 minutos a 94ºC y 30 ciclos con un paso de desnaturalización de 30 s a 94ºC, hibridación 45 s a 53ºC, extensión 72ºC por 45 s y extensión final de 7 minutos a 72ºC. Como cepa control se utilizó un aislamiento clínico de Salmonella Typhi, portador del gen intl1 (Gen Bank EF051039).
Determinación del casete genético (aac6´).
La mezcla contenía: Taq Flexi 1U, oligonucleótidos 1 µM, dNTPs 200 µM, ADN 200 ng, en un volumen final de 20 µl. La desnaturalización inicial se hizo durante 4 minutos a 94ºC y 30 ciclos con un paso de desnaturalización de 30 s a 94ºC, hibridación de 45 s a 55ºC, extensión a 72ºC por 45 s y extensión final de 7 minutos a 72ºC. Como cepa control se utilizó un aislamiento clínico de Salmonella Typhi, portador del gen intl1 (Gen Bank EF051039).
Secuenciación. El producto de amplificación del gen intl1 obtenido de la cepa de S. epidermidis fue extraído del gel y purificado (Kit Exosapit GE Healthcare Latin America). La secuenciación se realizó por el método de terminación de cadena (Big dye terminator, Applied Biosystem, Foster City, CA) en un secuenciador automático (Applied Biosystem 3730xl DNA analyzer software, version 4.0, Foster City CA). Cada fragmento fue secuenciado en ambas direcciones para su confirmación.
Resultados
Antibiograma por concentración inhibitoria mínima (CIM). La cepa aislada mostró resistencia a ampicilina (CIM=16 µ/ml), oxacilina (CIM=2 µ/ml) y gentamicina (CIM=16 µ/ml), y resultó sensible a ciprofloxacina (CIM=0,5 µ/ml).
Caracterización molecular. Se determinaron en la cepa de S. epidermidis dos genes específicos para evidenciar la presencia del mecanismo de resistencia causado por el integrón clase 1. Para esto, se amplificaron tanto el gen de la integrasa inti1, como el gen casete aac6´ (figura 1). Además, se determinó la presencia de los genes mecA y, aac6´-aph2´´ que confieren resistencia a los antibióticos ß-lactámicos y aminoglucósidos, respectivamente.
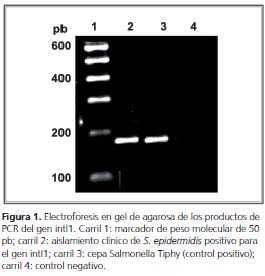
La secuencia de nucleótidos obtenida del gen intl1 se comparó con las reportadas en la base de datos del NCBI y se encontró una homología del 78% con lo informado en bacterias Gram negativas (figura 2).

Discusión
La resistencia bacteriana inducida por una constante exposición de las bacterias a los antibióticos, es un proceso de evolución natural y dinámico que permite la adquisición y la diseminación de genes mediante bacteriófagos, plásmidos y transposones, en la cual el ADN puede ser integrado al genoma bacteriano por transferencia horizontal o vertical de genes (15,16). La presencia de los integrones se considera como una de las mayores causas de multirresistencia en bacterias Gram negativas; sin embargo, en la última década ha sido relacionado con resistencia en bacterias Gram positivas (17,18).
En el Instituto Materno-Infantil de Bogotá se llevó a cabo un estudio en 67 cepas de Estafilococos coagulasa negativa (ECN) provenientes de hemocultivos y puntas de catéter de pacientes; se determinó la presencia del gen aac(6)-aph(2") en 59 (88,1 %) cepas, prevalencia idéntica a la informada por el European Study Group on Antibiotic Resistance (ESGAR) (19). No se encontró asociación estadísticamente significativa entre la presencia del gen aac(6)-aph(2") y la resistencia in vitro a gentamicina y amikacina; esta falta de correlación entre la presencia del gen y los resultados de las pruebas de susceptibilidad, podría deberse a la incorporación de elementos genéticos móviles, como los integrones (20).
En trabajos previos llevados a cabo por este grupo de investigación, se estudiaron 46 cepas de ECN, en las cuales se encontró el gen de la integrasa intl1 en los integrones clase 1 en 45,7% de las cepas, lo cual demuestra un nuevo mecanismo de resistencia bacteriana descrito recientemente, que favorece la propagación y la diseminación de genes intraespecies bacterianas y entre especies (2,3,18).
En este trabajo se reporta, por primera vez en Colombia, la secuencia del gen de la integrasa intl1 en un aislamiento de S. epidermidis, proveniente de un recién nacido con sepsis neonatal de la unidad de cuidados intensivos de un hospital de tercer nivel de Bogotá. El producto obtenido por PCR fue confirmado mediante secuenciación de nucleótidos. De acuerdo con el análisis bioinformático, usando la base de datos NCBI, se encontró una homología del 78% con la enzima integrasa codificada por el gen intl1 en integrones clase 1 de bacterias Gram negativas, lo que sugiere que puede estar relacionado con la transferencia horizontal de genes de resistencia entre especies, efectuado por medio de la combinación de elementos genéticos móviles o de un sistema receptor de enzimas celulares (21,22).
Los integrones presentan un sistema de recombinación específica de sitio para capturar casetes genéticos móviles desde el ambiente e incorporarlos; estos casetes se encuentran implicados en la resistencia bacteriana, particularmente a aminoglucósidos. Dentro del integrón clase 1, encontramos el casete genético aac6´ que tiene la capacidad de acetilar a la amikacina, preferiblemente, pero, también, a la gentamicina.
Este hallazgo concuerda con la prueba de susceptibilidad para la gentamicina, que mostró resistencia (CIM>16 µg/ml) y sugiere que la presencia de ese elemento genético móvil estaría implicado en la resistencia, tanto a amikacina como a gentamicina, en S. epidermidis aislado en nuestro paciente. Sin embargo, es importante tener en cuenta que, dependiendo del sitio donde se ubique el casete genético en relación con el marco de lectura, su expresión fenotípica puede variar (9,13).
La caracterización molecular de los genes que confieren resistencia a ß-lactámicos (mecA) y a aminoglucósidos (aac6´-aph2´´) en esta misma cepa de S. epidermidis, puede explicar el fenómeno de multirresistencia debida a exposición a antibióticos, antes que a una independiente selección in situ. Este punto de vista es sustentado por hallazgos similares observados en otros estudios, según los cuales algunas cepas resistentes son ocasionalmente introducidas para reemplazar la población susceptible (1,23). No obstante, permanecen sin explicar los mecanismos responsables de la diversidad genética que mantiene una alta prevalencia de resistencia en ausencia del uso de antibióticos.
En estudios adicionales realizados a la cepa informada en el presente artículo, también se encontraron los operones icaADBC e icaR, lo cual sugiere que esta cepa es formadora de biopelícula, la cual contribuye a la virulencia, resistencia y evasión del sistema inmune, protegiéndose de la opsonización y fagocitosis (24,25).
El hallazgo de diferentes tipos de genes bacterianos que confieren resistencia a S. epidermidis, considerado hasta hace poco como un simple contaminante, demuestran la importancia clínica de este germen. Esto es particularmente importante en neonatos, en quienes S. epidermidis se considera el principal causante de infecciones hospitalarias (26-28). La caracterización molecular de cepas causantes de sepsis neonatal puede constituirse en una tamización molecular sencilla y útil para el tratamiento y el control de la infección hospitalaria.
Agradecimientos
A Lourdes Loidi Fernández del Laboratorio de Medicina Molecular del Complejo Hospitalario Santiago de Compostela, España; a Adriana Velazco, Jennifer Gutiérrez y Martha Gómez del Universidad Colegio Mayor de Cundinamarca; a Tailandia Rodríguez, el Hospital Simón Bolívar, el Hospital de la Samaritana y la Universidad El Bosque (Laboratorio de Genética Molecular), por sus aportes y apoyo en la realización del presente estudio.
Correspondencia:
Gladys Pinilla, Grupo REMA, gpinilla@gmail.com
Bibliografía
1. Pallecchi L, Lucchetti CH, Bartoloni A, Bartalesi F, Mantella A, Gamboa H, et al. Population structure and resistance genes in antibiotic-resistant bacteria from a remote community with minimal antibiotic exposure. J Antimicrob Chemother. 2007;51:1179-84. [ Links ]
2. Mazel D. Integrons: agents of bacterial evolution. Nat Rev Microbiol. 2006;4:608-20. [ Links ]
3. Pan J-C, Ye R, Meng D-M, Zhang W, Wang H-Q, Liu K-Z. Molecular characteristics of class 1 and class 2 integrons and their relationships to antibiotic resistance in clinical isolates of Shigella sonnei and Shigella lexneri. J Antimicrob Chemother. 2006;58:288-96. [ Links ]
4. Smets BF, Barkay T. Horizontal gene transfer: perspectives at a crossroads of scientific disciplines. Nat Rev Microbiol. 2005;3:675-8. [ Links ]
5. Nield BS, Holmes AJ, Gillings MR, Recchia GD, Mabbutt BC, Nevalainen KM, et al. Recovery of new integron classes from environmental DNA. FEMS Microbiol Lett. 2001;195:59-65. [ Links ]
6. Gilling M, Boucher Y, Labbate M, Holmes A, Krishnan S, Holley M, et al. The evolution of class 1 integrons and the rise of antibiotic resistance. J Bacteriol. 2008;190:5095-100. [ Links ]
7. González G, Mella S, Zemelman R, et al. Integrones y cassettes genéticos de resistencia: estructura y rol frente a los antibacterianos. Rev Méd Chile. 2004;132:619-26. [ Links ]
8. García J. Estructura, funcionamiento y significado de los integrones bacterianos. Actualidad SEM. 1999;28:18-22. [ Links ]
9. Stokes HW, Holmes AJ, Nield BS, Holley MP, Nevalainen KM,Mabbutt BC, et al. Gene cassette PCR: sequence-independent recovery of entire genes from environmental DNA. Appl Environ Microbiol. 2001;67:5240-6. [ Links ]
10. Martinez-Freijo P, Fluid A, Schnitmitz F, Greck V, Verhoef J, Jones M. Class I integrons in Gram-negative isolates from different European hospital and association with decreased susceptibility to multiple antibiotics compounds. J Antimicrob Chemoter. 1998;42:689-96. [ Links ]
11. Demarre G, Frumerie C, Gopaul DN, Mazel D. Identification of key structural determinants of the IntI1 integron integrase that influence attC_attI1 recombination efficiency. Nucleic Acids Res. 2007;35:6475-89. [ Links ]
12. Fluit AC, Schmitz FJ. Resistance integrons and super-integrons. Clin Microbiol Infect. 2004;10:272-88. [ Links ]
13. Mendes R, Castanheira M, Toleman M, Sader H, Jones R, Walsh T. Characterization of an integron carrying blaIMP-1 and a new aminoglycoside resistance gene, aac(6´)-31, and its dissemination among genetically unrelated clinical isolates in a Brazilian Hospital. Antimicrob Agents Chemother. 2007;51:2611-4. [ Links ]
14. Martineau f, Picard FJ, Lansac N, Ménard C, Roy PH, Ouellette M, et al. Correlation between the resistance genotype determined by multiplex PCR assays and the antibiotic susceptibility patterns of Staphylococcus aureus and Staphylococcus epidermidis. Antimicrob Agents Chemother. 2000;44:231-8. [ Links ]
15. De la Cruz F, Davies J. Horizontal gene transfer and the origin of species: lessons from bacteria. Trends Microbiol. 2000;8:128-33 [ Links ]
16. Nemergut DR, Robeson MS, Kysela RF, Martin AP, Schmidt SK, Knight R. Insights and inferences about integron evolution from genomic data. BMC Genomics. 2008;9:261-72. [ Links ]
17. Xu Z, Shi L, Zhang C, Zhang L, Li X, Cao Y, et al. Nosocomial infection caused by class 1 integron-carrying Staphylococcus aureus in a hospital in South China. Clin Microbio Infect. 2007;13:980-4. [ Links ]
18. Pinilla G, Muñoz L, Gallego E, Chavarro B, Fandiño J. Presencia de integrones clase 1 en aislamientos de Staphylococcus epidermidis de las unidades de neonatología del Instituto Materno-Infantil de Bogotá. NOVA. 2006;6:55-7. [ Links ]
19. Dornbusch K, Miller G, Hare R, Shaw K. Resistance to aminoglycoside antibiotics in Gram-negative bacilli and staphylococci isolated from blood. Report from a European collaborative study. The ESGAR Study Group (European Study Group on Antibiotic Resistance). J Antimicrob Chemother. 1990;26:131-44. [ Links ]
20. Muñoz L, Pinilla B, Ruiz AI, Cifuentes Y, Gallego E. Determinación del gen aac(6)-aph(2") asociado con resistencia aminoglucósidos en cepas de estafilococos coagulasa negativo aisladas en un hospital de tercer nivel de Bogotá, Colombia. Abstract Infectio. 2006; 10:118 [ Links ]
21. Shi L, Zheng M, Xiao Z, Asakura M, Su J, Li L, et al. Unnoticed spread of class 1 integrons in gram-positive clinical strains isolated in Guangzhou, China. Microbiol Immunol. 2006;50:463-7. [ Links ]
22. Vinué L, Sáenz Y, Somalo S, Escudero E, Moreno MA, Ruiz-Larrea F, et al. Prevalence and diversity of integrons and associated resistance genes in faecal Escherichia coli isolates of healthy humans in Spain. J Antimicrob Chemother. 2008;62:934-7. [ Links ]
23. Härtel C, Osthues I, Rupp J, Haase B, Röder K, Göpel W, et al. Characterisation of the host inflammatory response to Staphylococcus epidermidis in neonatal whole blood. Arch Dis Child Fetal Neonatal Ed. 2008;93:F140-5. [ Links ]
24. Deighthon M, Borland R, Capstick JA. Virulence of Staphylococcus epidermidis in a mouse model: significance of extracellular slime. Epidemiol Infect. 1996;117:267-80. [ Links ]
25. Götz F. Staphylococcus and biofilms. Mol Microbiol. 2002;43:1367-78. [ Links ]
26. Klingenberg C, Rønnestad A, Anderson AS, Abrahamsen TG, Zorman J, Villaruz A, et al. Persistent strains of coagulase- negative staphylococci in a neonatal intensive care unit: virulence factors and invasiveness. Clin Microbiol Infect. 2007;13:1100-11. [ Links ]
27. Venkatesh MP, Pham D, Fein M, Kong L, Weisman LE. Neonatal coinfection model of coagulase-negative Staphylococcus (Staphylococcus epidermidis) and Candida albicans: fluconazole prophylaxis enhances survival and growth. Antimicrob Agents Chemother. 2007;51:1240-5. [ Links ]
28. Cifuentes Y, Ruiz A, Leal AL, Muñoz L, Herrera M, Jiménez L. Perfil microbiológico de aislamientos en unidades neonatales en un hospital de tercer nivel de Bogotá, Colombia. Rev Salud Pública. 2005;7:121-9. [ Links ]














