Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Infectio
Print version ISSN 0123-9392
Infect. vol.14 no.4 Bogotá Oct./Dec. 2010
ARTÍCULO ORIGINAL
1 Grupo de Inmunovirología, Universidad de Antioquia, Medellín, Colombia
2 Centro de Análisis Molecular, Bogotá, D.C., Colombia
Recibido: 18/08/2010; Aceptado: 30/11/2010
Resumen
Objetivo. Caracterizar el fenómeno de la resistencia a los diferentes medicamentos antirre-trovirales en pacientes en quienes se practicó el estudio de genotipificación en Colombia, durante el período 2000-2007.
Diseño. Descriptivo, retrospectivo y basado en el laboratorio.
Métodos. Entre los años 2000 y 2007, se obtuvo la secuencia de los genes de la proteasa y transcriptasa inversa del virus de la inmunodeficiencia humana (VIH) de 650 pacientes presuntamente en tratamiento antirretroviral, procedentes de diferentes regiones de Colombia. Las secuencias se procesaron con el programa Geno2pheno resistance, el cual infiere la resistencia a partir de la secuencia.
Resultados. El 82,1% de las cepas virales fue resistente a uno o más medicamentos. La frecuencia de resistencia fue mayor para la lamivudina (55,4%), la nevirapina (54,3%) y el efavirenz (52,6%), y más baja para la estavudina (11,0%). El 45,1% fue resistente a la zidovudina. La resistencia a los inhibidores de proteasa osciló entre 30% y 38%. Se observó que la frecuencia de resistencia va en aumento para los inhibidores de la transcriptasa inversa, pero no para los inhibidores de proteasa.
Conclusiones. La frecuencia de resistencia fue mayor en los antirretrovirales con una baja barrera genética y en aquéllos cuyo uso se ha incrementado en los últimos años.
Palabras clave. VIH, sida, tratamiento antirretroviral, mutación, resistencia, genotipificación, Colombia.
Abstract
Objective. To characterize the resistance to anti-retroviral drugs in patients subject of genotypic sensitivity testing in Colombia during 2000-2007.
Design. Descriptive, retrospective, laboratory based.
Methods. Between the years 2000 and 2007 the sequences of the HIV protease and transcriptase genes were obtained from 650 patients presumably under antiretroviral treatment throughout the country. The sequences were processed with the program “geno2pheno resistance”, which infers the resistance by using the sequences. The results were statistically analyzed.
Results. 82.1% of the viral strains were resistant to one or more drugs. The frequency of resistance was higher for lamivudine (55.4%), nevirapine (54.3%), and efavirenz (52.6%), and lower for stavudine (11.0%). 45.1% were resistant to zidovudine. Resistance to protease inhibitors varied between 30% and 38%. We observed that the frequency of resistance is rising for the reverse transcriptase inhibitors but not for the protease inhibitors.
Conclusions. The frequency of resistance was higher for the antiretroviral drugs with a lower genetic barrier and for those which use has increased during these last years.
Key words. HIV, AIDS, antiretroviral therapy, resistance, mutation, genotyping, Colombia.
Introducción
El número de personas infectadas por el virus de la inmunodeficiencia humana (VIH) sigue en aumento, al igual que el número de muertos por su causa. Se estima que en el 2007, aproximadamente, 33,2 millones de personas (30,6 a 36,1 millones) se encontraban infectadas por el VIH en el mundo (1). En Colombia, se estimó que en el 2007 el número de adultos de 15 a 49 años con VIH/sida era de 170.000, con una prevalencia entre 0,4% y 0,8% de la población (2).
Desde finales de los años 80, numerosos fármacos antirretrovirales han sido aprobados y, desde mediados de los años 90, se encuentra disponible el tratamiento HAART (Highly Active Antiretroviral Therapy), que consiste en la combinación de tres o más medicamentos (3). Estos pueden clasificarse en cinco grupos: inhibidores nucleósidos de transcriptasa inversa, inhibidores no nucleósidos de transcriptasa inversa, inhibidores de proteasa, inhibidores de entrada e inhibidores de integrasa (tabla 1). Los primeros tres grupos han sido utilizados por más de una década, mientras que los inhibidores de entrada y de integrasa fueron introducidos muy recientemente y su uso aún no se ha generalizado en Colombia, por lo que este estudio se centra en los tres primeros.
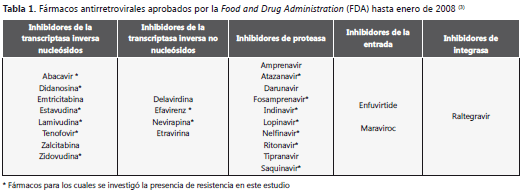
Hasta 1996 el uso de medicamentos antirretrovirales en Colombia fue irregular. En 1997 se incluyeron en el plan obligatorio de salud (POS) los siguientes fármacos: zidovudina, didanosina, lamivudina, indinavir y ritonavir. En los años siguientes se fueron agregando otros medicamentos en dicho plan; por ejemplo, estavudina, nelfinavir y nevirapina se incluyeron en 2002, abacavir, efavirenz y lopinavir/ritonavir, en 2004, y saquinavir, fosamprenavir y atazanavir, en 2006. Sin embargo, estos mismos medicamentos estuvieron comercialmente disponibles, y tenían registro sanitario, varios años antes de su inclusión en el POS, por lo que muchos pacientes pudieron acceder a ellos a través de la medicina privada o mediante el recurso de la tutela.
El esquema de tratamiento inicial más utilizado en Colombia, durante los años 90 y principios del siglo XXI, fue la combinación de zidovudina/ lamivudina más un inhibidor de proteasa, generalmente indinavir o ritonavir (4). Posteriormente, al hacerse evidentes los efectos secundarios de estos inhibidores de proteasa, ellos fueron progresivamente reemplazados por un inhibidor no nucleósido de transcriptasa inversa, usualmente nevirapina o efavirenz, por abacavir (un inhibidor nucleósido de transcriptasa inversa) o, en otros casos, por otros inhibidores de proteasa mejor tolerados como nelfinavir o lopinavir/ ritonavir, casi siempre junto a la combinación zidovudina/lamivudina (5,6). También se han usa do otras combinaciones, especialmente en los casos con falla terapéutica, y últimamente, combinaciones que utilizan medicamentos de más reciente desarrollo.
Los esquemas actuales de tratamiento han logrado un gran impacto en la morbimortalidad asociada a la infección por el VIH. Sin embargo, los beneficios de estos tratamientos se encuentran limitados por el alto costo de los fármacos, sus frecuentes efectos secundarios y por el fenómeno de la resistencia a los antivirales, definido como la capacidad del virus de multiplicarse en presencia de un fármaco antiviral, y producida por mutaciones en los genes virales que codifican las proteínas que son blanco del tratamiento (7).
De 20% a 45% de los pacientes que inician el tratamiento antirretroviral no logran alcanzar la meta de niveles indetectables de ARN viral, lo que aumenta la probabilidad de aparición de mutaciones que reducen la sensibilidad a los fármacos y que, posteriormente, se fijan por la presión selectiva del tratamiento (8,9,10). Otros factores que predisponen al desarrollo de resistencia son la falta de cumplimiento del tratamiento y el contagio de la infección a partir de un paciente previamente expuesto a medicamentos antirretrovirales (11).
Se han desarrollado pruebas genotípicas y fenotípicas para detectar la resistencia a los medicamentos antirretrovirales. Las primeras son las más usadas y se basan en la secuenciación de los genes virales de la transcriptasa inversa y la proteasa (7,12).
En Colombia, las pruebas genotípicas fueron introducidas desde el 2000 en el Centro de Análisis Molecular, laboratorio privado que opera en Bogotá. Como su introducción en el POS sólo se oficializó en 2008, la mayoría de los pacientes accedieron a ellas con recursos propios o mediante la tutela. Aunque su uso se ha concentrado, principalmente, en pacientes con falla terapéutica repetida, única indicación recomendada en las guías de atención del VIH/sida en Colombia (13), a algunas personas que utilizan la medicina privada se les ha realizado la prueba después de la primera falla terapéutica o aun antes de iniciar el tratamiento. Por consiguiente, la población incluida en este estudio es una mezcla de cepas virales con diferente grado de exposición a los medicamentos antirretrovirales, pero con un fuerte predominio de casos con algún grado de falla terapéutica.
Mediante el análisis consolidado de los resultados obtenidos en el Centro de Análisis Molecular entre 2000 y 2007, nos propusimos: 1. Identificar las mutaciones que más frecuentemente se asocian a la resistencia a los antirretrovirales, 2. Comparar la frecuencia de resistencia a los diferentes fármacos, y 3. Describir las variaciones en dicha frecuencia durante el período de estudio en Colombia.
Este trabajo fue ejecutado con la expectativa de que esta información pueda contribuir a la formulación de futuras pautas de tratamiento del VIH/sida en el país.
Materiales y métodos Tipo de estudio. Este estudio es descriptivo y retrospectivo.
Población y muestra. El estudio fue realizado con los resultados de 650 pruebas genotípicas de resistencia a los antirretrovirales, realizadas en el Centro de Análisis Molecular de Bogotá, durante el periodo de 2000 a 2007, a partir del plasma de pacientes de diferentes regiones del país, positivos para VIH. éstas constituyen la totalidad de las pruebas realizadas en dicho laboratorio y representan la mayoría de las pruebas ejecutadas en Colombia en ese lapso.
Recolección y procesamiento de la información. Los resultados analizados se obtuvieron utilizando la técnica HIV-1 Trugene® (Siemens Healthcare Diagnostics, Germany) con la cual, después de la extracción del ARN viral del plasma del paciente infectado, se amplifican y secuencian los genes de la transcriptasa inversa y de la proteasa del VIH.
Las secuencias obtenidas se procesaron por medio del programa Geno2pheno Resistance (http://www.geno2pheno.org) y utilizando la aplicación SVM (Support Vector Machine) que integra la información de todas las mutaciones presentes en cada genoma para predecir el fenotipo viral como sensible o resistente a cada medicamento (14,15).
El programa identifica las mutaciones asociadas con resistencia a siete inhibidores de proteasa (saquinavir, indinavir, ritonavir, nelfinavir, fosamprenavir, lopinavir y atazanavir), dos inhibidores no nucleósidos de transcriptasa inversa (nevirapina y efavirenz) y seis inhibidores nucleósidos de transcriptasa inversa (zidovudina, didanosina, estavudina lamivudina, abacavir y tenofovir). La aplicación proporciona, además, una predicción del subtipo viral. Los resultados se analizaron utilizando métodos de estadística descriptiva convencional.
Aspectos éticos. A las secuencias se les asignó un código para ser utilizadas en forma anónima. Los únicos datos que se conservaron asociados a la secuencia, fueron la fecha de toma de la muestra y su procedencia. Los datos clínicos no estaban disponibles y no se realizaron otros estudios de laboratorio diferentes a la secuenciación.
Resultados Las secuencias incluidas en el estudio se obtuvieron de muestras provenientes de 14 ciudades del país, principalmente de Bogotá (44,6%), Cali (12,5%), Medellín (10,7%) y Pereira (5,9%). En 16,8% de los casos, la procedencia era desconocida. Para algunas de las 650 secuencias procesadas, el programa Geno2pheno Resistance no arrojó un resultado, razón por la cual las frecuencias se calcularon sobre 637 secuencias para los inhibidores nucleósidos de transcriptasa inversa e inhibidores no nucleósidos de transcriptasa inversa y, sobre 648, para los inhibidores de proteasa. El 96% de las secuencias fueron clasificadas por el programa como de subtipo B y, 4%, como pertenecientes a otros subtipos.
La aplicación detectó más de 400 mutaciones y polimorfismos potencialmente asociados con resistencia a los distintos antirretrovirales, localizados en 176 posiciones de la transcriptasa inversa o de la proteasa. La frecuencia de las mutaciones varió entre 0,2% y 71,2%. La tabla 2 muestra las mutaciones encontradas con mayor frecuencia.

El número de medicamentos analizados para los cuales se detectó resistencia varió entre 0 y 15. En 82,1% de las secuencias se encontró resistencia, al menos, a uno de los antirretrovirales evaluados, 75,7% fue resistente a 2 o más medicamentos, 52,1%, a 5 o más, y 1,8% fue resistente a todos los 15 fármacos evaluados (figura 1).

La frecuencia de resistencia para los diferentes inhibidores nucleósidos de transcriptasa inversa varió entre 11% (estavudina) y 55,4% (lamivudina); para los inhibidores no nucleósidos de transcriptasa inversa, entre 52,6% (efavirenz) y 54,3% (nevirapina), y para los inhibidores de proteasa, entre 30,2% (fosamprenavir) y 38,0% (nelfinavir) (figura 2).

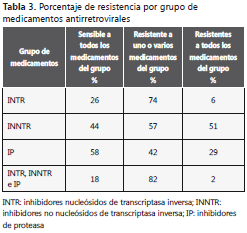
Según el grupo de fármacos, 74%, 57% y 42% de los casos estudiados exhibía resistencia, al menos, a un inhibidor nucleósido de transcriptasa inversa, inhibidor no nucleósido de transcriptasa inversa e inhibidor de proteasa, respectivamente, pero sólo 6%, 51% y 29% corresponde a virus resistentes a todos los medicamentos de cada grupo, en el mismo orden (tabla 3).
La frecuencia de resistencia ha variado en el tiempo. Para el año 2002, 59%, 18% y 55% de las secuencias estudiadas correspondieron a virus resistentes a uno o más medicamentos de los grupos de inhibidores nucleósidos de transcriptasa inversa, de inhibidores no nucleósidos de transcriptasa inversa y de inhibidores de proteasa, respectivamente (figura 3A). En los años subsiguientes se observó un rápido aumento en la resistencia a algún inhibidor no nucleósido de transcriptasa inversa, el cual se estabilizó alrededor de 60% a partir del 2004. También, hubo un aumento considerable, pero más gradual, en la resistencia a los inhibidores nucleósidos de transcriptasa inversa, hasta cerca de 85% en 2007. El porcentaje de resistencia a algún inhibidor de proteasa, exhibió fluctuaciones con una leve tendencia al descenso. Similares resultados se encontraron cuando se examinó individualmente la variación en la resistencia de algunos de los medicamentos de uso más frecuente durante el período del estudio. Hay una tendencia al aumento de la resistencia a la lamivudina, aumento seguido de estabilización para efavirenz y zidovudina, y una ligera disminución para indinavir (figura 3B).

Discusión La resistencia a los antirretrovirales ha sido poco estudiada en Colombia. Recientemente, Díaz Granados et al. publicaron el primer estudio de este tipo en el país, el cual incluyó muestras de 103 pacientes no tratados previamente y de 77 pacientes con una primera falla terapéutica (6). En el primer grupo se detectaron mutaciones asociadas a resistencia en 5,8% de los casos, mientras que en los pacientes con la primera falla estas mutaciones se encontraron en 85,7%. Esta última cifra es cercana a 82,1% de resistencia, al menos, a un fármaco encontrada en el presente estudio.
Las mutaciones en el gen de la transcriptasa inversa más frecuentemente asociadas a resistencia a inhibidores nucleósidos de transcriptasa inversa e inhibidores no nucleósidos de transcriptasa inversa en el estudio de DíazGranados et al., M184V (62,3%) y K103N (48,1%), también fueron las más frecuentes en este trabajo, aunque con una frecuencia un poco menor (53,1% y 35,4%, respectivamente). En cambio, en el gen de la proteasa las frecuencias son mucho más altas en nuestro estudio. Por ejemplo, las mutaciones de efecto mayor más frecuentes en este estudio, M46L/I, L90M/I, V82A/T/S e I54V, se encontraron en 28,9%, 28,0%, 24,8% y 21,8% de los casos en este estudio, pero sólo en 3,9%, 11,7%, 5,2% y 2,6% en el estudio citado (6). Sólo la mutación D30N fue más frecuente en ese estudio que en el nuestro (7,8% Vs. 4,9%).
Estas diferencias probablemente reflejan el menor uso de los inhibidores de proteasa en años recientes cuando se realizó el estudio citado, mientras que nuestro estudio recoge más casos que fueron tratados con el viejo esquema de dos inhibidores nucleósidos de transcriptasa inversa más un inhibidor de proteasa. En el caso de la mutación D30N, su mayor frecuencia en el estudio de DíazGranados et al. probablemente refleja un mayor uso del nelfinavir en la población de ese estudio (6).
En este estudio se observaron frecuencias de resistencia muy diversas para los inhibidores nu cleósidos de transcriptasa inversa; la mayor fue para la lamivudina (55,4%) y la menor para la estavudina (11,0 %). Por esta razón, aunque el 74% de los casos fueron resistentes a algún inhibidor nucleósido de transcriptasa inversa, sólo 6% fue resistente a todos los fármacos de este grupo (tabla 3), dejando, al menos, un medicamento como opción disponible para casos en los cuales se requiera cambio de tratamiento.
Sorprendentemente, la zidovudina, el medicamento antirretroviral más usado hasta ahora, es sólo el cuarto en frecuencia de resistencia entre los inhibidores nucleósidos de transcriptasa inversa, por debajo del abacavir y el tenofovir, medicamentos de introducción más reciente. Esto probablemente se debe a la alta frecuencia de la mutación M184V (53,1%), seleccionada por el uso de lamivudina, la cual aumenta la sensibilidad a la zidovudina, retardando, o aun revirtiendo, algunas de las mutaciones que confieren resistencia a este fármaco (11,16). La misma mutación, en presencia de otras, confiere resistencia cruzada al abacavir, lo que explica en parte la alta frecuencia (46,9%) de resistencia a este medicamento (16).
La alta frecuencia de resistencia al tenofovir (49,1%) pudo deberse a la acumulación de mutaciones como M41L (24,3%), D67N (23,5%), K70R (19,2%), L210W (19,2%), T215Y (42,5%) y K219Q/E (18,9%). Estas son las llamadas “mutaciones asociadas a análogos de timidina”, las cuales son seleccionadas por el uso de zidovudina o estavudina y están asociadas con resistencia cruzada a todos los inhibidores nucleósidos de transcriptasa inversa cuando se presentan tres o más de ellas en un mismo genoma (16).
Con respecto a los inhibidores no nucleósidos de transcriptasa inversa, se observaron frecuencias de resistencias de 52% y 55% para los dos medicamentos de este grupo incluidos en el análisis, y 51% de los virus fue resistente a ambos medicamentos. En 35,4% de los casos, esto se explica por la presencia de la mutación K103N asociada con resistencia completa a efavirenz y nevirapina, y probablemente, seleccionada por el incremento en el uso de este tipo de medicamentos en años recientes, como lo sugiere el rápido incremento en la resistencia entre 2002 y 2004 (figura 3). Otras mutaciones, como L100I, Y181C y Y188L, explican el resto de los casos. La baja “barrera genética a la resistencia”, definida como el número de mutaciones necesarias para generar un fenotipo resistente, parece ser el principal factor que predispone a la alta frecuencia de resistencia a este grupo de fármacos.
En el polo opuesto del rango de la barrera genética están los inhibidores de proteasa. En estos medicamentos, la presencia de una sola mutación asociada a resistencia tiene poco efecto y en la mayoría de los casos son necesarias, al menos, tres mutaciones (11).
Estas mutaciones suelen clasificarse como “mayores”, si afectan directamente la afinidad del fármaco por la enzima, o “menores”, si cumplen un papel compensatorio de la pérdida de la eficacia de replicación (fitness) asociada a las mutaciones mayores (10). En ausencia de estas últimas, las mutaciones menores no suelen generar ventaja selectiva para el virus.
Un hallazgo inesperado de este estudio fue que las cuatro substituciones más frecuentes en la proteasa fueron mutaciones menores (L10I/F/V, M36I/L) u otras no asociadas claramente a resistencia (L63P/R/S/T, I93L), las cuales se observaron con frecuencias entre 30% y 72%; mientras que las mutaciones mayores más frecuentemente encontradas, como M46L/I, L90M y V82A, presentaron frecuencias inferiores a 29% (tabla 2).
Una hipótesis para explicar este resultado es que en muchos casos un tratamiento anterior con inhibidores de proteasa pudo haber inducido tanto mutaciones mayores como menores, generando resistencia; al cambiarse el esquema de tratamiento para eludir la resistencia, las mutaciones mayores revirtieron, mientras que las menores pudieron persistir, ya que ellas son aproximadamente neutras en ausencia del fármaco (9,11). Como alternativa, las mutaciones menores pudieron preexistir al tratamiento, ya que se han encontrado como polimorfismos en personas que nunca han recibido tratamiento antirretroviral. La carencia de la historia medicamentosa de los pacientes nos impide discernir entre estas hipótesis.
En general, la resistencia a los inhibidores de proteasa fue menor que para los demás grupos (figura 2). Esto tiene dos posibles explicaciones: su alta barrera genética a la resistencia y la reducción de su uso en años recientes en favor de otros medicamentos, como nevirapina, efavirenz y abacavir. La tendencia decreciente en la resistencia a los inhibidores de proteasa observada en este estudio (figura 3), parece favorecer esta última explicación.
Aunque el programa empleado en este estudio no incluye a los antirretrovirales de más reciente desarrollo, la disponibilidad de las secuencias de la transcriptasa inversa y la proteasa, nos permite hacer anotaciones sobre la respuesta al tratamiento con algunos de ellos.
Es el caso de emtricitabina, cuyo perfil de resistencia es, aproximadamente, igual al de lamivudina (16), por lo cual se infiere una alta resistencia a ella en la población estudiada. La etravirina, un nuevo inhibidor no nucleósido de transcriptasa inversa, no parece afectarse con la mutación K103N, pero sí-con otras encontradas con frecuencia de 10% o menos (16), por lo que se puede predecir que su uso sería de utilidad en algunos, mas no en todos los casos que presentan resistencia a los inhibidores no nucleósidos de transcriptasa inversa incluidos en este estudio.
Con respecto a los inhibidores de proteasa de reciente desarrollo, darunavir y tipranavir, se puede predecir que el primero de ellos sería útil en muchos de los casos actuales de resistencia a este grupo de medicamentos, pues no se afecta con las mutaciones M46L/I, L90M y V82A/T/S, las más frecuentes mutaciones mayores encontradas en la proteasa. Con respecto a tipranavir, el pronóstico no es tan claro, pues se afecta en alguna medida con las mutaciones mencionadas, especialmente las de la posición 82. Sería necesario analizar todo el conjunto de mutaciones en cada caso, para evaluar la posibilidad de resistencia a este fármaco.
La mayor limitación de este estudio estuvo en que no se disponía de la historia medicamentosa de los pacientes de los cuales se obtuvo la secuencia. Otra limitación es que, por tratarse de un estudio realizado principalmente en pacientes previamente tratados con antirretrovirales, los resultados no se pueden utilizar para recomendar un esquema de tratamiento en pacientes sin tratamiento previo (naive) en Colombia; sin embargo, se pueden hacer algunas anotaciones que podrían ser de utilidad.
Recientemente, se recomendó la combinación tenofovir/emtricitabina, más un inhibidor no nucleósido de transcriptasa inversa o un inhibidor de proteasa reforzado con ritonavir, como esquema de primera elección para el inicio del tratamiento (17). Este esquema tiene ventajas desde el punto de vista farmacodinámico y de efectos secundarios sobre el esquema más utilizado durante el período del estudio y que consiste en la combinación zidovudina/lamivudina más un inhibidor no nucleósido de transcriptasa inversa o un inhibidor de proteasa.
Sin embargo, la alta frecuencia encontrada de la mutación M184V (53,1%), que confiere resistencia cruzada a la emtricitabina, y la alta frecuencia de resistencia inferida para tenofovir (49,1%), sugieren que ésta no sería una combinación de mucha utilidad en pacientes previamente tratados (11).
La presencia de algunos casos con resistencia a todos, o casi todos los fármacos disponibles actualmente en nuestro medio (figura 1), demuestra la importancia de introducir los grupos de medicamentos de más reciente desarrollo, como los inhibidores de la fusión, los bloqueadores del correceptor y los inhibidores de integrasa, como la única alternativa para estos casos.
En resumen, este estudio indica que la fijación de mutaciones generadoras de resistencia es un fenómeno frecuente y creciente en nuestro medio. Los niveles de resistencia a los distintos medicamentos están influenciados por la frecuencia de su uso, como lo revelan los cambios en los patrones de resistencia en el tiempo (figura 3), y por la baja barrera genética que favorece la resistencia a medicamentos como lamivudina y los inhibidores no nucleósidos de transcriptasa inversa. Sin embargo, el número de los medicamentos a los cuales el virus es resistente oscila ampliamente entre 0 y 15 (figura 1) y los patrones de resistencia varían considerablemente de un caso a otro.
En el estudio de DíazGranados et al. (2010), se predice que el tratamiento de segunda línea recomendado por las guías nacionales, en ausencia de la prueba genotípica, sería exitoso en 84% de los casos y atribuye esta relativamente alta tasa de éxito a la baja frecuencia de mutaciones asociadas a análogos de timidina en su estudio (11,7%) (6). En nuestro estudio, sin embargo, la frecuencia de dichas mutaciones fue más alta (18-42%) lo que indica que ese pronóstico debería tomarse con precaución.
La conveniencia de realizar pruebas de resistencia en pacientes con diagnóstico reciente de VIH y en aquellos que van a iniciar tratamiento por primera vez, ha sido recomendada por varios autores (12,17,18). Dicha conveniencia no fue respaldada por el trabajo de DíazGranados et al., quienes encontraron sólo 5,8% de resistencia en pacientes no previamente expuestos a fármacos antirretrovirales (6).
El presente estudio no permite hacer inferencias sobre el éxito de los tratamientos iniciales sin estudio previo, pero acogemos la recomendación derivada del mismo de mantener una constante vigilancia de estos niveles de resistencia en personas no tratadas, porque la transmisión de cepas resistentes es un hecho demostrado y la presencia de mutaciones en estas personas podría elevarse en un futuro.
Agradecimientos Los autores desean agradecer a la Universidad de Antioquia y al Centro de Análisis Molecular por proporcionar a los autores el tiempo y los recursos necesarios para realizar este estudio, al ingeniero Juan Pablo Morales de Ingenian Software, por su ayuda en la recuperación de las secuencias, y a la doctora María Teresa Rugeles por su revisión crítica del artículo.
Correspondencia: Francisco J. Díaz, Sede de Investigación Universitaria, Calle 62 N° 52-59, laboratorio 532, Medellín, Colombia. Dirección electrónica: franciscodiaz314@gmail.com
Referencias
1. Joint United Nations programme on HIV/AIDS (UNAIDS). 2007 AIDS epidemic update. Fecha de consulta: 12 de diciembre de 2008. Disponible en: http://www.unaids.org/en/KnowledgeCentre/HIVData/EpiUpdate/EpiUpdArchive/2007/ [ Links ]
2. Eyzaguirre L, Bautista CT, Ayala C, Acosta J, Negrete M, Sateren WB, et al. First case of HIV type 1 subtype F among men who have sex with men in Colombia. AIDS Res Hum Retroviruses. 2006;22:808-11. [ Links ]
3. US Food and Drug Administration. Drugs used in the treatment of HIV Infection, 2008. Fecha de consulta: 17 de abril de 2008. Disponible en: http://www.fda.gov/oashi/aids/virals.html [ Links ]
4. DíazGranados CA, Silva A, Bermúdez A, Roncancio D, DiRuggiero P, Mantilla M. Rate and predictors of optimal virologic response to antirretroviral therapy in Colombia. Int J Infect Dis. 2007;11:531-5. [ Links ]
5. Machado JE, Alzate JA. Patrones de prescripción de antirretrovirales en 997 pacientes colombianos. Biomédica. 2008;28:78-86. [ Links ]
6. DíazGranados CA, Mantilla M, Lenis W. Antiviral drug resistance in HIV-infected patients in Colombia. Int J Infect Dis. 2010;14:e298-303. [ Links ]
7. Shafer RW. Genotypic testing for HIV-1 drug resistance. Clin Microbiol Rev. 2002;15:247-77. [ Links ]
8. Kaufmann D, Muñoz M, Bleiber G, Fleury S, Lotti B, Martínez R, et al. Virological and immunological characteristics of HIV treatment failure. AIDS. 2000;14:1767-74. [ Links ]
9. Perno CF, Cozzi-Lepri A, Balotta C, Forbici F, Violin I, Bertoli A, et al. Secondary mutations in the protease region of human immunodeficiency virus and virologic failure in drug-naive patients treated with protease inhibitor-based therapy. J Infect Dis. 2001;184:983-91. [ Links ]
10. Chen R, Quiñones Mateu ME, Mansky LM. Drug resistance, virus fitness and HIV-1 mutagenesis. Curr Pharmaceutical Design. 2004;10:4065-70. [ Links ]
11. Clavel F, Hance AJ. HIV drug resistance. N Engl J Med. 2004;350:1023-35. [ Links ]
12. Gallant JE. Antiretroviral drug resistance and resistance testing. Topics HIV Med. 2005;13:138-42. [ Links ]
13. Ministerio de la Protección Social. Programa de apoyo a la Reforma de Salud (PARS). Guía para el manejo de VIH/sida basada en la evidencia, Colombia. Fecha de consulta: 10 de diciembre de 2008. Disponible en: http://www.minproteccionsocial.gov.co/VBeContent/library/documents/DocNewsNo15675 DocumentNo3250.PDF. [ Links ]
14. Beerenwinkel N, Lengauer T, Selbig J, Schmindt B, Hauke W, Korn K, et al. Geno2pheno: Interpreting genotypic HIV drug resistance tests. IEEE Intelligent Systems in Biology. 2001;16:35-41. [ Links ]
15. Beerenwinkel N, Daumer M, Oette M, Korn K, Hoffmann D, Kaiser R, et al. Geno2pheno: Estimating phenotypic drug resistance from HIV-1 genotypes. Nucl Acids Res. 2003;31:3850-5. [ Links ]
16. Johnson VA, Vezinet-Brun F, Clotet B, Gunthard HF, Kuritzkes D, Pillay D, et al. Update of the drug resistance mutations in HIV-1: December 2009. Topics in HIV Med. 2009;17:138-45. [ Links ]
17. Panel on Antiretroviral Guidelines for Adults and Adolescents. Guidelines for the use of antiretroviral agents in HIV-1-infected adults and adolescents, 2008. Department of Health and Human Services. Fecha de consulta: 12 de diciembre de 2008. Disponible en: http://www.aidsinfo.nih.gov/ContentFiles/AdultandAdolescentGL.pdf. [ Links ]
18. Lauria FN, Angeletti C. Cost-effectiveness analysis of using antiretroÂviral drug resistance testing. Scandinavian J Infect Dis. 2003;106:54-7. [ Links ]














