Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Facultad Nacional de Agronomía Medellín
Print version ISSN 0304-2847
Rev. Fac. Nac. Agron. Medellín vol.65 no.2 Medellín June/Dec. 2012
HONGOS NATIVOS CON POTENCIAL DEGRADADOR DE TINTES INDUSTRIALES EN EL VALLE DE ABURRÁ, COLOMBIA
NATIVE FUNGI WITH INDUSTRIAL DYE DEGRADING POTENTIAL IN THE ABURRÁ VALLEY, COLOMBIA
Xiomara Chanagá Vera1; Jersson Plácido Escobar2; Mauricio Marín Montoya3 y María del Socorro Yepes Pérez4
1 Bacterióloga. Universidad Nacional de Colombia - Sede Medellín - Facultad de Ciencias. A.A. 1779, Medellín, Colombia. <xiomarachanagav@gmail.com>
2 Ingeniero Biológico. Universidad Nacional de Colombia - Sede Medellín - Facultad de Ciencias. A.A. 3840, Medellín, Colombia. <jerssonplacido@gmail.com>
3 Profesor Asociado. Universidad Nacional de Colombia - Sede Medellín - Facultad de Ciencias. A.A. 3840, Medellín, Colombia.<mamarinm@unal.edu.co>
4 Profesora Asistente. Universidad Nacional de Colombia - Sede Medellín - Facultad de Ciencias. A.A. 3840, Medellín, Colombia. <msyepes@unal.edu.co>
Recibido: Febrero 28 de 2012; aceptado: Agosto 25 de 2012.
Resumen. Los colorantes industriales poseen estructuras químicas estables que dificultan su tratamiento mediante procesos fisicoquímicos convencionales. En los últimos años, como una alternativa biotecnológica para la degradación de compuestos recalcitrantes, se han utilizado hongos ligninolíticos de diferentes grupos taxonómicos, que producen enzimas oxidantes de dichas moléculas. El aislamiento e identificación de especies fúngicas nativas con potencial decolorante, resulta promisorio para biorremediar efluentes provenientes de industrias textiles. En esta investigación se identificaron, con base en análisis de secuencias de las regiones ITS1 e ITS2 y 28S del ADNr, y por sus características morfológicas, cuatro hongos nativos aislados de material lignocelulósico colectado en el Valle de Aburrá (Antioquia, Colombia). Los aislamientos fueron identificados como el ascomycete Leptosphaerulina sp., y los hongos anamórficos Trichoderma viride (dos cepas) y Aspergillus niger.
Palabras clave: Biorremediación, decoloración, hongos ligninolíticos, secuenciación.
Abstract. Synthetic dyes have stable chemical structures that hinder their treatment by conventional physicochemical processes. In recent years, as a biotechnological alternative for degradation of these recalcitrant compounds, wood degrading fungi of different taxonomic groups have been used. These fungi produced enzymes with oxidative potential for those molecules. The isolation and identification of ligninolytic fungi with potential for discoloration is promising for bioremediation of effluents from textile industries. In this research, we identified four native fungi isolated from lignocellulosic material in the Aburra Valley (Antioquia, Colombia). Identification was made based on sequence analysis of ITS1-ITS2 regions and 28S rDNA as well as morphological characteristics. The fungi were identified as the ascomycete Leptosphaerulina sp., and the anamorphic species Trichoderma viride (two strains) and Aspergillus niger.
Key words: Biorremediation, decoloration, ligninolytic fungi, sequencing.
Los colorantes industriales que se emplean en la industria textil, se seleccionan no sólo por la gran diversidad de colores que brindan, sino también por la estabilidad química. Durante el proceso de tintura, una parte de los tintes pasa al agua de enjuague, la que luego es vertida a los efluentes que generalmente rodean éstas factorías (Corso y Maganha, 2009).
El impacto negativo de estos colorantes está asociado principalmente a la alteración de los procesos fotosintéticos del fitoplancton, afectando gravemente el equilibrio de los ecosistemas acuíferos en los que son vertidos (Michniewicz et al., 2008). Además, son compuestos con efectos carcinogénicos y mutagénicos en diferentes organismos, incluyendo los humanos (Weisburger, 2002; Oliveira et al., 2007). En consecuencia, es prioritario el desarrollo de tecnologías que conlleven a la degradación de los colorantes industriales que permanecen inalterados en ríos y mares, debido a su alto nivel de recalcitrancia y contaminación.
Los hongos que crecen sobre la corteza vegetal despolimerizan y mineralizan la lignina, accediendo a los carbohidratos de la celulosa y la hemicelulosa. Esta capacidad se ha aprovechado para desarrollar sistemas biológicos direccionados a tratar contaminantes de alta estabilidad, como los colorantes sintéticos textiles (Swamy y Ramsay, 1999; Robinson et al., 2001).
Entre los hongos que han mostrado potencial para remover tintes a través de procesos de degradación y/o adsorción, se encuentran levaduras (Niladevi y Prema, 2008), Ascomycetes filamentosos y sus formas anamórficas (hongos mitospóricos) y diversos Basidiomycetes asociados fundamentalmente a la podredumbre blanca (Toh et al., 2003; Kandelbauer y Guebitz, 2005).
Considerando la importancia de la degradación enzimática como una alternativa para el biotratamiento de colorantes industriales, en esta investigación se identificaron, mediante métodos morfológicos y análisis filogenéticos, hongos nativos obtenidos de residuos celulolíticos, que en estudios previos mostraron capacidad de decolorar algunos tintes utilizados en la industria textil del Valle de Aburrá.
MATERIALES Y MÉTODOS
Especies fúngicas. Los hongos depositados en la colección del grupo de investigación Producción, Estructura y Aplicación de Biomoléculas (PROBIOM), de la Universidad Nacional de Colombia, denominados LVN6, LVN49 y LVN72, fueron aislados de material lignocelulósico del Valle de Aburrá; mientras que LVN93, corresponde a un saprófito comúnmente presente en dicha región. Estos microorganismos fueron seleccionados por la capacidad de decolorar los tintes Azul Turquesa Hispasol®, Negro Remazol B® y Rojo Novacrón® (Plácido, 2011). Los aislados se cultivaron en papa dextrosa agar (PDA) a temperatura ambiente y se almacenaron a 4 °C hasta su uso posterior.
Purificación de los hongos nativos. Los hongos fueron purificados a partir de cultivos monohifales. Para ello, se cultivaron en medio PDA durante ocho días a 28 °C. Luego, cuadrantes de 0,5 cm2 de micelio fueron transferidos a un medio de agar-agua al 2%. Al cabo de 12 a 24 h, se evaluó la generación de hifas germinativas bajo estereoscopio. Finalmente, se transfirió una única hifa en crecimiento activo a nuevas cajas con medio PDA y se incubó por ocho días a 28 °C.
Extracción de ADN. Los hongos purificados se transfirieron a erlenmeyers de 250 mL con 50 mL de medio líquido extracto de malta al 2% (ME), tomando tres porciones de 1 cm2 de micelio. Los cultivos fueron mantenidos a temperatura ambiente durante 16 a 20 días. Seguidamente, se realizó la filtración y secado del micelio, utilizando una bomba de vacío y papel aluminio perforado. El filtrado fue sometido a maceración física con nitrógeno líquido.
La extracción de ADN se llevó a cabo mediante dos métodos: CTAB 3X y utilización del kit comercial DNeasy plant mini (Qiagen®). Para el primer caso, se partió de 200 mg de micelio macerado en tubos eppendorf de 1,5 mL, adicionándose 400 mL de buffer CTAB 3X (CTAB 3% (p/v), 100 mM Tris HCL, pH 8,0, 1,4 M de NaCl, 20 mM de EDTA pH 8,0) y 150 mL de SDS 20%. El conjunto se incubó a 65 °C por 30 min. Luego, se agregó 1 vol. de fenol-cloroformo (1:1), mezclándose por inversión durante 10 min, para proceder a centrifugar a 13.000 rpm por 15 min.
Para la precipitación de los ácidos nucleicos se utilizó 1 vol. de isopropanol frío y 0,1 vol. de acetato de sodio 3 M. La mezcla se centrifugó a 13.000 rpm por 30 min. El pellet generado fue lavado con 400 mL de etanol al 70%, centrifugado a 13.000 rpm por 5 min y resuspendido en 50-100 mL de agua destilada estéril. Finalmente, se eliminó el ARN por tratamiento enzimático con 3 mL de ARNasa A (10 mg mL-1), a 37 °C por 24 h.
La integridad del ADN obtenido se evaluó por electroforesis en gel de agarosa al 1% en buffer TBE 1X (45 mM Tris-Borato, 1 mM EDTA pH 8,0), suplementado con bromuro de etidio (0,5 mL bromuro/10 mL), durante 60 min a 70 V. La visualización del gel se realizó bajo luz ultravioleta en un transiluminador Bio Doc Analyze (Biometra®) y su cuantificación mediante lecturas a 260 nm en Nanodrop 2000C (Thermo ®).
Cuando el ADN obtenido no era amplificable mediante PCR, se utilizó el kit comercial a partir de 100 mg de micelio macerado con buffer de extracción AP1 y 4 mL de ARNasa A, siguiendo las instrucciones del fabricante.
PCR. Los hongos se identificaron molecularmente, a partir de la amplificación y secuenciación de regiones ITS del ADNr, mediante la utilización de los cebadores universales ITS1 (5' TCC GTA GGT GAA CCT GCG G 3') e ITS4 (5' TCC TCC GCT TAT TGA TAT GC 3') (White et al., 1990) y de la región 5´del ADNr 28S con los cebadores LROR (5' ACC CGC TGA ACT TAA GC 3') y LR6 (5' CGC CAG TTC TGC TTA CC 3') (Zuluaga et al., 2011).
Las reacciones de PCR contenían 1X Buffer PCR (100 mM Tris-HCl pH 8,8, 500 mM KCl, 0,8% v/v Nonidet P40), 0,2 mM dNTPs, 2 mM MgCl2, 0,4 mM de los cebadores, 2 U de Taq Polimerasa (Fermentas®), 0,2 mL de BSA (10 mg/mL) y 25 ng de ADN, completándose con H2Odd hasta 25 mL. La amplificación se inició con una desnaturalización a 95 °C durante 3 min, seguida de 40 ciclos de 94 °C por 30 s, 58 °C por 50 s, 72 °C por 1 min, y una extensión final a 72 °C por 5 min. Los amplicones generados se visualizaron en electroforesis de agarosa al 1,5%, utilizando como patrón de referencia el marcador de peso molecular GeneRuler 100 pb (Fermentas®).
Los amplicones del tamaño esperado se purificaron mediante los kits QIAquick PCR Purification y QIAquick Gel Extraction (Qiagen®), dependiendo de su especificidad. Los fragmentos fueron secuenciados en ambos sentidos mediante el sistema Big Dye Terminator Cycle Sequencing Ready Reaction (PE Applied Biosystems®) y su corrido se realizó en un secuenciador ABI Prism 3730xl (PE Applied Biosystems®) de la compañía Macrogen.
Análisis de secuencias de las regiones ITS y 28S del ADNr. Las secuencias generadas con cada cebador, se editaron mediante el software Chromas 1.45., construyéndose los consensos con el software Bioedit 6.0.6. Luego, se dedujeron las secuencias consenso evaluándose su identidad preliminar por comparación con las bases de datos moleculares, mediante el programa BLAST (http://www.ncbi.nlm.nih.gov/BLAST/).
Posteriormente, se procedió a su alineamiento con el algoritmo Clustal W incluido en el software Bioedit 6.0.6, con respecto a secuencias de referencia obtenidas del GenBank. La matriz generada fue utilizada para realizar un análisis filogenético basado en máxima parsimonia (MP) con búsqueda heurística y TBR (tree-bisection-reconnection), mediante el software PAUP 4,0b10 (Swofford, 1998), realizando 1.000 remuestreos y tratando los espacios (gaps) como quinta base. El soporte de la topología interna de los dendrogramas fue considerado mediante análisis de bootstrap con 1.000 iteraciones (Felsenstein, 1985).
Caracterización morfológica. Para la caracterización morfológica, cada hongo se cultivó por triplicado en cajas Petri con los medios PDA, EM y agar-jugo V8, ubicándose en el centro de las cajas un disco de 4 mm de diámetro del hongo. Con el fin de inducir la esporulación, cada replica se mantuvo bajo diferentes condiciones de fotoperíodo: luz constante, oscuridad constante y 12 h luz/12 h oscuridad, por 30 días.
Las observaciones se realizaron en un microscopio óptico Zeiss® acoplado a un sistema fotográfico digital. Se utilizaron como base de comparación de las estructuras observadas, las herramientas de búsqueda del Mycobank (http://www.mycobank.org/).
Finalmente, comparando los resultados morfológicos con los análisis filogenéticos, se identificaron las especies en cuestión.
Análisis por RAPD. Tres diferentes réplicas del aislamiento LVN6, seleccionado por la alta capacidad de decoloración de tintes sintéticos, fue sometido a análisis por RAPDs (Random Amplification of Polymorphic DNA) para su genotipificación. El ADN de los tres individuos (LVN6-1, LVN6-2 y LVN6-3), fue obtenido por los métodos de extracción antes descritos. Para los RAPDs se utilizaron seis cebadores aleatorios (Tabla 1) en reacciones individuales de 20 mL, que incluyeron 10,8 mL H2Odd, 1X Buffer PCR (100 mM Tris-HCl pH 8,8, 500 mM KCl, 0,8% (v/v) Nonidet P40), 0,2 mM dNTPs, 3 mM MgCl2, 0,4 mM del cebador, 2 U de Taq Polimerasa (Fermentas®) y 25 ng de ADN. La amplificación se inició con una desnaturalización a 98 °C durante 3 min, seguida de 40 ciclos de 94 °C por 1 min, 36 °C por 1 min, 72 °C por 2 min, y una extensión final a 72 °C por 7 min. Se incluyó un control negativo para cada cebador.
Los amplicones generados se visualizaron en electroforesis de agarosa al 2,5% a 60 V durante 2,5 h, tal y como se describió anteriormente. Se utilizaron como patrones de referencia los marcadores de peso molecular GeneRuler 100 pb y 1 kb DNA Ladder (Fermentas®).
RESULTADOS Y DISCUSIÓN
Análisis filogenético de ITS. En los cuatro aislamientos bajo estudio se logró la amplificación del ADN, generándose bandas de 500 a 600 pb para las regiones ITS del ADNr (Figura 1).
Los análisis BLAST y de identidad de las secuencias, permitieron la identificación preliminar de estos hongos como miembros de los géneros: Leptosphaerulina, Trichoderma y Aspergillus (Tablas 2 y 3).
El análisis filogenético se realizó con el fin de identificar los aislamientos a nivel de especie e incluyó las cuatro secuencias completas, obtenidas en ambas direcciones en este trabajo. Además, se utilizaron 27 secuencias de hongos depositadas en el GenBank seleccionadas por presentar altos niveles de identidad con los aquí obtenidos y dos hongos-roya de las especies Melampsora laricis populina y Uromyces trifolii-repentis, utilizados como grupos externos de análisis (outgroups).
El alineamiento final de las secuencias alcanzó 629 sitios, 154 de los cuales eran caracteres constantes, 89 variables pero no informativos y 362 informativos. La longitud del árbol filogenético con máxima parsimonia fue de 1.420, con índices de consistencia (IC) y retención (IR) de 0,71 y 0,90, respectivamente. En el dendrograma se presentaron cinco clados bien definidos, todos ellos con soportes de bootstrap >90% (Figura 2).
Estos clados permitieron identificar a nivel de especie a los hongos LVN72 y LVN49 como Trichoderma viride; mientras que el análisis filogenético no ofreció una resolución suficiente para determinar las especies de los hongos identificados como miembros de los géneros Aspergillus y Leptosphaerulina.
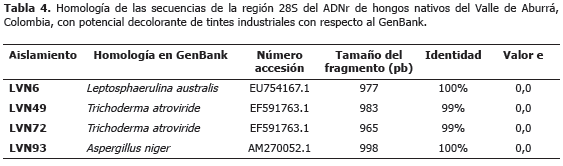
Análisis filogenético de 28S. La amplificación de la región 5´ del ADNr 28S generó fragmentos de cerca de 1.100 pb (Figura 1). Los análisis BLAST y de identidad de dichas secuencias, permitieron confirmar los géneros de los hongos identificados en el trabajo como L. americana y T. atroviride y adicionalmente, determinar la identidad del cuarto hongo como Aspergillus niger P.E.L. van Tieghem (Tablas 4 y 5).
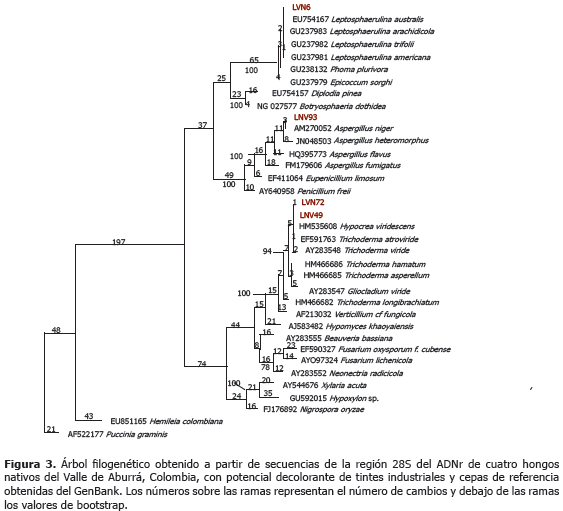
El alineamiento final para este análisis alcanzó 1.046 sitios, de los cuales 573 caracteres eran constantes, 92 variables pero no informativos y 381 informativos. La longitud del árbol filogenético con máxima parsimonia fue de 1.077, con índices de consistencia (IC) y retención (IR) de 0,65 y 0,89, respectivamente y su enraizamiento se efectúo con base en secuencias de los hongos-roya Hemileia colombiana y Puccinia graminis (Figura 3).
Caracterización morfológica. Las observaciones microscópicas y macroscópicas permitieron confirmar las identificaciones filogenéticas (Figuras 4 y 5).
Las mediciones de las estructuras microscópicas para el aislamiento LVN6, son características del género Leptosphaerulina, aunque no fue posible su identificación a nivel de especie. Se observaron peritecios agregados con gran cantidad de células pseudoparenquimatosas de paredes gruesas y melanizadas, ascas bitunicadas con ocho ascosporas hialinas elipsoides del tipo dictiosporas y rodeadas de una matriz gelatinosa.
Hasta donde se conoce, no existen reportes de L. americana, siendo utilizado para la decoloración de tintes industriales; aunque Simon et al. (1979), describieron la presencia y las características de lacasas en la especie L. briosinia, lo cual representa su potencial como degradador de colorantes. En el trabajo previo a esta investigación, Plácido (2011), encontró que la cepa LVN6 presenta una alta actividad de producción de enzimas ligninolíticas, por lo cual es fundamental continuar con la identificación a nivel de especie de este aislamiento, utilizando análisis de múltiples loci que incluyan secuencias de los genes b-Tubulina, Calmodulina y factor de elongación a, entre otros. Similarmente, es necesario emprender nuevas evaluaciones que determinen el efecto de este hongo (independiente y en conjunto con los otros hongos bajo estudio), sobre otros tipos de colorantes y bajo diferentes condiciones de cultivo, de manera que en el mediano plazo sea posible su aplicación en nuestro medio para el tratamiento de efluentes contaminados con colorantes industriales.
Con respecto a los aislamientos LVN48 y LVN72 de T. viride, se encontraron conidias ovaladas de color verde oliva, con diámetros entre 3,75 - 4,50 mm y originadas sobre conidióforos ramificados en ángulos rectos. Además, presenta fiálides atenuadas en sus puntas, con cerca de 10 mm de longitud. Después de 30 días de incubación, estos aislamientos presentaron gran cantidad de clamidosporas terminales e intercalares.
Las especies pertenecientes al género Trichoderma, se caracterizan por ser ampliamente utilizados en la agricultura, principalmente para el control biológico de hongos fitopatógenos como Rhizoctonia, Fusarium y del oomycete Pythium. También se han registrado como decoloradoras de tintes industriales. Por ejemplo, T. viride ha sido estudiado por su potencial en la bio-absorción del azul de metileno y el tinte azoico Orange G, respectivamente (Saeed et al., 2009; Sivasamy y Sundarabal, 2011); mientras que con el uso de la enzima lacasa de T. harzianum, es posible disminuir el color de sistemas acuosos con rodamina 6G, azul ácido 9 y azul de Tripan (Sadhasivam et al., 2009). T. atroviride y T. longibrachiatum, se han descrito como productores de lacasas con potencial en biorremediación (Gochev y Krastanov, 2007).
Por otra parte, el aislamiento LVN93 fue identificado morfológicamente como A. niger al presentar conidias esféricas y oscuras, de diámetro entre 3,75 y 5,00 mm, ornamentadas con múltiples rugosidades y generadas en vesículas subesféricas de aproximadamente 80 mm de diamétro. Las células conidiogénicas tipo fiálides son biseriadas y las métulas generalmente duplican la longitud de las fiálides.
Diferentes especies de Aspergilllus han sido mencionadas por su capacidad para remover tintes sintéticos. Entre las más destacadas se presenta la especie detectada en este trabajo: A. niger, utilizada para la descontaminación de sistemas acuosos con azul drimaren K2-RL, Azul ácido 29, Azul básico 9, Rojo Congo, Rojo disperso 1 y Orange G (Fu y Viraraghavan, 2003; Andleeb et al., 2010; Sivasamy y Sundarabal, 2011). Similarmente, Jin et al. (2007) demostraron que A. fumigatus XC6 decoloró efluentes con una mezcla de colorantes textiles reactivos y diferentes aislamientos de las especies A. flavus, A. oryzae, A. parasiticus y A. ochraceus, removieron colorantes diversos por biosorción y/o degradación (Akar et al., 2009; Corso y Maganha, 2009; Telke et al., 2010; Lalitha et al., 2011).
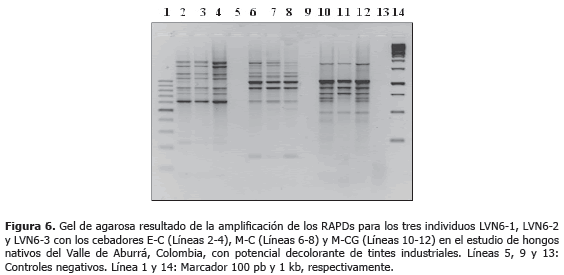
Análisis RAPD. De los seis cebadores evaluados para los marcadores RAPDs, fueron seleccionados EcoRI-C, MseI-C y MseI-CG, por presentar mayor polimorfismo, resolución y reproducibilidad experimental. Se compararon los perfiles electroforéticos de los tres individuos de Leptosphaerulina sp., lográndose identificar fragmentos polimórficos en el rango de 500 a 2.000 pb (Figura 6).
De acuerdo a los resultados, se estableció que los tres individuos analizados, obtenidos a partir de hifas individuales del aislado original LVN6, no presentaron variabilidad intraespecífica a través de RAPDs; por lo cual, dichos perfiles se pueden utilizar como base para la genotipificación de este hongo en ensayos de re-aislameinto y aplicaciones de biorremediación, siendo posible hacer un seguimiento en tiempo y espacio de las aplicaciones de dichos aislamientos, especialmente para evaluar su permanencia en el sustrato o efluente bajo análisis.
Este estudio constituye un paso en la selección de hongos para la biorremediación de efluentes con contaminantes orgánicos en la industria textil del Valle de Aburrá, y abre el camino a otros estudios para identificar los mecanismos fisiológicos, bioquímicos y moleculares que tienen estos hongos para tolerar, acumular, transformar y/o mineralizar tintes industriales.
CONCLUSIONES
Se identificaron con ayuda de herramientas morfológicas y moleculares, el ascomycete Leptosphaerulina sp., dos aislamientos de la especie mitospórica Trichoderma viride y una cepa de Aspergillus niger, a partir de material lignocelulósico colectado en el Valle de Aburrá (Antioquia, Colombia).
Los hongos mencionados tienen potencial para su utilización como agentes de biorremediación de efluentes de industrias textiles del Valle de Aburrá y de cuerpos de agua contaminados con tintes industriales de diferente naturaleza química.
BIBLIOGRAFÍA
Akar, S., T. Akar and A. Çabuk. 2009. Decolorization of a textile dye, Reactive Red 198 (RR198), by Aspergillus parasiticus fungal biosorbent. Brazilian Journal of Chemical Engineering 26(2): 399-405. [ Links ]
Andleeb, S., N. Atiq, M. Ali, F. Ur-Rehman, A. Hameed and S. Ahmad. 2010. Biodegradation of anthraquinone dye by Aspergillus niger SA1 in self designed fluidized bed bioreactor. Iranian Journal of Environmental Health Science and Engineering 7(5): 371-376. [ Links ]
Corso, C. and A. Maganha. 2009. Bioremediation of dyes in textile effluents by Aspergillus oryzae. Microbial Ecology 57(2): 384-390. [ Links ]
Felsenstein, J. 1985. Confidence limits on phylogenies: an approach using the bootstrap. Evolution 39(4): 783-791. [ Links ]
Fu, Y. and T. Viraraghavan. 2003. Column studies for biosorption of dyes from aqueous solutions on immobilised Aspergillus niger fungal biomass. Water SA 29(4): 465-472. [ Links ]
Gochev, V.K. and A.I. Krastanov. 2007. Isolation of laccase producing Trichoderma spp. Bulgarian Journal of Agricultural Science 13: 171-176. [ Links ]
Jin, X.C., G.Q. Liu, Z.H. Xu and W.Y. Tao. 2007. Decolorization of a dye industry effluent by Aspergillus fumigatus XC6. Applied Microbiology and Biotechnology 74(1): 239-243. [ Links ]
Kandelbauer, A. and G. Guebitz. 2005 Bioremediation for the decolorization of textile dyes: A review. pp. 269-288. In: Lichtfouse, E., J. Schwarzbauer and D. Robert (eds.). Environmental chemistry: green chemistry and pollutants in ecosystems. Springer, Berlin. 806 p. [ Links ]
Lalitha, P., N. Nageswara and K. Arunalakshmi. 2011. Decolorization of synthetic dyes by Aspergillus flavus. Bioremediation Journal 15(2) 121-132. [ Links ]
Michniewicz, A., S. Ledakowicz, R. Ullrich and M. Hofrichter. 2008. Kinetics of the enzymatic decolorization of textile dyes by laccase from Cerrena unicolor. Dyes and Pigments 77(2): 295-302. [ Links ]
Niladevi, K.N. and P. Prema. 2008. Effect of inducers and process parameters on laccase production by Streptomyces psammoticus and its application in dye decolourization. Bioresource Technology 99(11): 4583-4589. [ Links ]
Oliveira, D., P. Carneiro, M. Sakagami, M.V. Zanoni and G. Aragaõ. 2007. Chemical characterization of a dye processing plant effluent - identification of the mutagenic components. Mutation Research 626(1): 135-142. [ Links ]
Plácido, J.E. 2011. Aislamiento y selección de microorganismos con actividad decolorante. Tesis Maestría en Ciencias - Biotecnología. Facultad de Ciencias. Universidad Nacional de Colombia. Medellín. 244 p. [ Links ]
Robinson, T., B. Chandran and P. Nigam. 2001. Studies on the production of enzymes by white-rot fungi for the decolourisation of textile dyes. Enzyme and Microbial Technology 29(8): 575-579. [ Links ]
Sadhasivam, S., S. Savitha and K. Swaminathan. 2009. Redox-mediated decolorization of recalcitrant textile dyes by Trichoderma harzianum WL1 laccase. World Journal of Microbiology and Biotechnology 25(10): 1733-1741. [ Links ]
Saeed, A., M. Iqbal and S.I Zafar. 2009. Immobilization of Trichoderma viride for enhanced methylene blue biosorption: batch and column studies. Journal of Hazardous Materials 168(1): 406-415. [ Links ]
Simon, L.T., D.S. Bishop and G.R. Hooper. 1979. Ultrastructure and cytochemical localization of laccase in two strains of Leptosphaerulina briosiana (Pollaci) Graham and Luttrell. Journal of Bacteriology 137(1): 537-544. [ Links ]
Sivasamy, A. and N. Sundarabal. 2011. Biosorption of an azo dye by Aspergillus niger and Trichoderma sp. fungal biomasses. Current Microbiology 62(2): 351-357. [ Links ]
Swamy, J. and J.A. Ramsay. 1999. The evaluation of white rot fungi in the decoloration of textile dyes. Enzyme and Microbial Technology 24(2): 130-137. [ Links ]
Swofford, D.L. 1998. PAUP*: Phylogenetic analysis using parsimony (and other methods). Version: 4. software package. Sinauer Associates, Sunderland, MA. USA. [ Links ]
Telke, A.A., A.A. Kadam, S.S. Jagtap, J.P. Jadhav and S.J. Govindwar. 2010. Biochemical characterization and potential for textile dye degradation of blue laccase from Aspergillus ochraceus NCIM-1146. Biotechnology and Bioprocess Engineering 15(4): 696-703. [ Links ]
Toh, Y.C., J.J. Lin-Yen, J.P. Obbard and Y.P. Ting. 2003. Decolourisation of azo dyes by white-rot fungi (WRF) isolated in Singapore. Enzyme and Microbial Technology 33(5): 569-575. [ Links ]
Weisburger, J. 2002. Comments on the history and importance of aromatic and heterocyclic amines in public health. Mutation Research 506(507): 9-20. [ Links ]
White, T.J., T. Bruns, S. Lee and J. Taylor. 1990. Amplification and direct sequencing of fungal rRNA genes for phylogenetics, pp. 315-322. In: Innis, A.M., D.H. Gelfand, J.J. Sninsky and T.J. White (eds.). PCR Protocols: A guide to methods and applications. Academic Press, San Diego. 482 p. [ Links ]
Zuluaga, C., P. Buriticá and M. Marín. 2011. Filogenia de hongos roya (Uredinales) en la zona andina colombiana mediante el uso de secuencias del ADN ribosomal 28S. Revista de Biología Tropical 59(2): 517-540. [ Links ]